1. Calculs et tracés élémentaires dans une session R
On voudrait avec R tracer en bleu clair l'histogramme des classes d'une série de valeurs quantitatives, la loi normale sous-jacente en rouge, et la courbe de densité estimée par noyau en bleu foncé, comme pour le tracé ci-dessous.
On fournira aussi au passage les descripteurs usuels (moyenne, écart-type, médiane...)
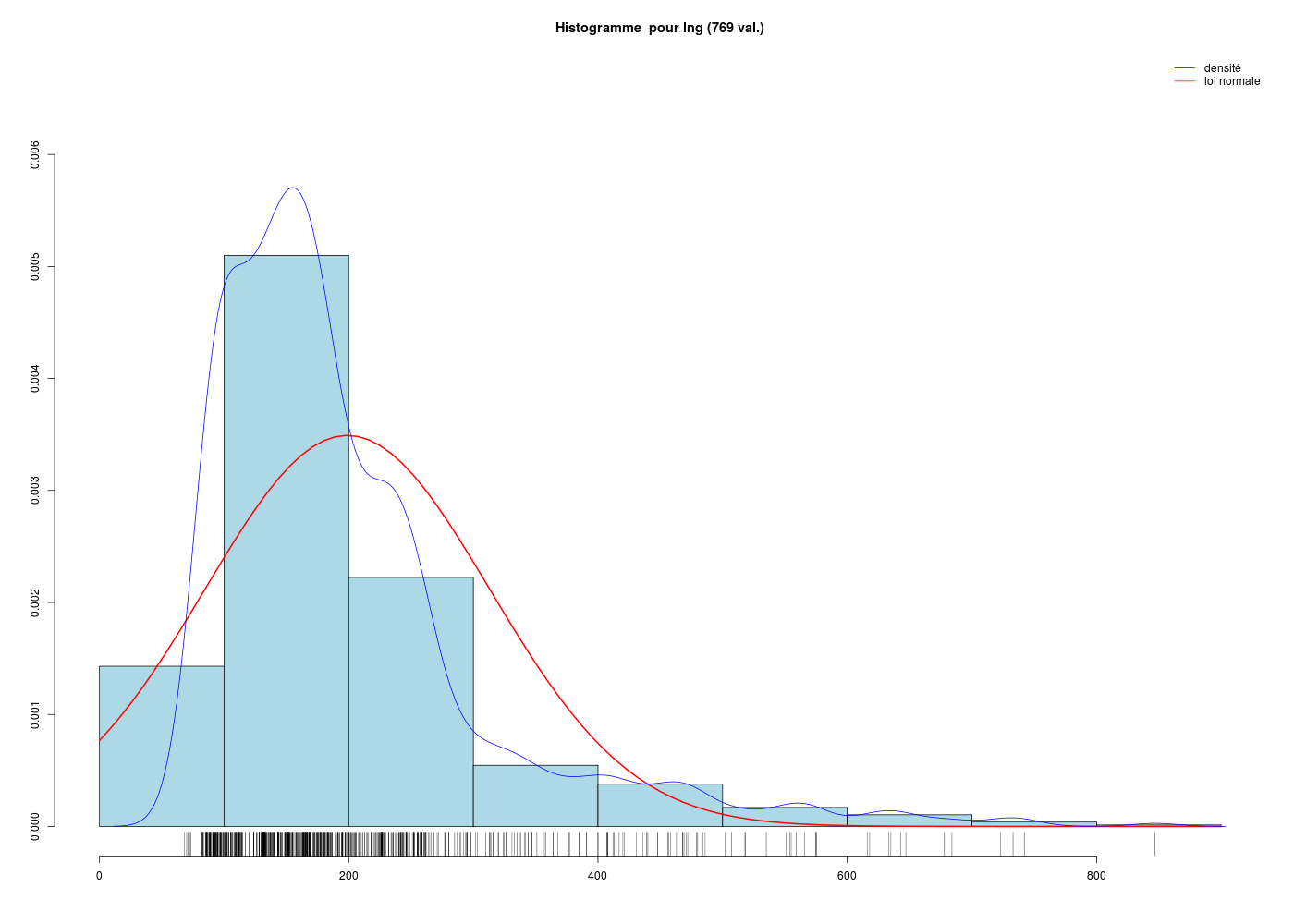
On pourra par exemple lire les données de longueur, issues du dossier LEADB avec les instructions suivantes :
source("http://www.info.univ-angers.fr/~gh/statgh.r",encoding="latin1")
lea <- lit.dar("http://www.info.univ-angers.fr/~gh/Datasets/lea.dar")
lng <- lea$length
Il y a visiblement
-
un histogramme en bleu ciel, tracé via la fonction hist(),
-
la loi normale en rouge, sans doute tracée via curve(),
-
la courbe de densité estimée par noyau en bleu foncé via lines(),
-
une estimation visuelle de la densité via un "peigne" sur l'axe x avec rug(),
-
une légende en haut et à droite obtenue par legend(),
-
et enfin un titre et les labels pour les axes produits par title().
Il faut donc sans doute six instructions de tracé pour réaliser le graphique. Vérification (tester par copier/coller de chaque ligne au fur et à mesure) :
# lecture des données
source("http://www.info.univ-angers.fr/~gh/statgh.r",encoding="latin1")
lea <- lit.dar("http://www.info.univ-angers.fr/~gh/Datasets/lea.dar")
lng <- lea$length
# histogramme (adapté sur l'axe Y pour la densité)
hist(lng,col="lightblue",main="",probability=TRUE,ylim=c(0,0.006),xlab="",ylab="")
# loi normale associée et son tracé
vnorm <- function(x) { return( dnorm(x,mean=mean(lng),sd=sd(lng)) ) }
curve(vnorm,add=TRUE,col="red",lwd=2)
# densité estimée par noyau et son tracé
dns <- density(lng)
lines(dns,col="blue")
# "peigne" de densité sur l'axe X
rug(lng)
# la légende en haut et à droite
legend(x="topright",c("densité","loi normale"),col=c("blue","red"),lty=c(1,1),bty="y")
# le titre et les labels des axes
titre <- paste("Histogramme des",length(lng),"longueurs")
title(main=titre,xlab="longueur des séquences",ylab="densité")
Les fonctions dnorm et density() font partie du package stats, bien sûr.
Pour obtenir des résumés statistiques comme la moyenne, la médiane, l'écart-type..., R dispose de nombreuses fonctions telles que mean() median() sd() et on peut donc écrire des instructions comme
# lecture des données
source("http://www.info.univ-angers.fr/~gh/statgh.r",encoding="latin1")
lea <- lit.dar("http://www.info.univ-angers.fr/~gh/Datasets/lea.dar")
lng <- lea$length
cat("Etude de la LONGUEUR en acides aminés des protéines de la LEADB\n\n")
print( summary(lng) )
cat("Nombre de valeurs ",length(lng)," appartements\n")
cat("Moyenne ",mean(lng)," aa\n")
cat("Médiane ",median(lng)," aa\n")
cat("Etendue ",max(lng)-min(lng)," aa\n")
cat("Variance ",var(lng)," aa x aa\n")
cat("Ecart-type ",sd(lng)," aa\n")
cat("\n")
pour obtenir
Etude de la LONGUEUR en acides aminés des protéines de la LEADB
Min. 1st Qu. Median Mean 3rd Qu. Max.
68.0 130.0 168.0 205.7 236.0 1864.0
Nombre de valeurs 773 protéines
Moyenne 205.6882 aa
Médiane 168 aa
Etendue 1796 aa
Variance 22109.43 aa x aa
Ecart-type 148.6924 aa
Mais ce n'est pas bien présenté, et donc pas très lisible. Avec un peu d'effort en programmation, on peut écrire des fonctions plus évoluées pour réaliser les mêmes traitements. Via les fonctions statgh, une seule instruction suffit, qui fournit aussi l'analyse statistique de la variable et trois autres graphiques, nommée decritQT() :
decritQT("Longueurs LEAdb",lng,"aa",TRUE)
##########################################################
#
# en mode long :
#
##########################################################
decritQT(
titreQT="Longueurs LEAdb",
nomVar=lng,
unite="aa",
lng="FR",
graphique=TRUE
) # fin de decritQT
DESCRIPTION STATISTIQUE DE LA VARIABLE Longeurs LEAdb
Taille 773 individus
Moyenne 205.6882 aa
Ecart-type 148.5962 aa
Coef. de variation 72 %
1er Quartile 130.0000 aa
Mediane 168.0000 aa
3eme Quartile 236.0000 aa
iqr absolu 106.0000 aa
iqr relatif 63.0000 %
Minimum 68 aa
Maximum 1864 aa
Tracé tige et feuilles
The decimal point is 2 digit(s) to the right of the |
0 | 777778888888999999999999999999999999999999999999999999999999999999999999999999
1 | 0000000000000000000000000000000000000000000000011111111111111111111111111111111111111111111122222222222223333333333333+273
2 | 0000000000000000000000000000111111111111111122222222222222333333333333333333333333333333333333333444444444444444444444+57
3 | 000000011111222223333344444445556666788889999
4 | 0011111112223444556666777778889
5 | 0122455667888
6 | 22344588
7 | 234
8 | 5
9 |
10 |
11 |
12 | 4
13 |
14 | 3
15 | 1
16 |
17 |
18 | 6
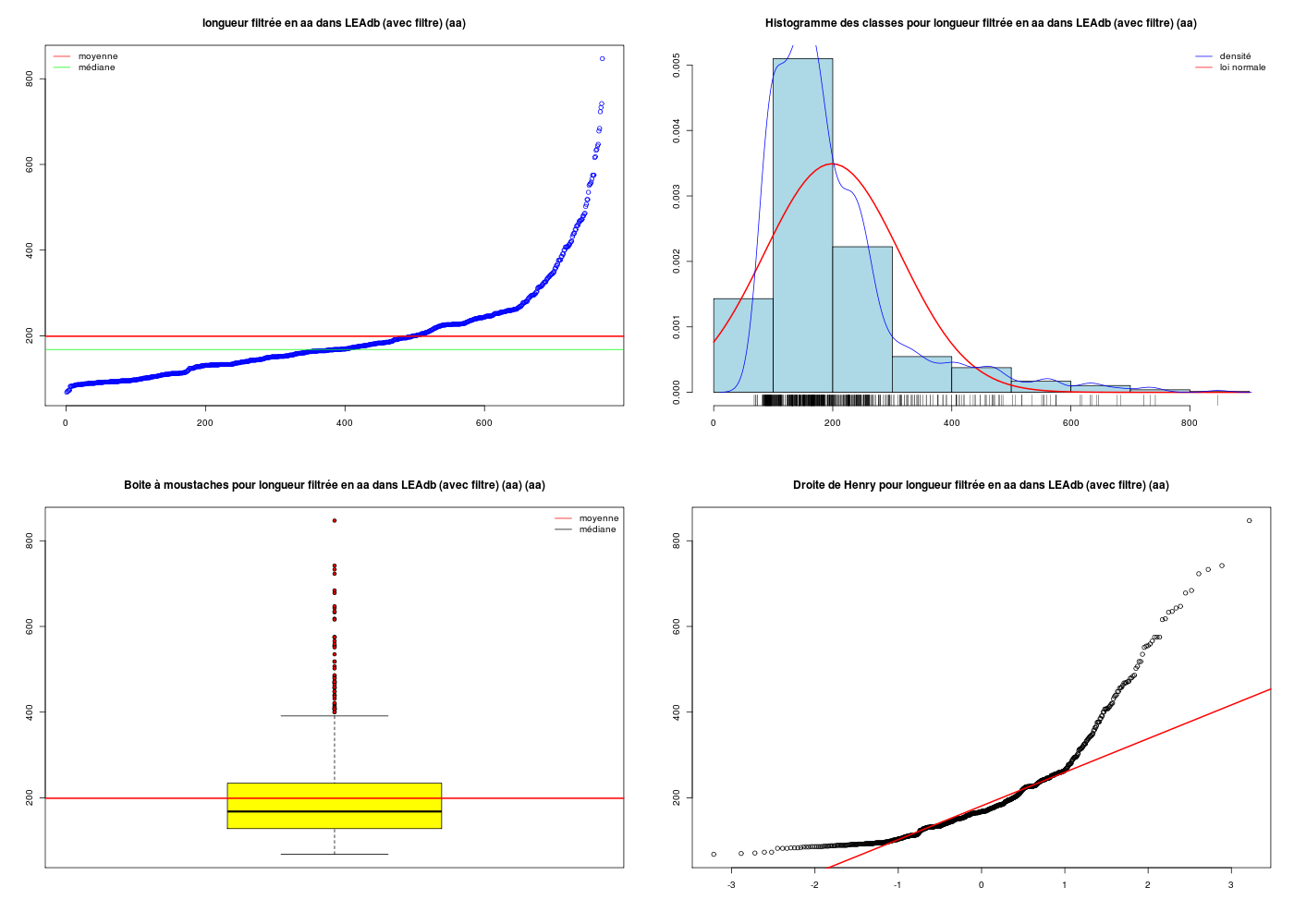
S'il semble très facile de tracer des histogrammes, des courbes de densité, les calculs et les choix (nombre de classes, valeur de la densité...) font appel à des connaissances statistiques avancées. Voir par exemple la page density estimation, sans doute plus intéressante que la page estimation par noyau dont une application intéressante est en géographie humaine ici.
2. Exécution de code R via PHP
Peut-on faire exécuter du code R par PHP ?
Peut-on récupérer dans un fichier le résultat de cette exécution ?
La réponse est oui, bien évidemment, puisque PHP peut exécuter des commandes et lancer des exécutables via l'instruction system().
Par exemple, si le fichier demo.r contient le code :
x <- 1
cat(" la variable x vaut ",x,"\n")
alors le programme PHP suivant, nommé phpetr01.php
<?php
echo "<pre>\n" ;
system("R --no-save < demo.r") ;
echo "<pre>\n" ;
?>
produit exactement ce que vous pensez. Il suffit de cliquer ici pour le vérifier. L'option --no-save (qui n'est sans doute pas suffisante) a pour but de ne pas charger l'environnement R et donc de fournir une exécution reproductible.
Tout cela n'est pas très paramétré. Rajoutons donc la redirection des sorties dans un fichier temporaire. Vérification ici à l'aide du programme phpetr02.php. On notera le passage de l'option --no-save à l'option --vanilla.
<?php
echo "<pre>\n" ;
$fs = tempnam("/tmp","demo.res") ;
echo "<!-- le fichier est $fs -->\n" ;
system("R --vanilla < demo.r > $fs") ;
$res = file_get_contents($fs) ;
echo $res ;
echo "<pre>\n" ;
?>
Bien sûr, ceci n'est pas très "propre". A l'aide des options d'exécution de R on peut faire nettement mieux, comme on le voit ici à l'aide du programme phpetr03.php
<?php
echo "<pre>\n" ;
$fs = tempnam("/tmp","demo.res") ;
echo "<!-- le fichier est $fs -->\n" ;
system("R --vanilla --slave < demo.r > $fs") ;
$res = file_get_contents($fs) ;
echo $res ;
echo "<pre>\n" ;
?>
Enfin, pour produire une "vraie" page Web, un include et quelques fontions conceptuelles via le programme phpetr04.php
<?php
include("std.php") ;
debutPage("Intégration R et PHP") ;
debutSection() ;
$fr = "demo.r" ;
h1("Voici le fichier R utilisé (".href($fr).") :") ;
pre_fichier($fr,"cadrejaune") ;
h1("Voici le résultat de l'exécution :") ;
$fs = tempnam("/tmp","demo.res") ;
echo "<!-- le fichier est $fs -->\n" ;
system("R --vanilla --slave < $fr > $fs") ;
pre_fichier($fs,"cadrebleu") ;
finSection() ;
finPage() ;
?>
assurent un rendu valide XHTML Strict habillé en CSS comme le montre la page phpetr04.php.
3. Production de calculs et graphiques à la volée en R et PHP
Reprendre l'exercice précédent où les données sont lues dans un formulaire.
Elémentaire, mon cher Watson ! Si les données sont dans un formulaire, une stratégie simple peut être de transférer les données dans un fichier temporaire et de faire exécuter par PHP un script R d'analyse sur ces données.
Dans la mesure où R peut éxécuter les instructions en ligne de commande, il suffit de préparer les commandes à exécuter, de les mettre aussi dans un fichier temporaire et de laisser R tout traiter.
On trouvera à l'URL aqt_det.php une implémentation possible des calculs et des graphiques avec des exemples prêts à l'emploi. Les fichiers utilisés sont calcstat.php, progstat et statuno. L'archive statprog.zip contient ces programmes qu'on essaiera d'utiliser, à défaut de les modifier ou de les réécrire.
Voici comment cela fonctionne : le formulaire de la page aqt_det.php envoie les données et le champ caché calc=aqtdet à la page web calcstat.php en mode post Ce fichier calcstat.php se contente d'utiliser la "bonne" fonction sur les données :
<?php
# ceci est le fichier calcstat.php
error_reporting(E_ALL | E_NOTICE ) ;
include_once("../std7.php") ;
include("progstat.php") ;
debutPage("calculs statistiques simples") ;
debutSection() ;
blockquote() ;
$calc = "?" ;
if (isset($_GET["calc"])) { $calc = $_GET["calc"] ; } ;
if (isset($_POST["calc"])) { $calc = $_POST["calc"] ; } ;
if ($calc=="comppct") { compPct() ; } ;
if ($calc=="compmoy") { compMoy() ; } ;
if ($calc=="chideux") { chiDeux() ; } ;
if ($calc=="deccl") { deccl() ; } ;
if ($calc=="tigef") { tigef() ; } ;
if ($calc=="gdr") { gdr() ; } ;
if ($calc=="comproc") { comproc() ; } ;
if ($calc=="aqtdet") { aqtdet() ; } ;
if ($calc=="smax") { smax() ; } ;
if ($calc=="?") { erreur("calcul") ; } ;
pvide() ;
p() ;
echo href("montresource.php?nomfic=calcstat.php","Code-source de cette page","orange_stim nou")." ; " ;
echo " fichiers inclus : ".href("montresource.php?nomfic=progstat.php","progstat","orange_stim nou")." et " ;
echo " ".href("../montresource.php?nomfic=statuno.php","statuno","orange_stim nou").". " ;
finp() ;
finblockquote() ;
finSection() ;
finPage() ;
exit(-1) ;
?>
Les fonctions utilisées par calcstat.php sont définies dans le fichier inclus progstat. La fonction aqt_det() affiche les données dans un élément <textarea> puis les écrit dans un fichier temporaire dont le nom commence par aqt_det.data. Le reste du nom du fichier est déterminé par la fonction PHP nommée tempnam() de façon à éviter un conflit en cas de calculs concurrents. La fonction aqt_det() prépare aussi les noms des deux fichiers pour les graphiques. Si on met la variable $dbg à 1 dans cette fonction, on peut alors voir à l'éxécution un élément <pre> qui contient quelque chose comme :
ficdat est : /tmp/aqt_det.dataFqFdiZ
ficgr1 est : /home/gh/public_html/wtmp/aqt_det_graph1K6E7yd
pdwtmp vaut 20
fin de nom est donc : /wtmp/aqt_det_graph1K6E7yd
soit, en relatif :
../wtmp/aqt_det_graph1K6E7yd.png
et ../wtmp/aqt_det_graph2EGQ1Pr.png
La fonction aqt_det() vient ensuite mettre dans un tableau PHP les instructions R à exécuter, soit un contenu similaire à :
Array (
[0] => source("statgh.r")
[1] => lesd <- read.table("/tmp/aqt_det.dataFqFdiZ")
[2] => attach(lesd)
[3] => vqtGH <- V1
[4] => cat("STAT_DEB")
[5] => dtQT("DataWeb",vqtGH,"kg",TRUE,
"/home/gh/public_html/wtmp/aqt_det_graph1K6E7yd.png",
"/home/gh/public_html/wtmp/aqt_det_graph2EGQ1Pr.png")
[6] => cat("STAT_FIN")
)
avant d'appeler la fonction execute_R() qui est définie dans le fichier statuno. Cette fonction execute_R() transfère les commandes R dans un fichier temporaire avant d'appeler le logiciel R avec une redirection des entrées et des sorties. Une variable $dbg mise à 1 dans cette fonction montre aussi une trace de ce qui se passe.
R --quiet --vanilla < /tmp/statuno_calcstats.rgo3RnU > /tmp/statuno_calculs.sorSEiNE8 2>> /tmp/statuno_calculs.sorSEiNE8
Comme on peut le voir à l'instruction [5], PHP demande à R de mettre les graphiques dans des fichiers dont le nom a été généré par tempnam(). Le répertoire utilisé, soit tmp pour le est en fait un répertoire local de l'utilisateur, soit /home/gh/public_html/wtmp dont les droits sont 777 afin que le serveur Web puisse y écrire. Pour PHP il s'agit donc d'un répertoire relatif, lié au serveur Web alors que pour R il s'agit d'un répertoire local, définir par son chemin absolu. Pour que PHP puisse ensuite afficher les images, il faut modifier le chemin d'accès aux images.
La fonction que R utilise pour traiter les données est nommée dtQt() et fait partie de nos fonctions R.
4. Statistiques et graphiques sur des données Excel
Reprendre les mêmes calculs et graphiques juste avec la colonne poids du fichier Excel her_extrait.xls puis avec toutes les colonnes. Les unités respectives sont les années, les kilos et les centimètres. Les données sont un extrait du dossier HER. On pourra utiliser les fonctions de statgh.r.
Est-ce facilement généralisable à tout fichier Excel ?
Il y a deux stratégies possibles : lire les données avec PHP puis effectuer les calculs et graphiques avec R ou tout faire avec R. Dans la mesure où R sait bien lire les fichiers Excel, cette dernière solution est sans doute la meilleure. On trouvera ici et ci-dessous une interface pour choisir le nom de la ou des colonnes et lancer l'analyse.
<?php
# # (gH) -_- f_aqtExcel.php ; TimeStamp (unix) : 24 Décembre 2015 vers 19:25
error_reporting(E_ALL | E_NOTICE ) ;
include("std7.php") ;
$titre = "Formulaire d'analyse de données QT dans un fichier Excel " ;
debutPage($titre,"strict","","") ;
debutSection() ;
h1($titre) ;
$fExcel = "her_extrait.xls" ;
p() ;
echo "Vous pouvez cliquer ".href($fExcel,"ici")." pour voir le fichier Excel nommé ".b($fExcel).". " ;
finp() ;
# ---------------------------------------------------------------------
form("aqtExcel.php") ;
p() ;
echo "Choisissez ce que vous voulez traiter :" ;
nbsp(2) ;
listeSelectFromTxt("varQt","AGE TAILLE POIDS toutes") ;
nbsp(2) ;
echo "puis cliquez sur " ;
input_submit("analyser","","bouton_fin nou vert_pastel") ;
finp() ;
finform() ;
# ---------------------------------------------------------------------
p() ;
echo s_span("Code-source de cette page","orange_stim nou")." : " ;
echo href("montresource.php?nomfic=f_aqtExcel.php","f_aqtExcel.php")."." ;
finp() ;
# ---------------------------------------------------------------------
finSection() ;
finPage() ;
?>
Le traitement en R est alors assez simple puisqu'on peut préparer toutes les commandes R à l'avance. On trouvera en bas de la page aqtExcel (ignorer le message d'erreur affiché) le code-source pour le traitement. Voici par exemple le code R à exécuter pour la variable POIDS :
source("http://forge.info.univ-angers.fr/~gh/wstat/statgh.r",encoding="latin1")
library(gdata)
data <- read.xls("her_extrait.xls")
decritQT("Variable POIDS",data$POIDS,"kg",TRUE,"aqtPOIDS.png")
Pour exécuter cela via PHP on peut utiliser la fonction analyse() suivante :
<?php
# # (gH) -_- aqtExcel.php ; TimeStamp (unix) : 24 Décembre 2015 vers 19:34
error_reporting(E_ALL | E_NOTICE ) ;
function analyse($nom,$unite="?") {
# 1. préparation des noms de fichier
$fig = "aqt_det_graph" ;
$ficgr = tempnam("../wtmp/",$fig) ;
$png = "$ficgr.png" ;
$pdwtmp = strpos($ficgr,"/wtmp") ;
$fdn = substr($ficgr,$pdwtmp) ;
$fpng = "..$fdn.png" ;
# 2. préparation du tableau des commandes R
$dbg = 1 ; # mettre 1 pour voir le détail si cela "plante"
$nbcmd = -1 ;
$tcmdR = array() ;
$nbcmd++ ; $tcmdR[$nbcmd] = ' source("http://forge.info.univ-angers.fr/~gh/wstat/statgh.r") ' ;
$nbcmd++ ; $tcmdR[$nbcmd] = " library(\"gdata\",lib.loc=\"../rpkgs\") \n" ;
$nbcmd++ ; $tcmdR[$nbcmd] = ' data <- read.xls("her_extrait.xls") ' ;
$nbcmd++ ; $tcmdR[$nbcmd] = " cat(\"STAT_DEB\\n\") ; \n" ;
$nbcmd++ ; $tcmdR[$nbcmd] = " cat(\"\\n\") \n" ;
$nbcmd++ ; $tcmdR[$nbcmd] = " decritQT('Variable $nom',data['$nom'],'$unite',TRUE,'$png') \n" ;
$nbcmd++ ; $tcmdR[$nbcmd] = " cat(\"\\n\") \n" ;
$nbcmd++ ; $tcmdR[$nbcmd] = " cat(\"STAT_FIN\\n\") ; \n" ;
if ($dbg==1) {
p() ;
echo " on utilise la fonction ".b("execute_R()")." avec comme paramètre le tableau suivant " ;
finp() ;
pre() ;
print_r($tcmdR) ;
finpre() ;
} ; # fin si
# 3. exécution du tableau des commandes R
pre() ;
execute_R($tcmdR,$dbg) ; # la fonction execute_R est dans statuno.php
finpre() ;
# 4. affichage du graphique
pvide() ;
p() ;
echo href($fpng,img($fpng,"png",600)) ;
finp() ;
} ; # fin de fonction analyse
?>
Sur le principe, cet exercice est facilement généralisable : il suffit de choisir un fichier Excel, de trouver le nom des colonnes et demander quelle colonne on veut traiter. En pratique, l'analyse est infaisable car toutes les colonnes ne sont pas analysables au sens des variables quantitatives. Que faire, par exemple de la colonne 1 (IDEN) du fichier her_extrait.xls ? De plus, il manquerait aussi l'unité sans laquelle l'analyse n'a pas de sens. Supposer que tous les fichiers Excel puissent être formatés avec un codage adapté (ligne 1, nom des colonnes ; ligne 2, indication quantitative ou non ; ligne 3 unité éventuelle) est irréaliste.
5. Statistiques et graphiques sur des données issues d'Internet
Reprendre maintenant avec les données de la page paysUE dont une copie locale avec code source "aménagé" est ici. On fournira un tableau avec le nom des pays, leur superficie et leur population. Ce tableau sera triable. Afficher ensuite un graphique des superficies et des populations. Vous trouverez à l'adresse paysUE.php un exemple de ce que l'on doit obtenir.
Exerice non corrigé volontairement. L'accès au code-source de page solution paysUE.php sera expliqué en TP.
6. Tracé d'un graphe avec R
Exécuter le code R suivant et expliquer ce que fait chaque ligne non vide.
# éventuellement : install.packages("igraph",dependencies=TRUE,repos="http://cran.univ-lyon1.fr/")
library(igraph)
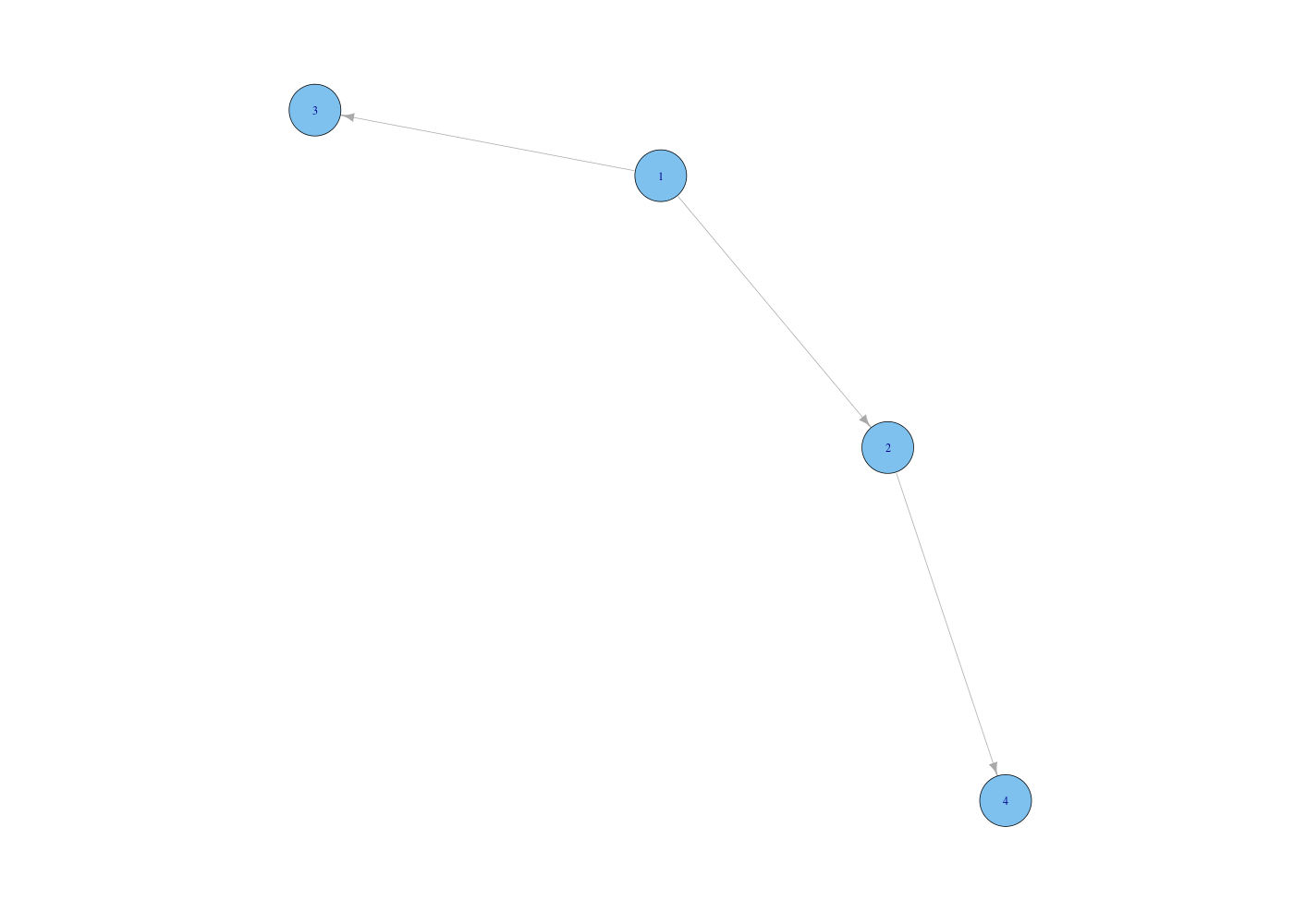
gxhtml <- graph(edges=c(1,2,1,3,2,4),directed=TRUE)
V(gxhtml)$name <- c("html","head","body","title")
print( get.adjacency(gxhtml) )
plot(gxhtml)
Ecrire ensuite une fonction R qui lit un fichier de listes d'adjacence, qui construit l'objet graphe associé puis qui affiche sa matrice d'adjacence et en donne une représentation graphique. On utilisera le package igraph.
Commençons par numéroter les lignes du script R :
0001 # éventuellement : install.packages("igraph",dependencies=TRUE,repos="http://cran.univ-lyon1.fr/")
0002
0003 library(igraph)
0004
0005 gxhtml <- graph(edges=c(1,2,1,3,2,4),directed=TRUE)
0006 V(gxhtml)$name <- c("html","head","body","title")
0007
0008 print( get.adjacency(gxhtml) )
0009
0010 plot(gxhtml)
La ligne 1 est un commentaire, ignorée par R. Elle est mise ici pour rappeler comment installer le package igraph. La ligne 3 charge ce package. S'il n'est pas installé, R ne va plus loin. La ligne 5 crée un objet de type igraph nommé gxhtml -- ce qu'on peut vérifier avec l'instruction class(gxhtml). Il s'agira d'un graphe orienté à cause de directed=TRUE et egdes=... indique les arcs du graphe. Les valeurs numériques correspondent aux numéros des sommets (ou vertex en anglais). Par défaut, ceux-ci ont comme label leur numéro. La ligne 6 permet de changer ces labels pour les valeurs "html","head","body","title". La ligne 8 affiche la matrice d'adjacence du graphe et la ligne 10 l'affiche graphiquement. En fait, la fonction plot de la ligne 10 fait appel à la fonction plot.igraph du package.
Voici le résultat de l'exécution de ces lignes :
4 x 4 sparse Matrix of class "dgCMatrix"
html head body title
html . 1 1 .
head . . . 1
body . . . .
title . . . .
Et le graphique produit :
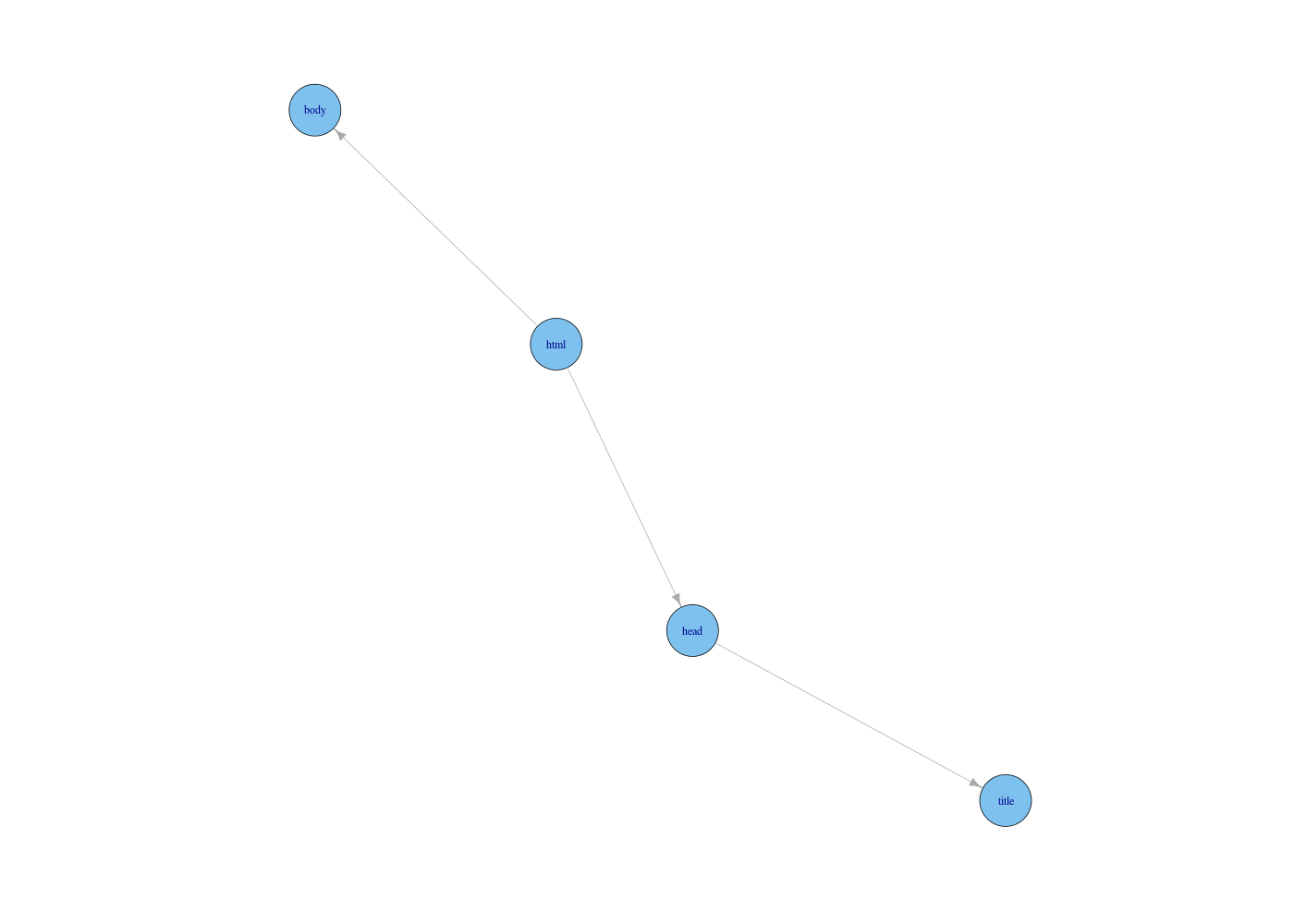
On peut se servir de la solution PHP de la série précédente d'exercices comme trame de la fonction showGraph() en R. Voici une implémentation possible :
showGraph <- function (nomFichier="") {
# aide éventuelle
if (missing(nomFichier) | (nomFichier=="")) {
cat("\n")
cat(" syntaxe : showGraph(fichier)\n")
cat("\n")
return( invisible(NULL)) ;
} # finsi
# si le fichier n'existe pas, on le dit et on sort
if (!file.exists(nomFichier)) {
cat("fichier ",nomFichier," non vu. STOP.\n")
cat("\n")
return( invisible(NULL)) ;
} # finsi
# lecture du fichier, on en déduit les noms des sommets
cat("\nGraphe défini par les listes d'adjacence de ",nomFichier,"\n\n")
ladj <- readLines(nomFichier)
nbs <- length(ladj)
sommets <- rep(NA,nbs)
for (iliste in seq(ladj)) {
# pour debug : print(iliste)
listec <- unlist(strsplit(ladj[iliste],"\\s+",perl=TRUE))
# pour debug : print(listec)
sommets[iliste] <- listec[1]
cat(" sommet numéro ",sprintf("%2d",iliste)," : ",sommets[iliste],"\n")
} # fin pour liste
# pour debug : cat(" voici la liste des sommets : \n") ; print(sommets)
# construction du vecteur "edges", version lente
gedges <- c() # initialisation
for (iliste in seq(ladj)) {
listec <- unlist(strsplit(ladj[iliste],"\\s+",perl=TRUE))
for (ivertex in seq(listec)) {
if (ivertex==1) {
dep <- iliste
} else {
gedges <- c(gedges,dep,which(sommets==listec[ivertex]))
} # fin si
} # fin pour ivertex
} # fin pour liste
# pour debug : cat(" voici la liste des arcs : \n") ; print(gedges)
# on peut alors construire le graphe et l'analyser, le tracer...
newGraph <- graph( edges=gedges, directed=TRUE)
V(newGraph)$names <- sommets
print( get.adjacency(newGraph) )
plot(newGraph)
return( invisible(newGraph)) ;
} # fin de fonction showGraph
Et des exemples d'utilisation :
> showGraph()
syntaxe : showGraph(fichier)
> showGraph("")
syntaxe : showGraph(fichier)
> showGraph("pomme")
fichier pomme non vu. STOP.
> showGraph("listeadj.txt")
Graphe défini par les listes d'adjacence de : listeadj.txt
sommet numéro 1 : 0
sommet numéro 2 : 1
sommet numéro 3 : 2
sommet numéro 4 : 3
sommet numéro 5 : 4
sommet numéro 6 : 5
sommet numéro 7 : 6
sommet numéro 8 : 7
8 x 8 sparse Matrix of class "dgCMatrix"
[1,] . 1 . . 1 . . .
[2,] . . 1 . . . 1 .
[3,] . . . . . . . .
[4,] . . 1 . . . 1 .
[5,] . . . . . . . .
[6,] 1 . 1 . . . . .
[7,] . . . . . . . .
[8,] . . . . . . 1 .
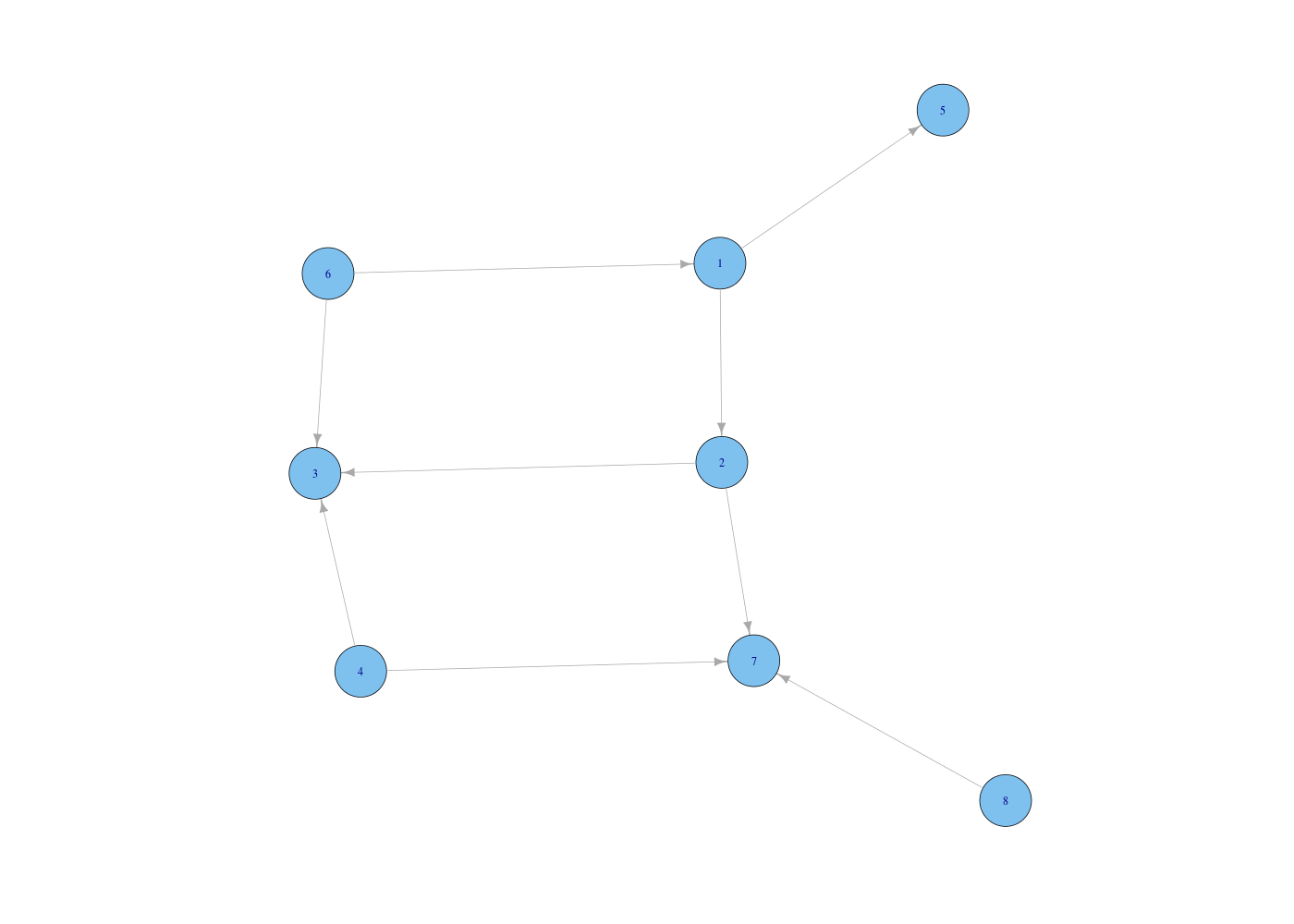
> showGraph("xhtml.txt")
Graphe défini par les listes d'adjacence de : xhtml.txt
sommet numéro 1 : html
sommet numéro 2 : head
sommet numéro 3 : body
sommet numéro 4 : title
4 x 4 sparse Matrix of class "dgCMatrix"
[1,] . 1 1 .
[2,] . . . 1
[3,] . . . .
[4,] . . . .
Avec les graphiques associés :
Code-source de cette page ; fichiers inclus : pagsd_inc.php et pagsd.js.
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)




