DECRA, T.P. 3 :
Programmation en R
Décomposition, Conception et Réalisation d'Applications
gilles.hunault "at" univ-angers.fr
1. Tuteur R, Rstudio et Jupyter notebook
Partie 1.1
Commencer par relire le tuteur de programmation R.
Pour les plus courageuses et les plus courageux, essayer de répondre aux dix exercices de la section 1. I don't know R de la R «warm-up session». Où sont les solutions ?
Partie 1.2
Reproduire ensuite le tableau des puissances inférieures ou égales à 1 million. On nommera boucle.r le fichier correspondant qu'on exécutera d'abord dans Rstudio. Comment exporter le résultat pour obtenir le fichier boucle.txt suivant :
table des puissances inférieures ou égales à 1 million
1 1 1 1 1 1 1 1 1 1
2 4 8 16 32 64 128 256 512 1024
3 9 27 81 243 729 2187 6561 19683 59049
4 16 64 256 1024 4096 16384 65536 262144
5 25 125 625 3125 15625 78125 390625
6 36 216 1296 7776 46656 279936
7 49 343 2401 16807 117649 823543
8 64 512 4096 32768 262144
9 81 729 6561 59049 531441
10 100 1000 10000 100000 1000000
Serait-ce plus simple à exécuter et à exporter dans un terminal ?
Serait-ce plus simple à exécuter et à exporter via un Jupyter Notebook ? Pour mémoire, dans les salles du département informatique, il faut, dans un terminal lancer la commande _jupyter_notebook c'est-à-dire jupyter_notebook précédé du souligné _ pour pouvoir utiliser un serveur local de notebooks. Le disque D et le répertoire Mes_notebooks permettent alors d'enregistrer les fichiers.
Partie 1.3
Reproduire ensuite les calculs liés à microbenchmark( a <- runif(n), b <- rnorm(n) ) puis ceux liés à l'optimisation de sum(v==maxv) et de which(v==maxv). Est-ce normal d'avoir des résultats différents du tuteur ?
Partie 1.4
Télécharger puis compléter le Jupyter Notebook nommé introduction-a-R.
gilles.hunault@g105-0:~$ cd /media/D/Mes_notebooks/
gilles.hunault@g105-0:~$ wget http://forge.info.univ-angers.fr/~gh/Notebooks/introduction-a-R.ipynb
gilles.hunault@g105-0:~$ _jupyter_notebook
Il n'y a aucune question pour la partie 1.1.
Les solutions de la R «warm-up session» sont cachées sous le bouton bleu nommé play tout en bas de la page, ce qui correspond au fichier answers.txt. On peut alors exécuter chacune des instructions données en solution.
Au niveau de la partie 1.2, il faut commencer par écrire le code R nommé boucle.r suivant
## (gH) -_- boucle.r ; TimeStamp (unix) : 26 Septembre 2018 vers 18:47
# boucles imbriquées et BREAK
cat("table des puissances inférieures ou égales à 1 million\n")
for (indi in 1:10) {
for (indj in 1:10) {
puiss <- indi**indj
if (puiss<=10**6) {
cat(sprintf("%9d",puiss))
} else {
break
} # fin si
} # fin pour indj
cat("\n")
} # fin pour indi
Sous Rstudio, on peut copier/coller ou exporter les lignes affichées, mais ce n'est pas très professionnel. Il est préférable de passer par le couple sink/source comme suit :
fs <- "boucle.txt"
unlink(fs)
sink(fs,split=TRUE)
source("boucle.r",encoding="latin1",print.eval=TRUE)
sink()
cat("\n\n le fichier ",fs," contient les résultats...\n")
Par contre en ligne de commandes, une simple redirection de la sortie suffit
$gh> Rscript --encoding=latin1 boucle.r > boucle.txt
$gh> cat boucle.txt
table des puissances inférieures ou égales à 1 million
1 1 1 1 1 1 1 1 1 1
2 4 8 16 32 64 128 256 512 1024
3 9 27 81 243 729 2187 6561 19683 59049
4 16 64 256 1024 4096 16384 65536 262144
5 25 125 625 3125 15625 78125 390625
6 36 216 1296 7776 46656 279936
7 49 343 2401 16807 117649 823543
8 64 512 4096 32768 262144
9 81 729 6561 59049 531441
10 100 1000 10000 100000 1000000
Il n'est pas plus simple d'utiliser un Jupyter notebook ici.
Si, pour la partie la partie 1.3, on écrit le code R nommé performance.r suivant
library("microbenchmark")
n <- 3*10**5
microbenchmark( a <- runif(n), b <- rnorm(n) )
f <- function(v) {
return(list( sum(v==max(v)),which(v==max(v)) ))
} # fin de fonction f
g <- function(v) {
maxv <- max(v)
return(list( sum(v==maxv), which(v==maxv) ))
} # fin de fonction g
v <- runif(10**6)
microbenchmark(
v <- runif(10**6),
f(v) ,
g(v)
) # fin de microbenchmark
il y a de fortes chances pour qu'il ne fonctionne pas, parce qu'il faut d'abord installer le package microbenchmark. Là, le plus simple est certainement de passer par Rstudio, via l'onglet Package du panneau en bas et à droite : on clique sur le sous-onglet Install, on met le mot microbenchmark dans la zone de saisie texte et il suffit de cliquer sur le bouton Install pour disposer du package. Notre script s'exécute alors correctement.
Voici le résultat de son exécution (au bout d'environ 10 secondes en moyenne sur nos ordinateurs) :
R --vanilla --quiet --slave --encoding=latin1 --file=performance.r
Unit: milliseconds
expr min lq mean median uq max neval
a <- runif(n) 9.521207 9.525817 11.67906 9.849912 10.09587 22.75988 100
b <- rnorm(n) 22.943703 22.973644 24.66577 23.241508 23.58944 36.49613 100
cld
a
b
Unit: milliseconds
expr min lq mean median uq max
v <- runif(10^6) 30.625618 31.052000 36.42614 31.388036 31.81875 83.44705
f(v) 9.102046 9.992811 16.47826 10.397777 11.02663 62.71794
g(v) 7.917192 8.682063 19.10798 9.036907 10.11800 61.10956
neval cld
100 b
100 a
100 a
Il est normal qu'on n'ait pas exactement les mêmes résultats que dans le tuteur parce qu'on utilise des valeurs aléatoires et parce que l'exécution du code dépend de l'état de la machine...
Partie 1.4 : le code complet associé au Jupyter notebook nommé introduction-a-R se nomme introduction-a-R-solution. A lire sans modération !
2. Exercices simples d'entrainement pour "snobber" Excel
Partie 2.1
Ecrire un script R nommé extraitFichierExcel.r qui affiche les cinq premières lignes et les cinq dernières lignes d'un fichier Excel dont le nom est passé en paramètre, par exemple le fichier iris.xlsx. Exemple de fonctionnement :
$gh> Rscript --encoding=latin1 --vanilla extraitFichierExcel.r iris.xlsx
Extrait des 150 lignes et des 6 colonnes du fichier Excel iris.xlsx
--------------------------------------------------------------------
iden espece longsep largsep longpet largpet
1 I001 1 5.1 3.5 1.4 0.2
2 I002 1 4.9 3.0 1.4 0.2
3 I003 1 4.7 3.2 1.3 0.2
4 I004 1 4.6 3.1 1.5 0.2
5 I005 1 5.0 3.6 1.4 0.2
[...]
iden espece longsep largsep longpet largpet
146 I146 3 6.7 3.0 5.2 2.3
147 I147 3 6.3 2.5 5.0 1.9
148 I148 3 6.5 3.0 5.2 2.0
149 I149 3 6.2 3.4 5.4 2.3
150 I150 3 5.9 3.0 5.1 1.8
Partie 2.2
Ecrire un script bash et une fonction R nommés tous deux decritFichierExcel qui utilisent un paramètre nommé fichierExcel avec le comportement suivant :
-
s'il n'y a pas de paramètre, on fournit un rappel de la syntaxe et un exemple puis on s'arrête.
-
si le fichier Excel désigné par le paramètre n'est pas présent, on l'indique et on s'arrête.
-
si le fichier est présent, on liste les colonnes présentes avec leur numéro et leur nom sous Excel (A pour 1, B pour 2...).
-
on utlisera un try/catch pour garantir que la lecture des données s'est bien passée.
Pour les expert(e)s, on pourra de plus fournir le type des colonnes, la valeur minimale, la valeur maximale et le nombre de valeurs manquantes, comme ci-dessous, pour le fichier iris.xlsx :
$gh> decritFichierExcel iris.xls
Description du fichier iris.xls
===============================
150 lignes et 6 colonnes lues dans ce fichier.
Numéro Colonne Excel NbVal Min Max Distinct Manquantes Type
iden 1 A 150 I001 I150 150 0 character
espece 2 B 150 1.000 3.000 3 0 numeric
longsep 3 C 150 4.300 7.900 35 0 numeric
largsep 4 D 150 2.000 4.400 23 0 numeric
longpet 5 E 150 1.000 6.900 43 0 numeric
largpet 6 F 150 0.100 2.500 22 0 numeric
Partie 2.3
Dès qu'on dépasse plusieurs centaines de lignes dans un fichier Excel, il est difficile d'écrire et de recopier des formules. Par exemple, pour le fichier iris.xlsx, il est un peu fastidieux de créer une colonne grandesPetales avec la valeur 0 si largpet est strictement inférieure à 2 et égale à 1 ailleurs. Avec R, c'est un jeu d'enfants. La preuve ? Ecrivez le script correspondant... On nommera irisG.xlsx le fichier Excel résultat.
Voici ce qu'on devrait obtenir :
$gh> R --vanilla --quiet --slave --encoding=latin1 --file=irisg.r
Ajout de la colonne grandesPetales
Distribution de cette colonne :
0 1
121 29
Vous pouvez utiliser le fichier irisG.xlsx qui contient
iden espece longsep largsep longpet largpet grandesPetales
1 I001 1 5.1 3.5 1.4 0.2 0
2 I002 1 4.9 3.0 1.4 0.2 0
3 I003 1 4.7 3.2 1.3 0.2 0
4 I004 1 4.6 3.1 1.5 0.2 0
5 I005 1 5.0 3.6 1.4 0.2 0
6 I006 1 5.4 3.9 1.7 0.4 0
[...]
-rw-rw-r-- 1 gh gh 10980 oct. 10 17:29 irisG.xlsx
Facile, c'est du R !
Pour la partie 2.1, sans aucune vérification du paramètre, on peut se contenter du code extraitFichierExcel.r suivant :
## (gH) -_- extraitFichierExcel.r ; TimeStamp (unix) : 10 Octobre 2018 vers 17:00
library("gdata") # ou (library("openxlsx") si c'est possible
args <- commandArgs(trailingOnly = TRUE)
nomFicExcel <- args[1]
lesDonnees <- read.xls(fichierExcel) # read.xlsx(fichierExcel) avec le package openxlsx
titre <- paste("Extrait des ",nrow(lesDonnees),"lignes et des",ncol(lesDonnees),"colonnes du fichier Excel",nomFicExcel)
cat("\n")
cat(titre,"\n")
cat(paste(rep("-",nchar(titre)),collapse=""),"\n")
cat("\n")
print(head(lesDonnees,n=5))
cat("[...]\n")
print(tail(lesDonnees,n=5))
Pour la partie 2.2, il faut écrire un fichier script et un fichier R. Nous choisissons ici d'utiliser une syntaxe longue d'appel de R. C'est R qui gère le paramètre...
Code du fichier decritFichierExcel rendu exécutable sous Linux via chmod +x :
R --vanilla --quiet --slave --encoding=latin1 --file=decritFichierExcel.r --args $1
Code du fichier R nommé decritFichierExcel.r :
## (gH) -_- decritFichierExcel.r ; TimeStamp (unix) : 24 Septembre 2018 vers 13:12
#########################################################################
#
# attention, ce programme s'utilise en ligne de commandes !
#
#########################################################################
library("gdata") # ou (library("openxlsx") si c'est possible
library("attempt")
options(width=230) # il y a parfois beaucoup de colonnes sous Excel...
source("decritFichierExcel-inc.r") # sous-programmes
#########################################################################
#
# programme principal
#
#########################################################################
args <- commandArgs(trailingOnly = TRUE)
argc <- length(args)
erreur <- 0
if (argc<1) {
erreur <- 1
cat("\nNombre de paramètres incorrect.\n")
cat("Vous devez fournir le nom du fichier Excel à décrire.\n\n")
} else {
## 2. vérification de l'existence du fichier
fichierExcel <- args[1]
if (!file.exists(fichierExcel)) {
msgErreur <- paste("Fichier modèle \"",fichierExcel,"\" non vu.\n")
cat(paste("Vous avez fourni",fichierExcel," comme nom de fichier Excel.\n"))
cat(paste("Mais... ce fichier n'existe pas dans le répertoire courant ",getwd()),"\n")
} # fin si test sur l'existence du fichier
} # fin si sur argc
if (erreur>0) {
stop("Impossible de continuer. STOP\n")
} # fin si sur erreur
## 3. si on arrive ici, on peut tenter l'aventure !
cats(paste("Description du fichier",fichierExcel))
erreur <- 0
try_catch(
ongletData <- read.xls(fichierExcel) , # read.xlsx(fichierExcel) avec le package openxlsx
.e = function(e) {
cat(paste("Problème de lecture dans le fichier Excel ",fichierExcel,". Abandon des calculs.STOP.\n\n",sep=""))
stop("Impossible de continuer. STOP\n")
} # fin de fonction anonyme
) # fin de try_catch
cat(paste(nrow(ongletData),"lignes et",ncol(ongletData),"colonnes lues dans ce fichier.\n\n"))
numNom <- sapply(FUN=toColExcel,X=1:ncol(ongletData))
descData <- lesColonnes(ongletData,ordered=FALSE,envar=TRUE,print=FALSE)
descData <- cbind(descData[,1],numNom,descData[,2:ncol(descData)])
names(descData)[1:2] <- c("Numéro","Colonne Excel")
print(descData)
# fin de script decritFichierExcel.r
Vérification du fonctionnement :
$gh> ./decritFichierExcel
Nombre de paramètres incorrect.
Vous devez fournir le nom du fichier Excel à décrire.
Erreur : Impossible de continuer. STOP
Exécution arrêtée
$gh> ./decritFichierExcel pomme.xls
Vous avez fourni pomme.xls comme nom de fichier Excel.
Mais... ce fichier n'existe pas dans le répertoire courant /home/gh/public_html/Decra
Description du fichier pomme.xls
================================
Problème de lecture dans le fichier Excel pomme.xlsx. Abandon des calculs.STOP.
Error in value[[3L]](cond) : Impossible de continuer. STOP
Calls: try_catch ... tryCatch -> tryCatchList -> tryCatchOne -> <Anonymous>
Exécution arrêtée
$gh> ./decritFichierExcel irisG.xls
Description du fichier irisG.xls
================================
150 lignes et 7 colonnes lues dans ce fichier.
Numéro Colonne Excel NbVal Min Max Distinct Manquantes Type
iden 1 A 150 I001 I150 150 0 character
espece 2 B 150 1.000 3.000 3 0 numeric
longsep 3 C 150 4.300 7.900 35 0 numeric
largsep 4 D 150 2.000 4.400 23 0 numeric
longpet 5 E 150 1.000 6.900 43 0 numeric
largpet 6 F 150 0.100 2.500 22 0 numeric
grandesPetales 7 G 150 0.000 1.000 2 0 numeric
$gh> Rscript --encoding=latin1 --vanilla extraitFichierExcel.r irisG.xls
Extrait des 150 lignes et des 7 colonnes du fichier Excel irisG.xls
--------------------------------------------------------------------
iden espece longsep largsep longpet largpet grandesPetales
1 I001 1 5.1 3.5 1.4 0.2 0
2 I002 1 4.9 3.0 1.4 0.2 0
3 I003 1 4.7 3.2 1.3 0.2 0
4 I004 1 4.6 3.1 1.5 0.2 0
5 I005 1 5.0 3.6 1.4 0.2 0
[...]
iden espece longsep largsep longpet largpet grandesPetales
146 I146 3 6.7 3.0 5.2 2.3 1
147 I147 3 6.3 2.5 5.0 1.9 0
148 I148 3 6.5 3.0 5.2 2.0 1
149 I149 3 6.2 3.4 5.4 2.3 1
150 I150 3 5.9 3.0 5.1 1.8 0
Le code du fichier inclus R nommé decritFichierExcel-inc.r est listé ci-dessous :
## (gH) -_- decritFichier-inc.r ; TimeStamp (unix) : 10 Octobre 2018 vers 17:43
#################################################################
cats <- function(chen="",soulign="=") { # sert à souligner
#################################################################
cat("\n")
cat(chen,"\n")
cat(copies(soulign,nchar(chen)),"\n")
cat("\n")
} # fin de fonction cats
#################################################################
copies <- function(string,nrep=0) {
#################################################################
# renvoie n copies de la chaine string
res <- ""
if (nrep>0) { for (idc in (1:nrep)) { res <- paste(res,string,sep="") } }
return(res)
} # fin de fonction copies
###################################################
toColExcel <- function(numCol) {
####################################################
# convertit un numéro de colonne, comme 30
# en une notation Excel, soit ici AD
if (numCol<1) { return("") }
res <- c()
while(numCol>0) {
rst <- (numCol-1) %% 26
res <- c(rst,res)
numCol <- (numCol-rst) %/% 26
} # fin tant que
return(paste(LETTERS[res+1],collapse=""))
} # fin de fonction toColExcel
#################################################################
lesColonnes <- function(df,ordered=TRUE,envar=FALSE,print=TRUE,title="") {
#################################################################
# lesColonnes(df) : les variables sont par ordre alphabétique
# lesColonnes(df,ordered=FALSE) : les variables sont par numéro de colonne croissant
# si envar=TRUE on renvoie la variable du tableau des colonnes
# si print=TRUE on affiche
nbc <- ncol(df)
nbl <- nrow(df)
if (print) {
cat("Voici l'analyse des ",nbc,"colonnes (stat. pour ",nbl," lignes en tout)")
if (title!="") {
cat(" pour ",title)
} # fin si
cat("\n\n")
} # fin si
noms <- names(df)
nbvs <- rep(NA,nbc)
mins <- rep(NA,nbc)
maxs <- rep(NA,nbc)
dist <- rep(NA,nbc)
miss <- rep(NA,nbc)
miss <- rep(NA,nbc)
typs <- rep("",nbc)
ddc <- cbind((1:nbc),nbvs,mins,maxs,dist,miss,typs)
row.names(ddc) <- noms
colnames(ddc) <- c("Num","NbVal","Min","Max","Distinct","Manquantes","Type")
pdc <- 1:nbc
for (idc in pdc) {
laCol <- df[,idc]
laCol <- laCol[!is.na(laCol)]
ddc[idc,2] <- length(laCol)
if (length(laCol)==0) {
ddc[idc,3] <- NA
ddc[idc,4] <- NA
} else {
if (is.factor(laCol)) {
lacoln <- as.numeric(laCol)
ddc[idc,3] <- levels(laCol)[min(lacoln)]
ddc[idc,4] <- levels(laCol)[max(lacoln)]
} else {
if (is.numeric(laCol)) {
ddc[idc,3] <- sprintf("%0.3f",min(laCol))
ddc[idc,4] <- sprintf("%0.3f",max(laCol))
} else {
ddc[idc,3] <- substr(min(laCol),1,10)
ddc[idc,4] <- substr(max(laCol),1,10)
} # fin si
} # fin si
} # fin si
ddc[idc,5] <- length(unique(laCol))
ddc[idc,6] <- sum(is.na(df[,idc]))
ddc[idc,7] <- paste(class(laCol),collapse=" ")
} # fin pour idc
if (ordered) { # tri par ordre alphabétique des noms de variables
idx <- order(noms)
ddc <- ddc[idx,]
} # fin si
if (print) {
print.data.frame(as.data.frame(ddc),quote=FALSE,row.names=TRUE)
cat("\n")
} # fin si
if (envar) {
return(as.data.frame(ddc))
} else {
return()
} # fin de si
} # fin de fonction lesColonnes
Pour la partie 2.3 on peut se contenter du fichier irisg.r suivant :
## (gH) -_- extraitFichierExcel.r ; TimeStamp (unix) : 10 Octobre 2018 vers 17:00
library(openxlsx)
cat("Ajout de la colonne grandesPetales\n")
nomFicExcel <- "iris.xlsx"
lesDonnees <- read.xlsx(nomFicExcel)
lesDonnees <- transform(lesDonnees,
grandesPetales=ifelse(largpet<2,0,1)
) # fin de transform
cat("Distribution de cette colonne :\n")
print(table(lesDonnees$grandesPetales))
write.xlsx(lesDonnees,file="irisG.xlsx")
cat("Vous pouvez utiliser le fichier irisG.xlsx qui contient\n")
print(head(lesDonnees))
cat("[...]\n")
print(system("ls -al irisG.xlsx"))
3. Calcul de co-occurrences en ligne de commandes
On s'intéresse ici à la production d'un tableau dit "tri croisé amélioré" qui comptabilise les croisements de deux variables qualitatives avec des marges correspondant aux pourcentages globaux.
Justifier rapidement que R est un langage adapté à ce problème.
Voici un exemple de fichier de données nommé elf1.txt :
Et le fichier de résultats recherché elf2.txt sachant qu'on s'intéresse aux champs SEXE et ETUD :
Homme Femme pct Etudes
NR 2 1 6.1 %
Primaire 1 5 12.2 %
Secondaire 3 13 32.7 %
Bac 5 4 18.4 %
Supérieur 6 9 30.6 %
pct Sexe 34.7 % 65.3 %
On trouvera la description des données à l'adresse ELF.
On essaiera de produire une solution qui fonctionne quelque soit le tableau de données en entrée. Pour les plus fort(e)s, on produira aussi les histogrammes de fréquences associés, à savoir :
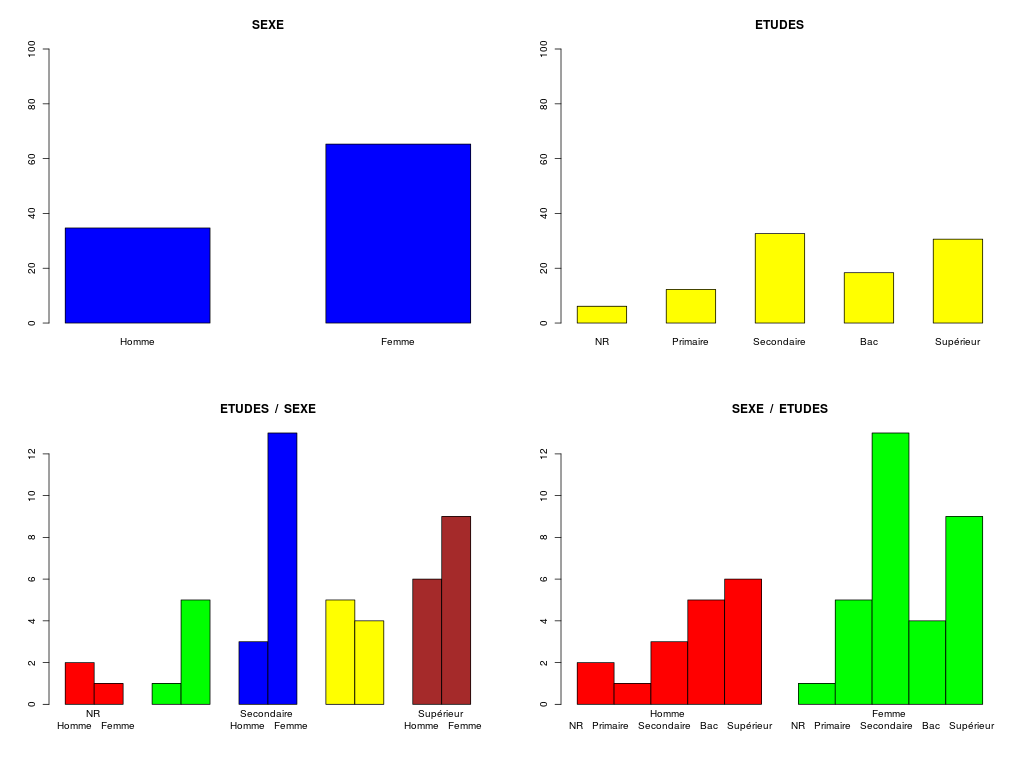
On réfléchira aux extensions et généralisations possibles. Nous fournissons à ce titre, deux fichiers de configuration possibles, nommés elf2Data.xml et autreCfg.xml.
Fichier elf2Data.xml
<TRICROISE>
<variable1>
<nom>SEXE</nom>
<modalites>
<modalite valeur="1">Homme</modalite>
<modalite valeur="2">Femme</modalite>
</modalites>
</variable1>
<variable2>
<nom>EtMarital</nom>
<modalites>
<modalite valeur="0">Seul</modalite>
<modalite valeur="1">En couple</modalite>
</modalites>
</variable2>
</TRICROISE>
Fichier autreCfg.xml
<TRICROISE>
<variable1>
<nom>nivEtudes</nom>
<modalites>
<modalite valeur="0">Sans</modalite>
<modalite valeur="1">Primaire</modalite>
<modalite valeur="2">Secondaire</modalite>
<modalite valeur="3">Bac</modalite>
<modalite valeur="4">Sup</modalite>
</modalites>
</variable1>
<variable2>
<nom>clAGE</nom>
<modalites>
<modalite valeur="0">Enfant</modalite>
<modalite valeur="1">Adulte</modalite>
</modalites>
</variable2>
</TRICROISE>
Remarque : Pour éviter de perdre du temps, on rapatriera en local les fichiers-texte des noms et prénoms via wget.
Pour utiliser R dans une "bonne configuration" dans les locaux du département informatique, vous pouvez utiliser les commandes R, Rscript et rstudio &. Le package XML est le plus adapté pour traiter les fichiers XML. Le plus simple est de l'installer via Rstudio. Il est alors aussi disponible pour une utilisation de R en ligne de commandes.
Il n'est sans doute pas difficile de comprendre ce qu'il faut calculer et tracer. Par contre, savoir quel langage utiliser pour résoudre simplement et rapidement ce problème (il s'agit d'une petite application) n'est pas si simple car calculer des sommes et des pourcentages est à la portée de tout langage de scripts. À nos yeux, un choix raisonnable semble être celui du langage R car il comporte pour chaque action à exécuter dans ces calculs et mises en forme une fonction prête à l'emploi. Comme ce n'est pas simple, il faut penser à écrire un plan de développement, comme par exemple celui défini dans tp3exo3.php.
Voici donc une version minimale nommée co-occur1.r qui traite uniquement l'exemple proposé :
## (gH) -_- co-occur1.r
# on lit le ficher elf1.txt, on ajoute au tri croisé
# des colonnes SEXE et ETUD les pourcentages globaux
# Utilisation :
# Rscript --encoding=latin1 --vanilla co-occur1.r
data <- read.table("elf1.txt",header=TRUE)
sexe <- data$SEXE
etud <- data$ETUD
m_sexe <- c("Homme","Femme")
m_etud <- c("NR","Primaire","Secondaire","Bac","Supérieur")
sex <- factor(sexe,levels=0:1,labels=m_sexe)
etu <- factor(etud,levels=0:4,labels=m_etud)
tc <- table(etu,sex)
pctsSexe <- paste(round(100*prop.table(table(sex)),1),"%")
pctsEtud <- paste(round(100*prop.table(table(etu)),1),"%")
tc <- rbind(tc,pctsSexe)
tc <- cbind(tc,c(pctsEtud," "))
row.names(tc)[1 + length(m_etud)] <- "pct Sexe"
colnames(tc)[ 1 + length(m_sexe)] <- "pct Etud"
print(tc,quote=FALSE)
Quelques commentaires s'imposent, pour une bonne compréhension du script :
-
La lecture des données est assurée par la fonction read.table() du package utils ; le paramètre header indique que la ligne 1 du fichier ne contient pas des données, mais le nom des colonnes.
-
La fonction factor() du package base permet de convertir une colonne de données numériques (qui sont des numéros de modalités) en une «vraie» variable qualitative ou facteur dans la terminologie de R.
-
La fonction table() du package base permet de compter le nombre d'occurrences de chaque modalité («tri à plat») pour un seul paramètre et le nombre d'occurrences de chaque couple de modalités («tri croisé») pour deux paramètres.
-
Pour calculer les pourcentages associés, on passe par la fonction prop.table(), toujours dans le package base.
-
Grâce à la fonction paste() on adjoint le symbole % à ces pourcentages et on les ajoute respectivement en ligne et en colonne via les fonctions rbind() et cbind().
-
Il ne reste plus qu'à mettre les bons noms de lignes et de colonnes respectivement via les fonctions row.names() et colnames() avant d'afficher via la fonction générique print() sans montrer les guillemets à l'aide du paramètre quote car R a converti au passage tout le tableau en chaines de caractères.
Si l'on veut généraliser ce script, la partie facile consiste à rajouter un paramètre correspondant au nom du fichier à traiter. Une partie plus délicate concerne le nom des deux variables, les valeurs numériques et les labels de leurs modalités. Comme cela peut faire beaucoup d'informations à gérer, le mieux est sans doute de mettre ces informations dans un fichiers structuré, disons au format XML ou JSON.
Comme la partie XML est la plus délicate, nous allons commencer par simuler cette partie XML et généraliser tout le reste, en testant tout ce qui doit l'être, comme l'existence des fichiers, des variables. Voici ce que cela donne dans le fichier co-occur2.r qui utilise un autre fichier de données nommé elf2Data.txt :
Fichier elf2Data.txt :
REF AGE EtMarital SEXE
I001 25 1 1
I002 30 0 2
I003 26 1 2
I004 40 1 2
I005 31 0 1
I006 50 1 2
I007 30 0 2
On passe donc à une deuxième version, où les instructions précédentes deviennent une fonction et où des tests garantissent un code robuste.
Fichier co-occur2.r :
### (gH) -_- co-occur2.r
# exemple d'utilisation :
# Rscript --encoding=latin1 --vanilla co-occur2.r
coOccurMarges <- function(dataFile,xmlFile) {
## 1. test du nombre de paramètres
if (nargs()<2) {
cat("\n")
cat("Nombre de paramètres incorrect.\n")
cat("Vous devez fournir un nom de fichier de données et un fichier XML de configuration.\n\n")
cat("Exemple : coOccurMarges(\"elf2Data.txt\",\"elf2Data.xml\")\n\n")
cat("Consulter http://forge.info.univ-angers.fr/~gh/Decra/co-occur.php pour le format des données.\n")
cat("\n")
stop(-1)
} # fin si sur argc
## 2. vérification de l'existence des fichiers
if (!file.exists(dataFile)) {
cat("\n")
cat("Fichier de données",dataFile,"non vu.\n")
stop(-2)
} # fin de si sur dataFile
if (!file.exists(xmlFile)) {
cat("\n")
cat("Fichier de configuration",xmlFile,"non vu.\n")
stop(-3)
} # fin de si sur dataFile
## 3. remplacement de la lecture du fichier XML de configuration par des affectations directes
variable1 <- "SEXE"
variable2 <- "EtMarital"
nums1 <- 1:2
modas1 <- c("Homme","Femme")
nums2 <- 0:1
modas2 <- c("Seul","En couple")
## 4. lecture des données et vérificaton de l'existence des colonnes
data <- read.table(dataFile,header=TRUE)
lesVars <- names(data)
if (!variable1 %in% lesVars) {
cat("Il n'y a pas de colonne nommée",variable1,"dans les données.\n") ;
stop(-4)
} # fin si sur variable1
if (!variable2 %in% lesVars) {
cat("Il n'y a pas de colonne nommée",variable2,"dans les données.\n") ;
stop(-5)
} # fin si sur variable1
## 5. construction du tri croisé amélioré
var1 <- data[,variable1]
var2 <- data[,variable2]
var1f <- factor(var1,levels=nums1,labels=modas1)
var2f <- factor(var2,levels=nums2,labels=modas2)
tc <- table(var1f,var2f)
pctsv1 <- paste(round(100*prop.table(table(var1f)),1),"%")
pctsv2 <- paste(round(100*prop.table(table(var2f)),1),"%")
tc <- rbind(tc,pctsv1)
tc <- cbind(tc,c(pctsv2," "))
row.names(tc)[1+length(modas1)] <- paste("pct",variable1,collapse=" ")
colnames(tc)[ 1+length(modas2)] <- paste("pct",variable2,collapse=" ")
print(tc,quote=FALSE)
} # fin de fonction coOccurMarges
# appel de la fonction
coOccurMarges("elf2Data.txt","elf2Data.xml")
Pour parachever le script, il reste à traiter le fichier XML de configuration. Voici tout d'abord un exemple de fichier XML à traiter, nommé autreCfg.xml :
<TRICROISE>
<variable1>
<nom>nivEtudes</nom>
<modalites>
<modalite valeur="0">Sans</modalite>
<modalite valeur="1">Primaire</modalite>
<modalite valeur="2">Secondaire</modalite>
<modalite valeur="3">Bac</modalite>
<modalite valeur="4">Sup</modalite>
</modalites>
</variable1>
<variable2>
<nom>clAGE</nom>
<modalites>
<modalite valeur="0">Enfant</modalite>
<modalite valeur="1">Adulte</modalite>
</modalites>
</variable2>
</TRICROISE>
Le détail des actions à effectuer sur le fichier XML de configuration est détaillé ci-dessous :
-
il faut charger le fichier XML de configuration en mémoire, ce qui se fait via la fonction xmlParse() qui est dans le package XML.
-
on récupère les informations recherchées (nom de variable, numéros et labels de modalités) à l'aide de la fonction getNodeSet() du package XML en utilisant de simples expressions XPATH ; le troisième paramètre indique ce qu'on veut faire de la donnée : pour les numéros de modalité, on veut convertir en nombre entier.
-
comme R renvoie des listes, on convertit en vecteur grâce à la fonction unlist() du package base
Voici un script plus complet avec prise en compte de XML dans le fichier co-occur3.r :
### (gH) -_- co-occur3.r
# on lit un ficher de données et un fichier XML de configuration
# on effectue le tri croisé complet des variables indiquées
# exemple d'utilisation :
# Rscript --encoding=latin1 --vanilla co-occur3.r elf2Data.txt elf2Data.xml
coOccurMarges <- function(dataFile,xmlFile) {
library("XML")
## 1. test du nombre de paramètres
if (nargs()<2) {
cat("\n")
cat("Nombre de paramètres incorrect.\n")
cat("Vous devez fournir un nom de fichier de données et un fichier XML de configuration.\n\n")
cat("Exemple : coOccurMarges(\"elf2Data.txt\",\"elf2Data.xml\")\n\n")
cat("Consulter http://forge.info.univ-angers.fr/~gh/Decra/co-occur.php pour le format des données.\n")
cat("\n")
stop(-1)
} # fin si sur argc
## 2. vérification de l'existence des fichiers
if (!file.exists(dataFile)) {
cat("\n")
cat("Fichier de données",dataFile,"non vu.\n")
stop(-2)
} # fin de si sur dataFile
if (!file.exists(xmlFile)) {
cat("\n")
cat("Fichier de configuration",xmlFile,"non vu.\n")
stop(-3)
} # fin de si sur dataFile
## 3. lecture du fichier XML de configuration
config <- xmlParse(xmlFile)
variable1 <- unlist(getNodeSet(doc=config,path="//variable1/nom/text()",fun=xmlValue))
modas1 <- getNodeSet(doc=config,path="//variable1/modalites/modalite",fun=xmlValue)
nums1 <- unlist(getNodeSet(doc=config,path="//variable1/modalites/modalite/@valeur",fun=as.integer))
variable2 <- unlist(getNodeSet(doc=config,path="//variable2/nom/text()",fun=xmlValue))
modas2 <- getNodeSet(doc=config,path="//variable2/modalites/modalite",fun=xmlValue)
nums2 <- unlist(getNodeSet(doc=config,path="//variable2/modalites/modalite/@valeur",fun=as.integer))
## 4. lecture des données et vérificaton de l'existence des colonnes
data <- read.table(dataFile,header=TRUE)
lesVars <- names(data)
if (!variable1 %in% lesVars) {
cat("Il n'y a pas de colonne nommée",variable1,"dans les données.\n") ;
stop(-4)
} # fin si sur variable1
if (!variable2 %in% lesVars) {
cat("Il n'y a pas de colonne nommée",variable2,"dans les données.\n") ;
stop(-5)
} # fin si sur variable1
## 5. construction du tri croisé amélioré
var1 <- data[,variable1]
var2 <- data[,variable2]
var1f <- factor(var1,levels=nums1,labels=modas1)
var2f <- factor(var2,levels=nums2,labels=modas2)
tc <- table(var1f,var2f)
pctsv1 <- paste(round(100*prop.table(table(var1f)),1),"%")
pctsv2 <- paste(round(100*prop.table(table(var2f)),1),"%")
tc <- rbind(tc,pctsv1)
tc <- cbind(tc,c(pctsv2," "))
row.names(tc)[1+length(modas1)] <- paste("pct",variable1,collapse=" ")
colnames(tc)[ 1+length(modas2)] <- paste("pct",variable2,collapse=" ")
print(tc,quote=FALSE)
} # fin de fonction coOccurMarges
# appel de la fonction
coOccurMarges("elf2Data.txt","elf2Data.xml")
On finit par faire un script complet avec test des paramètres, le code est dans le fichier co-occur4.r sachant que la fonction est définie dans le fichier co-occur.r :
### (gH) -_- co-occur4.r
# on se contente de tester le nombre de paramètres avant
# d'appeler la fonction lue dans le fichier co-occur.r
# exemple d'utilisation du script :
# Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt elf2Data.xml
## 1. test des arguments, aide éventuelle
args <- commandArgs(trailingOnly = TRUE)
argc <- length(args)
if (argc<2) {
cat("\n")
cat("Nombre de paramètres incorrect.\n")
cat("Vous devez fournir un nom de fichier de données et un fichier XML de configuration.\n\n")
cat("Exemple : Rscript --vanilla co-occur4.r elf2Data.txt elf2Data.xml\n\n")
cat("Consulter http://forge.info.univ-angers.fr/~gh/Decra/co-occur.php pour le format des données.\n")
cat("\n")
stop(-1)
} # fin si sur argc
## 2. vérification de l'existence des fichiers
ficDon <- args[1]
ficXml <- args[2]
## 3. lecture de la fonction
source("co-occur.r",encoding="latin1")
## 4. appel de la fonction
coOccurMarges(ficDon,ficXml)
Il est assez simple de tester les cinq cas d'erreur possibles prévus et un bon cas d'exécution :
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r
Nombre de paramètres incorrect.
Vous devez fournir un nom de fichier de données et un fichier XML de configuration.
Exemple : Rscript --vanilla co-occur4.r elf2Data.txt elf2Data.xml
Consulter http://forge.info.univ-angers.fr/~gh/Decra/co-occur.php pour le format des données.
Erreur : -1
Exécution arrêtée
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt2 elf2Data.xml2
Fichier de données elf2Data.txt2 non vu.
Error in coOccurMarges(ficDon, ficXml) : -2
Exécution arrêtée
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt elf2Data.xml2
Fichier de configuration elf2Data.xml2 non vu.
Error in coOccurMarges(ficDon, ficXml) : -3
Exécution arrêtée
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt elf3Data.xml
Il n'y a pas de colonne nommée SEX dans les données.
Error in coOccurMarges(ficDon, ficXml) : -4
Exécution arrêtée
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt elf4Data.xml
Il n'y a pas de colonne nommée EtMaritale dans les données.
Error in coOccurMarges(ficDon, ficXml) : -5
Exécution arrêtée
@ghchu4-27-p001nf~/public_html/Decra|(~gH) > Rscript --encoding=latin1 --vanilla co-occur4.r elf2Data.txt elf2Data.xml
Seul En couple pct EtMarital
Homme 1 1 42.9 %
Femme 2 3 57.1 %
pct SEXE 28.6 % 71.4 %
Pour les graphiques à produire, à savoir des histogrammes de fréquences (et non pas des histogrammes de classes), on pourra s'inspirer de notre introduction à R, séance 3, exercice 5
4. Programmation et développement en R
Comment fait-on du profilage, du déboggage en R ? Comment écrire des tests, des packages en R ? Faut-il passer au tidyverse ?
Pour répondre à ces questions, vous essaierez de réaliser la page d'exercices nommée progie9.
retour au plan de cours
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)
