1. Profilage et débogagge sous R
Peut-on facilement débugger du code R ? Et le «profiler, le benchmarker» ?
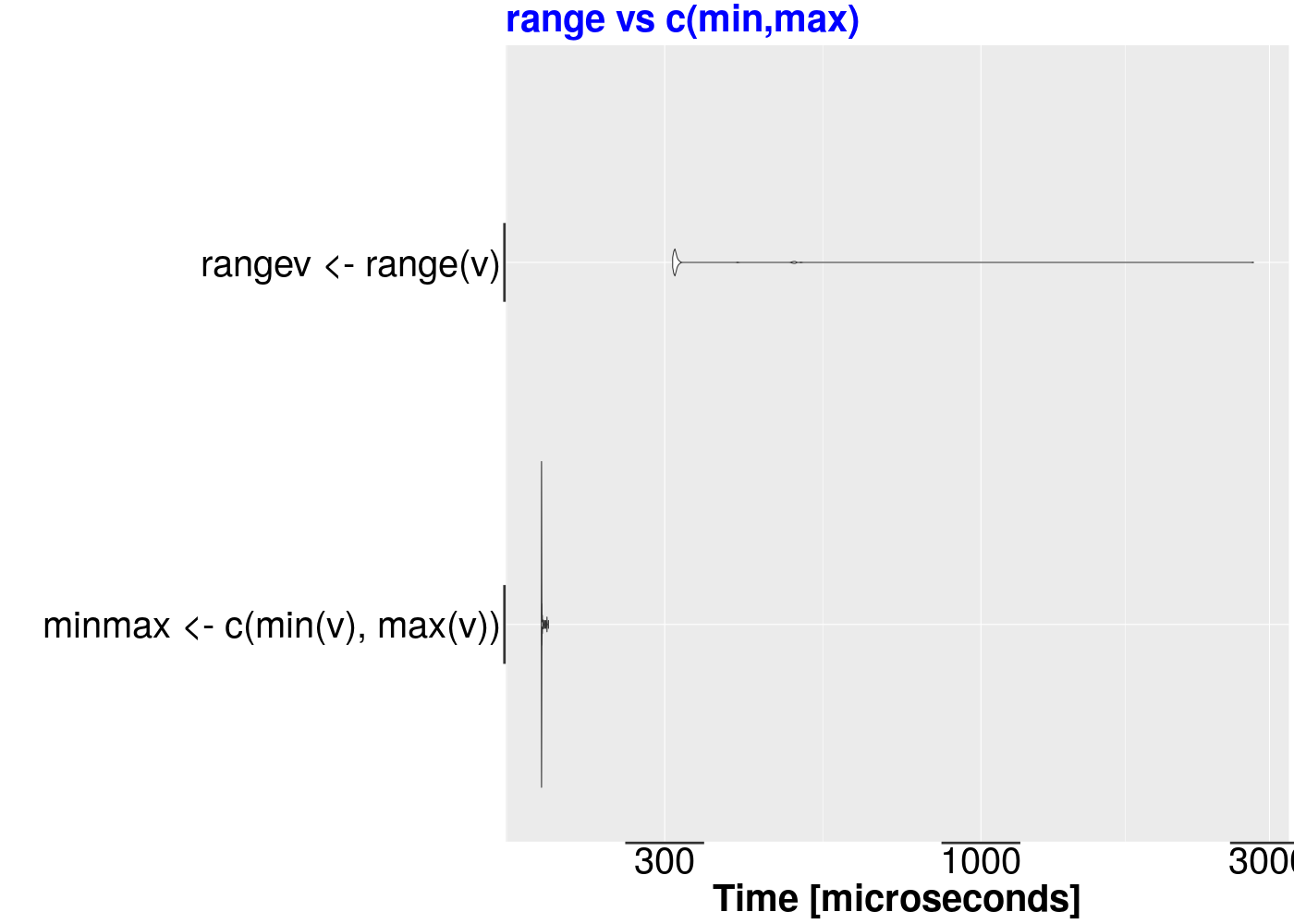
Par exemple, est-il plus rapide d'écrire c(min(v),max(v)) ou range(v) ?
Si vous ne savez pas répondre à cette question, c'est que vous n'avez pas fait la série précédente d'exercices ! Donc voici quelques mots pour vous rappeler ce qui a été dit à la séance 8.
R met à disposition de nombreux outils et packages, comme les fonctions debug(), browser(), recover(), et Rprof() ainsi que les packages pryr et lineprof.
On pourra consulter Rdebug.pdf (copie locale) sachant que le déboggage est intégré à Rstudio.
Vous pouvez aussi vous reporter à la séance 4 de nos cours de Programmation avancée avec R.
En ce qui concerne c(min(v),max(v)) ou range(v), le résultat est sans appel :
## comparaison de c(min(v),max(v)) et de range(v)
library(microbenchmark)
library(ggplot2)
options(width=100)
v <- runif(10**5)
resMB <- microbenchmark(
minmax <- c(min(v),max(v)) ,
rangev <- range(v)
) # fin de microbenchmark
g <- autoplot(resMB) +
ggtitle("range vs c(min,max)") +
theme(plot.title = element_text(size=40, face="bold",colour="blue")) +
theme(axis.title = element_text(size=40, face="bold")) +
theme(axis.ticks = element_line(size=40)) +
theme(axis.text = element_text(size=40,colour="black"))
plot(g)
Unit: microseconds
expr min lq mean median uq max neval cld
minmax <- c(min(v), max(v)) 187.465 187.5700 187.9684 187.6470 187.8935 191.302 100 a
rangev <- range(v) 481.804 483.7285 507.7412 484.6425 486.4650 2554.812 100 b
2. Packages pour tests en R
Comment fait-on des tests unitaires en R ? Quels packages sont prévus pour cela ? Application : écrire des tests pour la fonction ci-dessous avec les packages testthat et assertive.
################################################################################
#
# fonctions pour conversion pouce/cm
#
################################################################################
convPouceCm <- function(valeur,unite) {
if (missing(valeur) | missing(unite)) {
cat("\n")
cat(" syntaxe : convPouceCm(valeur,unite) avec unite = pouces ou cm\n")
cat(" exemples : convPouceCm(10,\"pouces\")\n")
cat(" convPouceCm(20,\"cm\")\n\n")
stop()
} ; # finsi
# pouce on multiplie, sinon on divise
facteurConv <- 2.54
uniteOk <- 0
if (unite=="pouces") {
autreu <- "cm" ;
uniteOk <- 1
} ;
if (unite=="cm") {
facteurConv <- 1/facteurConv ;
autreu <- "pouces" ;
uniteOk <- 1
} ; # fin si
if (uniteOk==0) {
cat(" unité incorrecte. il faut écrire exactement ",
" \"pouces\" ou \"cm\" comme unité.\n")
} else {
resultatConv <- valeur*facteurConv
cat(valeur," ",unite," = ", resultatConv," ",autreu,".\n",sep="") ;
} # finsi
return( c(resultatConv,autreu) )
} # fin de fonction convPouceCm
Expliquer pourquoi les fonctions de R ne fournissent pas d'aide si on les appelle sans paramètre, contrairement à nos fonctions :
> source("convpouces.r",encoding="latin1")
> convPouceCm()
syntaxe : convPouceCm(valeur,unite) avec unite = pouces ou cm
exemples : convPouceCm(10,"pouces")
convPouceCm(20,"cm")
Error in convPouceCm().
Plusieurs packages existent, dont assertr, ensurer, checkmate, tester, RUnit...
Parmi tous ces packages, deux semblent au-dessus du lot : assertive et testthat. On pourra consulter l'ouvrage testing R code pour s'en convaincre.

La fonction convPouces() comporte deux paramètres, valeur et unité qui doivent être respectivement un entier non négatif et une chaine de caractères. Voici les tests associés :
################################################################################
#
# fonction de conversion pouce/cm
#
# avec test "run-time" sur les paramètres
#
################################################################################
convPouceCm <- function(valeur,unite) {
if (missing(valeur) | missing(unite)) {
cat("\n")
cat(" syntaxe : convPouceCm(valeur,unite) avec unite = pouces ou cm\n")
cat(" exemples : convPouceCm(10,\"pouces\")\n")
cat(" convPouceCm(20,\"cm\")\n\n")
stop()
} ; # finsi
# test de "typage"
library(assertive)
assert_is_numeric(valeur)
assert_is_characterc(unite)
# pouce on multiplie, sinon on divise
facteurConv <- 2.54
uniteOk <- 0
if (unite=="pouces") {
autreu <- "cm" ;
uniteOk <- 1
} ;
if (unite=="cm") {
facteurConv <- 1/facteurConv ;
autreu <- "pouces" ;
uniteOk <- 1
} ; # fin si
if (uniteOk==0) {
cat(" unité incorrecte. il faut écrire exactement ",
" \"pouces\" ou \"cm\" comme unité.\n")
} else {
resultatConv <- valeur*facteurConv
cat(valeur," ",unite," = ", resultatConv," ",autreu,".\n",sep="") ;
} # finsi
return( c(resultatConv,autreu) )
} # fin de fonction convPouceCm
Cette fonction convPouces() est incorrecte car les variables resultatConv et autreu ne sont pas initialisées mais parfois quand même renvoyées. Voici les tests associés, nous vous laissons modifier la fonction en conséquence...
################################################################################
#
# fonction de conversion pouce/cm :
#
# tests "development-time" sur les résultats renvoyés
#
################################################################################
library("testthat")
# chargement de la fonction à tester
source("convpoucesOk.r")
# exécution des tests
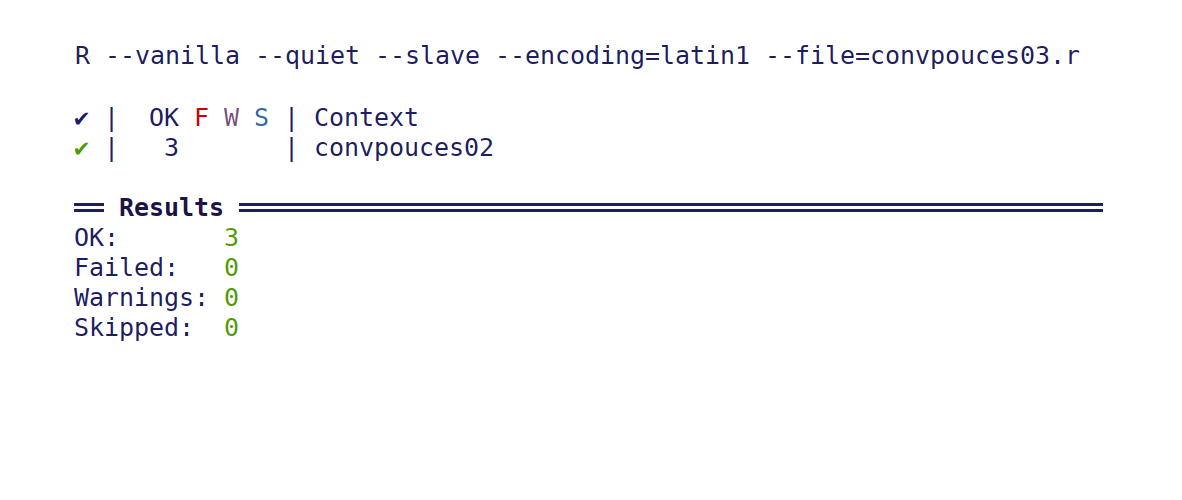
test_file("convpouces02.r")
## fonction de conversion pouce/cm : fichier convpouces02.r
# test 1 : conversion de pouces à cm
test_that(
"3 pouces font 7.62 cm",
{
expected <- c(7.62,"cm")
actual <- convPouceCm(3,"pouces")
expect_equal(actual,expected)
}
) # fin de test_that
# test 2 : conversion de cm à pouces
test_that(
"5 cm font 1.97 pouces",
{
expected <- c(1.97,"pouces")
actual <- convPouceCm(5,"cm")
expect_equal(actual,expected)
}
) # fin de test_that
# test 3 : sans paramètres, cela renvoie "error"
test_that(
"erreur si aucun paramètre" ,
{
expect_error( capture.output(convPouceCm()) )
}
) # fin de test_that
Les fonctions de R ne fournissent pas d'aide si on les appelle sans paramètre, contrairement à nos fonctions parce qu'elles sont toutes dans des packages. Donc leur aide s'obtient via help("laFonction"), ce qui n'est pas le cas de nos fonctions.
3. Comment définit-on des classes d'objets en R ?
Dans les langages de programmation classiques, on définit des classes d'objets. Peut-on le faire en R ?
Oui, bien sûr, il y a même deux langages à objets intégrés à R, historiquement : celui des classes S3 (ancien) et celui des classes S4 (nouveau). Depuis 2018 on a aussi des objets R6. Voir l'excellent document OO.html à ce sujet.
Un document de référence, disponible au cran est introduction to S4 (copie locale) qui est en anglais. Une version française existe aussi nommée petit manuel de S4 (copie locale).
Là encore, vous pouvez aussi vous reporter à la séance 4 de nos cours de Programmation avancée avec R pour voir un exemple concret de création d'une classe d'objets.
Vous trouverez ici la définition des classes et là leur utilisation, reproduit ci-dessous.
##########################################
#
# exemples de programmation objet S4 en R
#
##########################################
# définition des classes
source("demoObj1.r",encoding="latin1")
# utilisation de la classe VSqt
v7 <- VSqt(sonTitre="poids",saRef="dossier HER (gh)",sesValeurs=c(8,12,15),sonUnite="kg")
print( v7 )
getTitre( v7 )
getRef( v7 )
getValeurs( v7 )
getUnite( v7 )
setTitre( v7 ) <- "poidsG"
setRef( v7 ) <- "transf. en grammes de pois HER "
setValeurs( v7 ) <- getValeurs(v7)*1000
setUnite( v7 ) <- "g"
# traitement objet d'une qt
print( v7 )
getValeurs( v7 )
decrit( v7 )
# sur le même principe :
# not run : # traitement objet d'une ql
#
# maql <- Vsql(...
# print( maql )
# decrit( maql )
R --vanilla --quiet --slave --encoding=latin1 --file=demoObj2.r
Variable statistique : poids références : dossier HER (gh)
avec 3 valeurs uniques.
[1] "poids"
[1] "dossier HER (gh)"
[1] 8 12 15
[1] "kg"
Variable statistique : poidsG références : transf. en grammes de pois HER
avec 3 valeurs uniques.
[1] 8000 12000 15000
Min. 1st Qu. Median Mean 3rd Qu. Max.
8000 10000 12000 11667 13500 15000
4. Comment écrit-on un package R ?
Que contient exactement un package R ? Est-ce facile d'en écrire un, d'en déposer un au CRAN ?
Un package comporte des fonctions, des données, de l'aide et des exemples. Sur le principe, ce n'est pas difficile d'écrire un package. Mais c'est un peu fastidieux la première fois.
Pour éviter de vous reporter une nouvelle fois à la séance 4 de nos cours de Programmation avancée avec R qui montre l'écriture d'un tout petit package, nommé conv vous pouvez consulter la page de C. Genolini sur le sujet.
Comme indiqué à la question précédente, une fois le package chargé, les fonctions écrites dans le package ont le même statut que les autres fonctions de R, elles ont une complétion automatique, leur aide s'affiche dans le panneau d'aide etc.
On trouvera ci-dessous, issus des ressources de Rstudio le rappel de l'organisation d'un package, les commandes de construction et de test etc;
A titre d'exemple, sous Linux, créez un répertoire nommé conv et décompressez l'archive conv.zip dans ce répertoire. Dans le répertoire parent de conv, exécutez la commande R CMD check conv puis la commande R CMD build conv. Vous devriez disposer alors du fichier conv_1.0.tar.gz qu'on peut installer via Rstudio.
Le résultat de la commande check est rcmdcheck.txt et celui de la commande buil est rcmdbuild.txt.
Un autre exemple complet, avec du code R et C++ pour construire un package est ici qui aboutit au fichier hypotenuser.tar.gz que l'on peut installer dans Rstudio.
5. Est-il facile de reformater du code R ?
Certains écrivent du code R «n'importe comment». Y a-t-il des règles d'écriture de code R, des outils pour reformater le code R ?
Un outil très adapté est la fonction tidy_source() du package formatR.
Le plus important est de garder un même style d'écrire tout au long du code afin de faciliter la relecture.
6. Comment implémenter des "reproducible researches" en R ?
R et Rstudio ont un gros problème avec la notion d'environnement : certaines variables ont été crées "à la main", en interactif, et R les voit dans les fonctions... Comment produire et documenter du code "propre" et pouvoir reproduire des comportements fonctionnels en R ? Quels outils faut-il utiliser pour faire du DRY (qu'est-ce ?) en R ?
C'est très simple, il suffit d'utiliser knitr qui s'interface très bien avec Rstudio. Le code R y est exécuté à partir d'un environnement vierge à chaque fois, ce qui peut garantir la reproductibilité.
knitr est plus général et finalement plus simple à utiliser que Sweave. On consultera avec intérêt les ouvrages suivants :
A titre de vérification, on pourra reproduire intégralement le livre de C. Gandrud en suivant ce lien.
Pour DRY on peut consulter Baker (copie locale).
7. Bibliographie sur la programmation R et tidyverse
Il existe beaucoup de livres sur R vu sous l'angle des statistiques comme on peut le voir ici ici là. Mais pour la programmation, quels ouvrages trouve-t-on ?
Quels livres de H. Wickham (qui est-ce ?) sont en ligne ?
Expliquer ce que font les instructions suivantes écrites en R pour tidyverse.
x %>% f # curieux, n'est-ce pas ?
x %>% f(y) #
x %<>% f # il y a un symbole de plus qu'à la ligne 1
Réécrire en R/tidyverse le code suivant, solution d'un de nos exercices.
## comparaisons entre valeurs observées et théoriques, syntaxe classique
Vobs <- c(18,55,21,12,4)
Vthe <- c(6.875,27.5,41.25,27.5,6.875)
Ind <- 1:length(Vthe)
Dif <- Vthe - Vobs
Cntr <- Dif*Dif/Vthe
df <- data.frame(Ind,Vthe,Vobs,Dif,Cntr)
print(df)
# tri par contribution décroisssante
idx <- order(Cntr,decreasing=TRUE)
df <- df[idx,]
# ajout des pourcentages et du cumul
df[,"Pct"] <- 100*df$Cntr/sum(Cntr)
df$Cumul <- cumsum(df$Pct)
print(df)
Comment écrire sa thèse avec Rstudio ?
S'il y avait peu d'ouvrages en 2015, et aucun un peu long en français, sauf l'ouvrage de V. Goulet (copie locale), les choses ont beaucoup évolué, notamment en 2017. Nous vous conseillons toujours les trois livres qui suivent, du plus général (Gentleman) au plus spécialisé (Wickman) :
Aujourd'hui, en 2019, une recherche sur le web avec les termes r programming, r programming objects ou encore r programming functional fournir de nombreuses réponses, comme ci-dessous :
R programming

R programming objects
R programming functional

Hadley WICKHAM est l'un des auteurs de tidyverse. Voici ses livres en ligne :
Les nouveaux opérateurs ont pour but de rendre plus lisibles les opérations effectuées.
## compréhension de la synatxe %>% : équivalences avec le code R "usuel"
library(dplyr)
x <- 3.16758
# x %>% f correspond à f(x) et permet le chainage de fonctions
print(round(x))
x %>% round %>% print
# x %>% f(y) correspond à f(x,y)
print(round(x,2))
x %>% round(2) %>% print
# x %<>% met à jour x
library(magrittr)
x1 <- 3.16758
x1 <- round(x1,2)
print( x1 )
x1 <- 3.16758
print( x1 <- round(x1,2) )
x1 <- 3.16758
x1 %<>% round(2)
x1 %>% print
# il y a d'autres opérateurs % dans tidyverse
library(dplyr)
library(forcats)
library(ggplot2)
library(magrittr)
library(purrr)
library(readr)
library(stringr)
library(tibble)
library(tidyr)
opStd <- c("%*%","%/%","%%","%in%","%o%","%x%")
print( setdiff( apropos("%") , opStd ) ) # "%@%" "%+%" "%+replace%" "%||%" "%$%" "%T>%"
Cela change beaucoup la syntaxe des calculs :
## comparaisons entre valeurs observées et théoriques, syntaxe classique
Vobs <- c(18,55,21,12,4)
Vthe <- c(6.875,27.5,41.25,27.5,6.875)
Ind <- 1:length(Vthe)
Dif <- Vthe - Vobs
Cntr <- Dif*Dif/Vthe
df <- data.frame(Ind,Vthe,Vobs,Dif,Cntr)
print(df)
# tri par contribution décroisssante
idx <- order(Cntr,decreasing=TRUE)
df <- df[idx,]
# ajout des pourcentages et du cumul
df[,"Pct"] <- 100*df$Cntr/sum(Cntr)
df$Cumul <- cumsum(df$Pct)
print(df)
## comparaisons entre valeurs observées et théoriques en tidyverse
tibble(Obs = c(18,55,21,12,4),
The = c(6.875,27.5,41.25,27.5,6.875) ) %>%
mutate(Dif=The-Obs,Cntr=Dif*Dif/The) %>%
arrange(desc(Cntr)) %>%
mutate(Pct=100*Cntr/sum(Cntr),Cumul=cumsum(Pct)) %>%
print
Pour écrire sa thèse avec Rstudio, le mieux est d'utiliser le package bookdown :
Code-source php de cette page. Retour à la page principale du cours.
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)