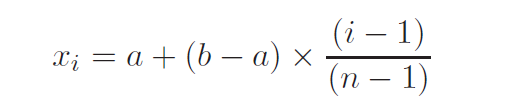1. Code lisible et debug
A-t-on besoin de la fonction debug() pour trouver pourquoi le code suivant ne fonctionne pas,
sachant qu'on cherche à renvoyer la chaine de caractères passée en paramètre
avec la lettre initiale en majuscule et toute le reste en minuscules ?
iniMaju <- function(chaine) {
return(paste(toupper(substr(chaine,0,1)),
tolower(substr(chaine,1,nchar(chaine))),sep=""))}
Sans doute que non. Ce code est mal écrit.
Vu qu'il y a une seule instruction dans le code proposé,
debug() ne peut rien pour nous. Démonstration :
# déboggage de la fonction iniMaju
> iniMaju <- function(chaine) {
+ return(paste(toupper(substr(chaine,0,1)),
+ tolower(substr(chaine,1,nchar(chaine))),sep=""))
+ }
> iniMaju("pomme") # incorrect !
[1] "Ppomme"
> debug(iniMaju)
> iniMaju("pomme")
debugging in: iniMaju("pomme")
debug à #1 :{
return(paste(toupper(substr(chaine, 0, 1)), tolower(substr(chaine,
1, nchar(chaine))), sep = ""))
}
Browse[2]>n
debug à #2 :return(paste(toupper(substr(chaine, 0, 1)), tolower(substr(chaine,
1, nchar(chaine))), sep = ""))
Browse[2]>
exiting from: iniMaju("pomme")
[1] "Ppomme"
> undebug(iniMaju)
# fin de déboggage de la fonction iniMaju
Pour que le déboggage soit efficace,
il vaudrait mieux décomposer chacune
des actions, avec quelque chose comme
iniMaju <- function(chaine) {
longueurChaine <- nchar(chaine)
initiale <- substr(chaine,0,1)
initialeMaju <- toupper( initiale )
resteDeChaine <- substr(chaine,1,longueurChaine)
resteEnMinu <- tolower(resteDeChaine)
nvlChaine <- paste(initialeMaju,resteEnMinu,sep="")
return( nvlChaine )
} # fin de fonction iniMaju
pour se rendre compte que les indices sont faux.
Avec le nouveau code proposé, on peut tester chaque appel de fonction séparément
et en déduire que la variable nommée initiale ne contient pas la bonne valeur, en
utilisant cette fois debug(). On peut alors corriger les indices pour initiale
et pour resteDeChaine qui étaient faux (mais qui ne généraient pas d'erreur) :
iniMaju <- function(chaine) {
longueurChaine <- nchar(chaine)
initiale <- substr(chaine,1,1) # OK !
initialeMaju <- toupper( initiale )
resteDeChaine <- substr(chaine,2,longueurChaine) # OK !!
resteEnMinu <- tolower(resteDeChaine)
nvlChaine <- paste(initialeMaju,resteEnMinu,sep="")
return( nvlChaine ) # renvoie "Pomme" si "pomme" en entrée
} # fin de fonction iniMaju
2. Optimisation
On dispose d'une fonction mathématique f et on voudrait calculer sa valeur
en n points répartis uniformément sur l'intervalle [a,b] à l'aide d'une boucle.
On propose la formule suivante pour
calculer la position de chacun des points xi, pour i de 1 à n, sachant que x1=a et xn=b :
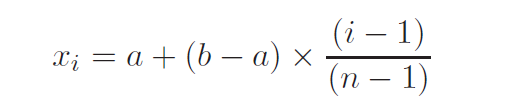
On veut par exemple tester ce calcul avec f(x)=x+1 sur l'intervalle [0,1]
avec n=100 points.
Quel serait le code optimisé pour cette boucle ?
Peut-on effectuer le calcul sans boucle ?
La formule fournie pour calculer chacun des xi est tout à fait correcte mathématiquement.
Sa programmation naïve ne pose aucun problème :
# calcul de chacun des points xi selon la formule
# xi = a + (b-a)*(i-1)/(n-1) pour i de 1 à n
# boucle naive
lesXi1 <- function(a,b,n) {
xi <- c()
for (i in (1:n) ) {
xc <- a + (b-a)*(i-1)/(n-1)
xi <- c(xi,xc)
} # fin pour i
return( xi )
} # fin de fonction lesXi1
Une première optimisation de la boucle consiste à sortir les "constantes" de la boucle
et à initialiser le vecteur de sortie, soit le code :
# calcul de chacun des points xi selon la formule
# xi = a + (b-a)*(i-1)/(n-1) pour i de 1 à n
# boucle un peu optimisée
lesXi2 <- function(a,b,n) {
xi <- rep(NA,n)
h <- (b-a)/(n-1)
for (i in (1:n) ) {
xc <- a + h*(i-1)
xi[ i ] <- xc
} # fin pour i
return( xi )
} # fin de fonction lesXi2
Une deuxième optimisation consiste à remarquer que les xi sont en progression arithmétique,
donc il suffit de rajouter le pas h de la progression pour passer d'un point à l'autre :
# calcul de chacun des points xi selon la formule
# xi = a + (b-a)*(i-1)/(n-1) pour i de 1 à n
# boucle très optimisée
lesXi3 <- function(a,b,n) {
xi <- rep(NA,n)
h <- (b-a)/(n-1)
xc <- a - h
for (i in (1:n) ) {
xc <- xc + h
xi[ i ] <- xc
} # fin pour i
return( xi )
} # fin de fonction lesXi3
Bien sûr le calcul vectoriel permet de se passer de boucle :
# calcul de chacun des points xi selon la formule
# xi = a + (b-a)*(i-1)/(n-1) pour i de 1 à n
# calcul vectoriel (sans boucle)
lesXi4 <- function(a,b,n) {
h <- (b-a)/(n-1)
xi <- a + h*((1:n)-1)
return( xi )
} # fin de fonction lesXi4
Et enfin, on peut se demander si la fonction seq() ne pourrait pas suffire :
# calcul de chacun des points xi selon la formule
# xi = a + (b-a)*(i-1)/(n-1) pour i de 1 à n
# calcul vectoriel (sans boucle) via seq
lesXi5 <- function(a,b,n) {
return( seq(from=a,to=b,length.out=n) )
} # fin de fonction lesXi5
Le jeu en vaut-il la chandelle ? Vérifions-le avec la fonction microbenchmark() :
> library("microbenchmark")
> a <- 0
> b <- 1
> n <- 10**2
> microbenchmark(
xi_fn1 = lesXi1(a,b,n) ,
xi_fn2 = lesXi2(a,b,n) ,
xi_fn3 = lesXi3(a,b,n) ,
xi_fn4 = lesXi4(a,b,n) ,
xi_fn5 = lesXi5(a,b,n)
) # fin de microbenchmark
Unit: microseconds
expr min lq mean median uq max neval cld
xi_fn1 150.420 157.1820 162.47964 160.5025 167.0080 211.472 100 e
xi_fn2 125.019 135.6610 140.03099 140.1665 143.9690 167.032 100 d
xi_fn3 101.009 107.5430 111.29040 110.9280 114.9815 136.324 100 c
xi_fn4 2.978 3.7280 4.20139 3.9315 4.1805 15.079 100 a
xi_fn5 9.348 11.9055 13.28538 12.6395 13.6635 38.468 100 b
Le résultat est sans appel : la solution 4, basée sur 1:n est de loin la plus performante,
puisqu'elle est près de 40 fois plus rapide que la solution naïve, en moyenne comme en médiane.
La solution 5 qui utilise la fonction seq()
n'est sans doute pas adaptée ici, même si elle est assez rapide.
Peut-on encore optimiser ? Oui, sans doute, mais à condition de tester des cas particuliers.
Par exemple, on peut remarquer que si a est égal à 0 alors il n'y pas d'addition à faire.
De même, si f est une fonction constante, on n'a pas besoin de calculer les xi
Au passage, vous pourriez vous demander pourquoi nous n'avons pas mis le calcul des f(xi) dans
les fonctions. La réponse est simple : ce qu'on doit comparer est la génération des xi.
Après avoir vérifié que les fonctions renvoient les mêmes
vecteurs,
les calculs des f(xi) prendront autant de temps. Les inclure dans les fonctions ajouterait
les durées de calcul des f(xi) qui peuvent être beaucoup plus importantes que celles des générations des xi
et masquer ainsi les durées de génération.
Vous vous rappelez sans doute comment tester que deux vecteurs sont égaux, mais voici quand même
ce qu'il faut écrire et ce qu'il ne faut pas écrire pour le tester :
# vérification que les xi générés sont bien les mêmes sur un exemple
# avec un pot-pourri de méthodes
a <- 0
b <- 1
n <- 10**2
eps <- 10**(-15)
# ce test échoue car certaines différences sont l'ordre de
# dix puissance moins seize
print( sum(lesXi1(a,b,n)==lesXi2(a,b,n))==n )
# vérification (cela affiche 6 8 11 12 15 18)
print( head( which( lesXi1(a,b,n) != lesXi2(a,b,n) ) ))
# pour le sixième élément, la différence vaut -6.938894e-18 :
print( lesXi1(a,b,n)[6] - lesXi2(a,b,n)[6] )
# un test plus correct
print( sum(abs(lesXi1(a,b,n)-lesXi2(a,b,n))<eps)==n )
# une solution avec une fonction de base de R
print( all.equal( lesXi2(a,b,n), lesXi3(a,b,n) ) )
# utilisation de la fonction compare du package testthat
print( compare( lesXi3(a,b,n), lesXi4(a,b,n) ) )
> print( sum(lesXi1(a,b,n)==lesXi2(a,b,n))==n )
[1] FALSE
> print( head( which( lesXi1(a,b,n) != lesXi2(a,b,n) ) ))
[1] 6 8 11 12 15 18
> print( lesXi1(a,b,n)[6] - lesXi2(a,b,n)[6] )
[1] -6.938894e-18
> print( sum(abs(lesXi1(a,b,n)-lesXi2(a,b,n))<eps)==n )
[1] TRUE
> print( all.equal( lesXi2(a,b,n), lesXi3(a,b,n) ) )
[1] TRUE
> print( compare( lesXi3(a,b,n), lesXi4(a,b,n) ) )
Equal
3. Vitesses d'exécution
Peut-on vraiment affirmer que le code mot %in% vec est vraiment plus
rapide que le code grep(mot,vec) ?
Et que any(v>0) est vraiment plus
rapide que le code sum(v>0)>0 ?
Faut-il "sortir" les calculs dans les écritures des bornes des boucles ? Ainsi,
faut-il écrire les boucles POUR et TANT QUE sous la forme
lng <- length(v)
pdv <- 1:lng
for (ind in pdc) {
traitement()
} # fin pour ind
ind <- 1
while (ind<=lng) {
traitement()
ind <- ind + 1
} # fin tant que
plutôt que de les écrire
for (ind in 1:length(v)) {
traitement()
} # fin pour ind
ind <- 1
while (ind<=length(v)) {
traitement()
ind <- ind + 1
} # fin tant que
Peut-on encore optimiser le code suivant qui calcule le
maximum d'un vecteur, son nombre d'occurrences et toutes ses positions ?
maxV <- max(V) # calcul une seule fois du maximum
list( maximum=maxV , # le maximum
nbOcc=sum(V==maxV) , # son nombre d'occurences
lstOcc=which(V==maxV) # toutes ses positions
) # fin de liste
Que faire si finalement R est lent, non dans les fonctions de base mais dans une fonction utilisateur ?
Il est certainement difficile de démontrer mathématiquement qu'une expression
s'exécute vraiment plus rapidement qu'une autre, sauf à être capable de calculer
la complexité des algorithmes sous-jacents. Par contre, avec suffisamment
d'exemples, on doit pouvoir se convaincre de la rapidité de tel ou tel code.
En ce qui concerne la détection de la présence d'un mot dans une liste, il semblerait bien que
l'expression mot %in% vec est vraiment plus
rapide que le code grep(mot,vec), même si ajouter le paramètre fixed=TRUE
diminue très sensiblement la durée d'exécution. Pour mémoire, mettre fixed=TRUE
comme paramètre dans l'appel de grep prévient R qu'on cherche une chaine
de caractères et non pas une expression régulière, ce qui est plus simple
à réaliser et donc plus rapide à exécuter :
> # vitesse d'exécution pour tester la présence
> # d'un mot dans un vecteur de mots
> library(microbenchmark)
> mot <- "le"
> txt <- readLines("cand.txt",encoding="latin1")
> vec <- unlist(strsplit(txt, " "))
> microbenchmark(
mot %in% vec ,
grep(mot,vec) ,
grep(mot,vec,fixed=TRUE)
) # fin de microbenchmark
Unit: microseconds
expr min lq mean median uq max neval cld
mot %in% vec 5.302 6.0495 6.60208 6.3320 6.690 15.872 100 a
grep(mot, vec) 137.979 139.4325 143.36609 140.2435 142.246 280.792 100 c
grep(mot, vec, fixed = TRUE) 24.304 24.9310 25.56462 25.3045 25.708 35.929 100 b
En ce qui concerne la deuxième question sur la présence d'au moins un élément positif dans un vecteur,
le code suivant qui utilise trois expressions vectorielles donne des résultats d'exécution différents suivant la position
des valeurs positives dans le vecteur :
# vitesse d'exécution pour tester la présence
# d'un élément positif dans V
library(microbenchmark)
microbenchmark(
sum(v>0)>0 ,
any(v>0) ,
length(which(v>0))>0
) # fin de microbenchmark
Par exemple, avec deux cas extrêmes où la seule valeur positive est située en début ou en fin d'un vecteur de 100 000 éléments,
ce n'est pas la même expression vectorielle qui est plus efficace :
n <- 10**5
v1 <- c(1,rep(-1,n-1))
v2 <- c(rep(-1,n-1),1)
Unit: microseconds
expr min lq mean median uq max neval cld
sum(v1 > 0) > 0 756.084 756.803 848.2423 852.2135 854.0935 4420.934 100 b
any(v1 > 0) 683.523 684.141 752.7270 779.2075 781.3320 1042.614 100 a
length(which(v1 > 0)) > 0 739.321 742.020 816.9807 838.5130 840.3690 1068.411 100 ab
Unit: microseconds
expr min lq mean median uq max neval cld
sum(v2 > 0) > 0 756.141 756.589 778.6586 756.9545 759.908 1010.385 100 a
any(v2 > 0) 790.961 791.349 853.7908 791.7970 797.756 2882.868 100 b
length(which(v2 > 0)) > 0 739.393 739.866 755.5119 740.2995 743.610 971.975 100 a
On pourrait penser écrire notre propre boucle tant que de détection du premier élément positif dans le vecteur,
comme dans le code suivant :
# test par boucle TANT QUE de la présence
# d'un élément positif dans un vecteur
positif <- function(v) {
lng <- length(v)
ind <- 1
while (ind<lng) {
if (v[ind]>0) { return (TRUE) }
ind <- ind + 1
} # fin tant que
return(FALSE)
} # fin fonction positif
Pour nos cas extrêmes, les résultats sont extrêmes aussi :
n <- 10**5
v1 <- c(1,rep(-1,n-1))
Unit: microseconds
expr min lq mean median uq max neval cld
sum(v1 > 0) > 0 755.597 756.3160 758.32397 756.7260 757.4355 805.808 100 c
any(v1 > 0) 683.288 683.7215 687.50737 683.9315 684.3150 912.142 100 b
length(which(v1 > 0)) > 0 739.108 739.7625 783.88399 741.3445 757.1050 2569.474 100 c
positif(v1) 1.341 2.0280 2.65313 2.3115 2.6595 13.815 100 a
v2 <- c(rep(-1,n-1),1)
expr min lq mean median uq max neval cld
sum(v2 > 0) > 0 756.149 756.9140 760.2720 758.0545 763.079 794.054 100 a
any(v2 > 0) 790.926 791.4035 794.3892 792.4030 796.607 810.160 100 a
length(which(v2 > 0)) > 0 739.431 740.5620 746.0218 745.0460 748.760 774.352 100 a
positif(v2) 61894.112 62900.3845 64621.5361 63258.6845 64135.034 116874.661 100 b
Il faut donc en retenir qu'optimiser du code suppose parfois qu'on connait ou qu'on est capable de "deviner" la distribution
des données, ce qui n'est pas une mince affaire.
La réponse à la troisième question concernant les conditions de sortie de boucle est un peu plus simple : il faut mettre les
calculs constants à l'extérieur des boucles. Il faut donc prendre l'habitude d'écrire
lng <- length(v)
pdv <- 1:lng
boucle1 <- function() {
for (ind in pdv) {
traitement()
} # fin pour ind
} # fin de fonction boucle1
boucle3 <- function() {
ind <- 1
while (ind<=lng) {
traitement()
ind <- ind + 1
} # fin tant que
} # fin de fonction boucle3
plutôt que
boucle2 <- function() {
for (ind in 1:length(v)) {
traitement()
} # fin pour ind
} # fin de fonction boucle2
boucle4 <- function() {
ind <- 1
while (ind<=length(v)) {
traitement()
ind <- ind + 1
} # fin tant que
} # fin de fonction boucle4
car même si le gain n'est pas très important, il est non négligeable, même sur cet exemple simple :
# vitesse d'exécution pour l'écriture des boucles
library(microbenchmark)
traitement <- function(v) {
# fonction vide volontairement
} # fin fonction positif
v <- c(1:10**5)
lng <- length(v)
pdv <- 1:lng
microbenchmark(
boucle1() ,
boucle2() ,
boucle3() ,
boucle4()
) # fin de microbenchmark
Unit: milliseconds
expr min lq mean median uq max neval cld
boucle1() 25.51333 26.51870 27.80427 27.20823 27.99950 80.73637 100 a
boucle2() 25.34205 26.74191 27.32803 27.53890 27.95463 29.82511 100 a
boucle3() 56.79907 58.77331 60.68954 60.03622 60.50969 115.15793 100 b
boucle4() 68.21926 68.84720 70.89746 70.30240 71.12628 124.15789 100 c
Pour finir cet exercice, il est facile de voir qu'on peut encore optimiser le code proposé
puisque l'expression vectorielle V==maxV est utilisée deux fois et donc calculée deux fois.
Si on ne calcule qu'une seule fois cette expression,
on dispose d'un code qui peut être jusqu'à deux fois plus rapide :
## test d'optimisation pour le calcul du maximum
max1 <- function(V) { # fonction non optimisée
maxV <- max(V) # calcul une seule fois du maximum
list( maximum=maxV , # le maximum
nbOcc=sum(V==maxV) , # son nombre d'occurences
lstOcc=which(V==maxV) # toutes ses positions
) # fin de liste
} # fin de fonction max1
max2 <- function(V) { # fonction optimisée
maxV <- max(V) # calcul une seule fois du maximum
eglV <- V==maxV # calcul une seule fois des égalités
list( maximum=maxV , # le maximum
nbOcc=sum(eglV) , # son nombre d'occurences
lstOcc=which(eglV) # toutes ses positions
) # fin de liste
} # fin de fonction max2
microbenchmark(
max1(V) , max2(V)
) # fin de microbenchmark
# pour V <- round(runif(n = 10**6,min=0,max=100))
Unit: milliseconds
expr min lq mean median uq max neval cld
max1(V) 19.36161 19.42726 21.14802 21.13138 21.22579 72.75764 100 b
max2(V) 12.15664 12.17525 13.91334 12.22388 13.91706 65.51350 100 a
# pour V <- rbinom(n=10**6,size=50,p=0.1)
Unit: milliseconds
expr min lq mean median uq max neval cld
max1(V) 6.262988 6.290247 8.850963 8.016538 8.071895 60.308370 100 b
max2(V) 4.302437 4.327025 4.949732 4.350346 6.077635 6.296828 100 a
Si au final, après une étude de complexité de l'algorithme, R est lent dans une de vos fonctions, une solution possible consiste à interfacer un programme C++ depuis R ou, dans l'autre sens, appeler R depuis un programme C++. Les packages Rcpp et RInside sont prévus pour cela. On pourra consulter le site rcpp.org et les livres suivants :

Il ne faut pas oublier non plus que R dispose, dans ses fameuses Task Views, d'une entrée HPC et d'une entrée ML.
4. Positions du maximum et gestion de la mémoire
On veut trouver les 5 premières positions du maximum dans un très grand vecteur d'entiers.
Est-ce un problème ?
Et si maintenant on veut stocker toutes les positions du maximum, comment faire
sachant qu'il peut y avoir potentiellement très peu de positions ou au contraire
un très grand nombre d'occurrences de ce maximum ?
Le mieux est sans doute de commencer par une solution naïve et de tester sa
vitesse d'exécution, soit le code :
## les 5 premières positions du maximum
## version naive mais vectorielle
posMax1 <- function( leVecteur ) {
leMax <- max( leVecteur )
posMax <- which( leVecteur == leMax )
print( posMax[1:5] ) # beaucoup pus rapide que head(posMax,n=5)
} # fin de fonction posMax1
Si c'est effectivement trop lent, on peut envisager un parcours du vecteur
à l'aide d'une boucle tant que et sortir dès qu'on a trouvé les 5 premières positions du maximum :
## les 5 premières positions du maximum
## version boucle TANT QUE à partir du début
posMax2 <- function( leVecteur ) {
lngv <- length( leVecteur )
leMax <- max( leVecteur )
posMax <- rep(NA,5)
nbMaxVu <- 0
indc <- 1 # indice courant
while ((nbMaxVu<5) & (indc<=lngv)) {
eltc <- leVecteur[ indc ]
if (leVecteur[indc]==leMax) {
nbMaxVu <- nbMaxVu + 1
posMax[ nbMaxVu ] <- indc
# on sort si on a vu 5 fois le maximum
if (nbMaxVu==5) {
return(posMax)
} # fin si
} # fin si maximum vu
indc <- indc + 1
} # fintant que
return(posMax)
} # fin de fonction posMax2
Il est sans doute possible d'encore optimiser à l'aide de méta-connaissances.
Par exemple si on sait que le vecteur dans lequel dans on recherche le
maximum est issu d'une méthode qui converge vers des grandes valeurs, on pourrait
commencer la recherche par la fin en initialisant l'indice courant de recherche indc avec
la longueur lngv du vecteur
au lieu de 1
et remonter vers le début du vecteur, donc en mettant (indc>=1)
comme deuxième test dans la condition de bouclage du TANT QUE
et en décrémentant indc :
## les 5 premières positions du maximum
## version boucle tant que à partir de la fin
## (extrait)
posMax3 <- function( leVecteur ) {
lngv <- length( leVecteur )
leMax <- max( leVecteur )
posMax <- rep(NA,5)
nbMaxVu <- 0
indc <- lngv # indice courant
while ((nbMaxVu<5) & (indc>=1)) {
[...]
indc <- indc - 1
} # fintant que
[...]
Si au contraire on sait que la méthode est perturbée ou relativement stationnaire
au début et en fin, on pourrait commencer à partir du milieu du vecteur et parcourir
alternativement à gauche et à droite de cette position de départ, ou commencer
avec une position aléatoire "pas trop proche" des bords...
Une deuxième difficulté consiste à choisir des exemples pour tester la vitesse d'exécution des trois solutions
présentées. Ainsi, vaut-il mieux générer
leVecteur à l'aide de l'expression round(runif(n=10**w,min=0,max=100))
ou avec rbinom(n=10**w,size=50,prob=p) ? Et avec quelle valeur de w et p ?
Serait-ce mieux d'utiliser une autre distribution, comment et pourquoi ?
En ce qui concerne toutes les positions du maximum, le problème est un peu plus compliqué
puisqu'on ne connait pas la taille du résultat. On a déjà vu que concaténer une valeur à
un vecteur est très lent, en d'autres termes, il faut éviter d'écrire
posMax <- c(posMax,pcdm) # pcdm : Position Courante Du Maximum
au profit d'une écriture en mémoire dans un vecteur de taille définie à l'avance soit
posMax[ ind ] <- pcdm # pcdm : Position Courante Du Maximum
Comme on ne peut pas prédire à l'avance la taille du résultat, si on doit absolument passer par une boucle POUR,
l'optimisation consiste à ne PAS changer la taille du vecteur résultat TROP SOUVENT.
Donc au lieu de changer cette cette taille à chaque passage dans la boucle lorsqu'on trouve une nouvelle fois le maximum,
il faut astuctieusement changer cette taille de temps en temps, quand la taille courante ne suffit plus.
Si (par sondage, expertise, test...) on connait en gros l'augmentation souhaitée de la taille,
on peut en profiter pour ne pas trop augmenter à chaque fois la taille ;
sinon on peut choisir de la doubler. Voici ce que cela donnerait sur exemple où nous avons décidé d'utiliser une
taille de n1 <- 5 éléments au départ et de rajouter n2 <- 7 élements à chaque fois qu'il y en a besoin.
Ces valeurs sont ici arbitraires et ne servent qu'à la démonstration. De "vraies" valeurs seraient plutot de l'ordre de
d'une grande puissance de 10 puisqu'il faudrait déjà que l'expression vectorielle which(V==maxV) ne soit pas
efficace...
Une remarque : si on augmente de temps en temps la taille du vecteur de résultats avec de NA, il ne faut pas oublier
de retirer à la fin ceux qui n'ont pas été remplacés par une vraie position du maximum. C'est bien sûr ce que nou avons
fait dans le code suivant :
posMax <- function(vect) {
# toutes les positions du maximum :
# on alloue au départ 5 positions de minimum (variable alloc)
# et on rajoute 7 NA quand on a rempli ces positions
n1 <- 5
n2 <- 7
if (missing(vect)) {
cat("\nSyntax: posMinV1bis(vect).\n")
} else {
alloc <- n1
pdm <- rep(NA,alloc)
nbe <- length(vect)
vm <- min(vect) # Valeur du Minimum
nb <- 0 # nombre de positions vues du minimum
for (ide in 1:nbe) {
if (vect[ide]==vm) {
nb <- nb + 1 # et une position de plus
# si cela risque de déborder, on alloue plus de place
if (nb>length(pdm)) {
length(pdm) <- length(pdm) + n2 # mette ici 2*length(alloc) pour doubler la place
} # fin si
pdm[nb] <- ide
} # fin si
# pour debug :
cat(" ide ",sprintf("%3d",ide)," x = ",vect[ide]," donc pdm " , pdm,"\n")
} # fin pour ide
# pour ne pas renvoyer de NA, il faut retier ceux qui restent éventuellement
pdm<- na.omit(pdm)
# soit
pdm <- pdm[ 1:nb ]
return(pdm)
} # fin si
} # fin de fonction posMax
Voici ce qu'on obtient sur un exemple :
Vecteur : 1 0 0 0 1 0 0 1 1 1 0 0 0 1...
ide 1 x = 1 donc pdm NA NA NA NA NA
ide 2 x = 0 donc pdm 2 NA NA NA NA
ide 3 x = 0 donc pdm 2 3 NA NA NA
ide 4 x = 0 donc pdm 2 3 4 NA NA
ide 5 x = 1 donc pdm 2 3 4 NA NA
ide 6 x = 0 donc pdm 2 3 4 6 NA
ide 7 x = 0 donc pdm 2 3 4 6 7
ide 8 x = 1 donc pdm 2 3 4 6 7
ide 9 x = 1 donc pdm 2 3 4 6 7
ide 10 x = 1 donc pdm 2 3 4 6 7
ide 11 x = 0 donc pdm 2 3 4 6 7 11 NA NA NA NA NA NA
ide 12 x = 0 donc pdm 2 3 4 6 7 11 12 NA NA NA NA NA
[...]
Positions : 2 3 4 6 7 11 12...
5. Tris et "big data"
On dispose d'un fichier issu de la bioinformatique dont la taille est
environ 4 Go.
Comment faire pour trier les lignes de ce fichier sachant que
la mémoire de l'ordinateur est seulement de 2 Go
et qu'on veut les cinq premières lignes triées ?
En fait, on ne s'intéresse qu'aux cinq lignes les plus longues de ce fichier.
Faut-il le trier par ordre de taille de ligne et n'en garder que les cinq premières
lignes ou peut-on écrire un algorithme plus efficace ?
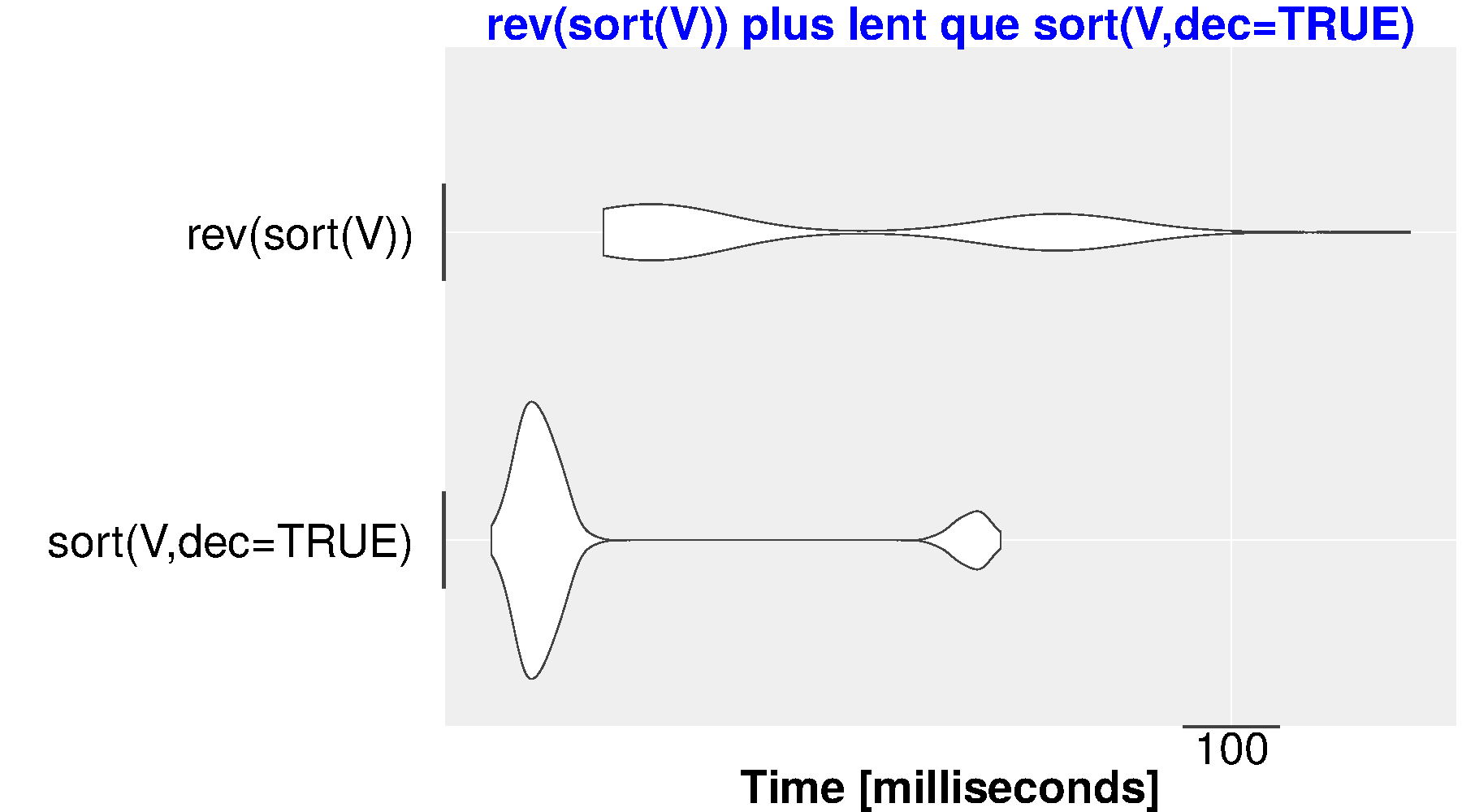
Peut-on vraiment affirmer que le code sort(V,dec=TRUE) est vraiment plus
rapide que le code rev(sort(V)) pour trier par ordre décroissant ?
Ah, la bioinformatique et ses gros fichiers !
On commence ici à entrer dans des problèmes plus délicats et c'est pourquoi ce sera
le dernier exercice de cette section.
Cet exercice peut être classé dans la catégorie exercices de la mort qui tue car
il est à la fois chronophage et "prise de tête". Voici par exemple ce qu'il serait si simple
d'exécuter si on avait assez de mémoire :
lignes <- readLines(fichier)
lignesTri <- sort(lignes)
cat(length(lignes),"lues dans",fichier,"\n")
cat("voici les 5 premières lignes (après tri)\n")
print(lignesTri[1:5])
Dans ce cas,
pour notre fichier de test, dont la taille est d'environ 3,9 giga-octets, la lecture
des 63,4 millions de lignes prend environ 5 minutes et le tri 20 minutes -- l'ordinateur
utilisé ici
est relativement assez puissant et, surtout, il a 32 giga-octets de mémoire. Il est clair
qu'avec une machine plus limitée en mémoire, cela va prendre encore plus de temps.
Nous avons essayé d'exécuter le même code sur une machine Windows avec effectivement
2 giga-octets de mémoire. En mode session traditionnelle, R ne se "plaint" pas
mais n'affiche rien. Le prompt réapparait au bout de quelques minutes mais plus rien ne fonctionne :
à chaque nouvelle instruction exécutée, R répond de façon sybilline :
Error: C stack usage 15938580 is too close to the limit.
Avec Rstudio, ce n'est guère mieux. Au bout de quelques minutes aussi apparait le message
cette fois un peu plus explicite :
Error: cannot allocate vector of size 125.0 Mb
Pour mettre au point le script qui effectue la lecture et le tri, il ne faut surtout pas
utiliser le gros fichier car mettre 30 minutes ou plus à chaque essai se révèlera catastrophique
en temps. Comme souvent dans ce genre de programmation, il faut écrire et affiner le script sur
un petit jeu d'essai avant de tester sans doute une et une seule fois l'exécution sur le gros fichier.
Puisqu'on ne peut pas charger le fichier des données en mémoire, il faut le
scinder en plusieurs petits fichiers puis faire un tri-fusion de ces petits fichiers.
Par exemple, 3,9 Go divisés par trois donne 1,3 Go qui est inférieur à la taille de
la mémoire de la machine.
Un tri-fusion consiste à trier chacun de deux fichiers obtenus, disons A et B
puis à fusionner les fichiers triés.
Pour cela, on lit au fur et à mesure dans chacun des deux fichiers triés, une ligne à
la fois, ce qui ne prend presque rien en mémoire, pour écrire dans le fichier
résultat C. Voici un exemple élémentaire d'exécution avec 3 lignes dans A
et deux lignes dans B.
Suppons que le contenu des fichiers soit :
# Contenu du fichier A | # Contenu du fichier B
|
abc # ligne 1 de A | ijk # ligne 1 de B
def # ligne 2 de A | zzz # ligne 2 de B
tmp # ligne 3 de A |
Alors l'exécution du tri-fusion des fichiers A et B se fait en 5 étapes :
étape 1 :
----------
on lit dans A abc # ligne 1 de A
on lit dans B ijk # ligne 1 de B
==> on écrit dans C abc # ligne 1 de A
étape 2 :
----------
on lit dans A def # ligne 2 de A
[pour B, on a ijk # ligne 1 de B]
==> on écrit dans C def # ligne 2 de A
étape 3 :
----------
on lit dans A tmp # ligne 3 de A
[pour B, on a ijk # ligne 1 de B]
==> on écrit dans C ijk # ligne 1 de B
étape 4 :
----------
[pour A, on a tmp # ligne 3 de A]
on lit dans B zzz # ligne 2 de B
==> on écrit dans C tmp # ligne 3 de A
étape 5 :
----------
[plus rien à lire dans A]
[pour B, on a zzz # ligne 2 de B
==> on écrit dans C zzz # ligne 2 de B
Après réflexion, nous n'avons sans doute pas besoin d'effectuer un tri-fusion de l'ensemble des fichiers
puisqu'on s'intéresse seulement aux cinq premières lignes. On doit pouvoir se contenter de trier chaque partie
de fichier lu et de ne conserver que les cinq premières lignes de chaque partie pour tout trier ensuite.
Voici donc ce qu'on va utiliser comme méthode :
-
déterminer le nombre n de lignes du fichier ;
-
lire la première moitié du fichier, trier et garder les 5 premières lignes ;
-
lire la seconde moitié du fichier, trier et garder les 5 premières lignes ;
-
fusionner les deux séries de 5 lignes, trier et ne retenir que les les 5 premières lignes.
Une première difficulté surgit avec la détermination du nombre de lignes en tout dans le gros fichier
et dans la lecture de chacune des parties : la fonction standard readLines() n'est pas prévue pour cela.
En effet, pour trouver le nombre de lignes du gros fichier initial, il n'est pas possible d'utiliser
la fonction de base readLines() car elle met en mémoire toutes les lignes du fichier.
Il faut utiliser la fonction determine_nlines() du package LaF dont le titre complet est
Fast access to large ASCII files. De même pour lire les lignes n1 à n2 d'un fichier,
il faut passer par la fonction
n.readLines() du package reader
dont le titre complet est Suite of Functions to Flexibly Read Data from Files.
Nous écrivons la fonction prem5(fichier) suivante pour automatiser les étapes 2 et 3
qui sont identiques, aux numéros de lignes à lire près :
library(reader) # pour la fonction n.readLines()
# fonction qui trie et renvoie les 5 premières lignes d'une
# partie de fichier
prem5 <- function(fichier,debut,fin) {
cat("lecture des lignes",debut,"à",fin,"dans le fichier",fichier,"\n")
lignes <- n.readLines(fichier, fin-debut+1,skip = debut-1, header = FALSE)
lignes <- sort(lignes)
return(lignes[1:5])
} # fin de fonction prem5
Dans la mesure où l'exécution prend beaucoup de temps, il est prudent de mettre un affichage dès qu'une étape
est réalisée. De plus, au niveau de chaque instruction, disons par exemple nbl <- determine_nlines(fichier),
dans une phase préliminaire, il est prudent d'afficher la durée d'exécution de l'instruction à l'aide la fonction
duree() des exercices de la séance 5, afin d'avoir une idée précise de la durée de chaque étape, soit le code :
duree( nbl <- determine_nlines(fichier) )
Une fois mis au moins et testé sur plusieurs fichiers,
le code qui implémente notre méthode est le suivant :
## les 5 premières lignes après tri dans un très gros fichier
library(LaF) # pour la fonction determine_nlines()
library(reader) # pour la fonction n.readLines()
# détermination du nombre de lignes du fichier
fichier <- "demo.fasta"
nbl <- determine_nlines(fichier)
milieu <- trunc(nbl/2)
cat("il y a",nbl," lignes dans ",fichier,"\n")
# lecture de la première moitié du fichier, tri et renvoi des 5 premières lignes
serie1 <- prem5(fichier,1,milieu)
cat("voici les 5 premières lignes de la première partie triée du fichier\n")
print(cbind(serie1[1:5]))
# lecture de la seconde moitié du fichier, tri et renvoi des 5 premières lignes
serie2 <- prem5(fichier,milieu+1,nbl)
cat("voici les 5 premières lignes de la seconde partie triée du fichier\n")
print(cbind(serie2[1:5]))
# fusion des deux séries de 5 lignes, tri et renvoi des 5 premières lignes
series <- sort( c(serie1,serie2) )
cat("voici les 5 premières lignes du fichier trié\n")
print(cbind(series[1:5]))
Pour en terminer avec cet exercice, on peut sans doute affirmer que le code sort(V,dec=TRUE) est vraiment plus
rapide que le code rev(sort(V)), avec un gain d'environ 10 % comme on peut s'en rendre
compte à l'aide nombreux exemples. Ainsi :
> V <- round(runif(n = 10**6,min=0,max=100))
> microbenchmark(
sort(V,dec=TRUE),
rev(sort(V))
) # fin de microbenchmark
Unit: milliseconds
expr min lq mean median uq max neval cld
sort(V, dec = TRUE) 79.60009 80.40262 82.45784 81.28188 82.04451 135.3483 100 a
rev(sort(V)) 85.17383 87.46311 93.85379 88.63074 89.68436 148.5425 100 b
Code-source php de cette page. Retour à la page principale du cours.
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)