Introduction à la programmation avec R
gilles.hunault "at" univ-angers.fr
Cours 8 - Déboggage, profilage et optimisation
Table des matières cliquable
1. But de cette section
2. Un comportement bizarre de R
3. Déboggage
4. Ecrire <<- n'est pas une faute de frappe
5. Profilage
6. Optimisation
7. Options d'exécution
Exercices : énoncés solutions [Retour à la page principale du cours]
1. But de cette section
Nous allons dans cette page essayer de répondre à trois questions :
-
Mon programme ne fonctionne pas comme je le voudrais. Où est l'erreur ?
-
Mon programme est lent. Quelle partie du code est la plus lente ?
-
Comment accélérer le code qui est lent ?
R fournit des outils qui permettent de répondre aux deux premières questions car après tout, il ne s'agit que d'analyser le code du programme ou son exécution. Par contre, c'est à vous de modifier le code pour le rendre plus rapide sachant que de par sa conception, R est parfois "lent" pour certaines opérations, bien que de nombreuses routines de base sont écrites en C ou C++.
Il est aussi possible que votre problème à traiter
soit un problème "difficile", NP-complet ou qu'il "explose combinatoirement", ce qui signifie
que de toutes façons il sera très très long à exécuter (si vous avez le choix, n'hésitez pas
à essayer de programmer ce genre de problèmes : comme c'est long, on peut passer beaucoup de
temps à la machine à café sans culpabiliser de peur de rater la fin de l'exécution
du programme !).
Nous essaierons de fournir pour cette dernière partie quelques pistes
de solutions, lorsqu'il doit être possible d'accélérer l'exécution, ce qui n'est
pas toujours le cas...
Mais commençons par un programme qui semble à la fois fonctionner et ne pas fonctionner,
et qui, en tous cas, ne s'arrête pas dès le début de l'exécution. Il ne s'agit donc pas
d'un problème simple à résoudre ou d'une erreur naïve de programmation.
2. Un comportement bizarre de R
Il est rare qu'un programme "marche" du premier coup. Parfois, même si
on est convaincu que R va faire ce qu'on lui demande et qu'à la main
on pense avoir résolu le problème, R semble se comporter bizarrement.
Voici un exemple de programme un peu "surprenant". On demande à R
de créer une matrice avec deux lignes et deux colonnes. L'utilisateur peut
alors supprimer la ligne ou la colonne de son choix et voir ce qu'il reste
de la matrice. Mais bizarrement, R a l'air de "perdre" la matrice
avant la fin...
Le code R associé, fourni ci-dessous, est court, simple à lire et à comprendre.
# un comportement bizarre de R
# 1. une matrice 2x2 a priori toute simple
mat <- matrix(1:4,nrow=2)
cat("\nVoici la matrice au départ,\n")
cat("avec",nrow(mat),"lignes et",ncol(mat),"colonnes\n")
print(mat)
# 2. suppression interactive de ligne ou de colonne
rep <- "OK"
while (rep=="OK") {
cat(" taper L pour supprimer une ligne, ")
cat(" ou C pour supprimer une colonne, ")
nature <- readline()
objet <- ifelse(nature=="L","ligne","colonne")
cat(" en quelle position ? ")
position <- as.numeric(readline())
if (objet=="ligne") {
mat <- mat[ -position, ]
} else {
mat <- mat[ ,-position ]
} # finsi
cat("Après suppression de la ",objet,"numéro",position,"\n")
cat("on dispose de la matrice suivante,\n")
cat("avec",nrow(mat),"lignes et",ncol(mat),"colonnes\n")
print(mat)
} # fin tant que
Mais l'exécution de ce code est un peu "mystérieuse" :
Voici la matrice au départ,
avec 2 lignes et 2 colonnes
[,1] [,2]
[1,] 1 3
[2,] 2 4
taper L pour supprimer une ligne, ou C pour supprimer une colonne, L
en quelle position ? 1
Après suppression de la ligne numéro 1
on dispose de la matrice suivante,
avec lignes et colonnes
[1] 2 4
taper L pour supprimer une ligne, ou C pour supprimer une colonne, C
en quelle position ? 1
Error in mat[, -position] : nombre de dimensions incorrect
Il semblerait que R n'arrive plus à trouver le nombre de lignes et de colonnes
de la matrice car au niveau de la dernière suppression, juste avant l'erreur,
il y a affiché "avec lignes" mais aucun nombre de lignes n'est affiché.
Pour un(e) professionnel(le) de R , ce comportement est normal car
il est décrit dans
l'aide (longue, très longue) de la fonction [() et dans l'aide de la fonction drop()
obtenues par help("[") et help(drop).
Sauf que les explications -- en anglais -- ne sont pas très explicites :
extrait de help("["])
---------------------
parameter drop: For matrices and arrays.
If 'TRUE' the result is coerced to the lowest
possible dimension.
extrait de help(drop)
---------------------
Array subsetting ('[') performs this reduction
unless used with 'drop = FALSE', but sometimes
it is useful to invoke 'drop' directly.
Pour essayer de comprendre ce qui se passe, on peut tenter d'aller
à la pêche (sous-entendu "aux informations") à l'aide de la fonction print() via print(mat)
ou de print(nrow(mat)).
Cela se révèlera sans doute difficile et peu productif parce que si x
n'est pas une matrice, nrow(x) renvoie NULL.
Est-ce un problème ?
Oui, mais c'est un peu subtil. Si R
affiche bien NULL pour NULL avec print(), concaténer NULL avec quelque chose
transforme NULL en chaine vide. De même, utiliser NULL dans cat()
renvoie aussi la chaine vide, et du coup on ne voit plus rien.
Voici avec un exemple pourquoi il est difficile de comprendre ce qui se passe avec un simple print() :
> print(NULL)
NULL
> cat(NULL)
> print(nrow(matrix(nrow=4)))
[1] 4
> cat(nrow(matrix(nrow=4)),"colonnes\n")
4 colonnes
> cat(nrow(vector(length=4)),"colonnes\n")
colonnes
Donc si on ne voit rien avec un print() de notre objet, il faudrait
sans doute essayer un print(class(mat)). Et là, miracle ! Voici la partie du code
qui a été légèrement modifié
cat("Après suppression de la ",objet,"numéro",position,"\n")
cat("on dispose d'une structure ",class(mat),"\n")
et l'affichage de l'exécution :
...
Après suppression de la ligne numéro 1
on dispose d'une structure integer
[1] 2 4
...
Nous avons donc identifié le problème : la variable mat change de nature. De "matrix"
elle passe à "integer", ce qui signifie vecteur d'entiers. Avec un peu plus d'entrainement,
c'était visible puisque R avait écrit, avec un seul indice
[1] 2 4
au lieu de l'affichage classique des matrices, avec deux séries d'indices :
[,1] [,2]
[1,] 2 4
Mais quand on débute en R , on n'est pas expert(e), bien sûr et ces subtilités d'affichage nous échappent
souvent.
Nous avons donc identifié l'erreur, ou du moins nous avons cerné le problème. Pour le résoudre,
l'aide de [() et de drop() indiquent clairement qu'il faut empêcher R de réduire la dimension
à l'aide du paramètre drop.
Donc en écrivant dans notre code
if (objet=="ligne") {
mat <- mat[ -position,, drop=FALSE ]
} else {
mat <- mat[ ,-position, drop=FALSE ]
} # finsi
le problème est résolu. Ouf !
Enfin, presque... comme souvent en programmation. Car du coup notre boucle... boucle !
Il faut donc prévoir d'arrêter de demander à supprimer des lignes ou des colonnes s'il n'y en a
plus, soit le code suivant juste avant la fin de la boucle tant que :
if ((nrow(mat)==0)|ncol(mat)==0) {
cat("il n'y a plus rien dans la matrice. Bye !\n")
rep <- "NO"
} # fin si
3. Déboggage
Ce que nous venons de faire est un peu du "bricolage" : pour trouver ce qui ne va pas, nous avons
ajouté des print() et nous avons réexécuté le programme.
R dispose d'outils beaucoup plus professionnels pour suivre le comportement des fonctions.
La première fonction utile à ce sujet est sans doute browser() -- à ne pas confondre
avec la fonction browseURL() qui affiche une URL dans le navigateur. Mettre browser()
dans le corps d'une fonction permet d'interrompre momentanément l'exécution de la fonction en cours
tout en ayant accès à R . On peut donc taper des commandes, interroger les variables ou les modifier...
Lorsque R exécute browser(), le prompt est modifié et affiche Browse[n].
Différentes commandes sont ajoutées à l'environnement habituel de R. Ainsi
n exécute juste une instruction,
c quitte ce browser et reprend l'exécution de la fonction en cours à l'instruction suivante dans la fonction,
f permet de terminer la boucle courante ou la fonction,
Q quitte le browser et la fonction.
Pour éviter d'avoir à ajouter browser() dans le code puis de l'enlever quand tout va bien -- il est sans doute
plus prudent de le mettre en commentaire et de le décommenter pour le réutiliser au prochain "bug" -- R fournit
les fonctions debug() et undebug. Ecrire debug(f) exécute systématiquement browser()
dès que la fonction f est appelée. undebug(f) vient annuler ce comportement. On peut se dispenser
d'exécuter undebug(f) si la fonction f est redéfinie, par exemple si on lit son code dans un fichier...
Pour bien comprendre comment tout cela fonctionne, supposons que la boucle "TANT QUE" de notre exemple précédent
soit dans la fonction gereMatrice, la matrice étant passée en paramètre.
Voici donc le code à dépanner :
# un comportement bizarre de R
gereMatrice <- function(mat,rep="OK") {
# affichage initial
cat("\nVoici la matrice au départ,\n")
cat("avec",nrow(mat),"lignes et",ncol(mat),"colonnes\n")
print(mat)
# suppression interactive de ligne ou de colonne
rep <- "OK"
while (rep=="OK") {
# même code qu'au début du chapitre
} # fin tant que
} # fin de fonction gereMatrice
L'encadré suivant montre un exemple de session interactive qui permet de suivre ce que fait R
au niveau du déboggage
(nous avons juste aménagé un peu le texte). On commence par indiquer à R
qu'on veut débugger notre fonction :
> debug(gereMatrice)
> gereMatrice( matrix(1:4,nrow=2) )
debugging in: gereMatrice(matrix(1:4, nrow = 2))
debug à bizar15.r#3 :{
[...] ### gh ### R affiche tout le code de fonction
Browse[2]>
On utilise ensuite n pour avancer dans l'exécution de la fonction.
R affiche chaque instruction avant de l'exécuter. Ici, nous avons appuyé 5
fois sur n, pour arriver à l'intérieur de la boucle :
Browse[2]> n
debug à bizar15.r#7 :cat("\nVoici la matrice au départ,\n")
Browse[2]> n
Voici la matrice au départ,
debug à bizar15.r#8 :cat("avec", nrow(mat), "lignes et", ncol(mat), "colonnes\n")
Browse[2]> n
avec 2 lignes et 2 colonnes
debug à bizar15.r#9 :print(mat)
Browse[2]> n
[,1] [,2]
[1,] 1 3
[2,] 2 4
debug à bizar15.r#13 :rep <- "OK"
Browse[2]> n
debug à bizar15.r#14 :while (rep == "OK") {
[...] ### gh ### R affiche tout le code de la boucle
Browse[2]>
Nous continuons l'exécution de la fonction avec des n successifs, pour arriver juste avant
l'exécution du code qui supprime une ligne ou une colonne :
Browse[2]> n
debug à bizar15.r#16 :cat(" taper L pour supprimer une ligne, ")
Browse[2]> n
taper L pour supprimer une ligne, debug à bizar15.r#17 :cat(" ou C pour supprimer une colonne, ")
Browse[2]> n
ou C pour supprimer une colonne, debug à bizar15.r#18 :nature <- readline()
Browse[2]> n
L
debug à bizar15.r#19 :objet <- ifelse(nature == "L", "ligne", "colonne")
Browse[2]> n
debug à bizar15.r#21 :cat(" en quelle position ? ")
Browse[2]> n
en quelle position ? debug à bizar15.r#22 :position <- as.numeric(readline())
Browse[2]> n
1
debug à bizar15.r#24 :if (objet == "ligne") {
mat <- mat[-position, ]
} else {
mat <- mat[, -position]
}
Browse[2]>
Maintenant, interrogeons nos divers objets, exécutons la suppression et regardons la classe et le
contenu de la matrice :
Browse[2]> objet
[1] "ligne"
Browse[2]> nature
[1] "L"
Browse[2]> class(mat)
[1] "matrix"
Browse[2]> n
debug à bizar15.r#25 :mat <- mat[-position, ]
Browse[2]> n
debug à bizar15.r#30 :cat("Après suppression de la ", objet, "numéro", position,
"\n")
Browse[2]> class(mat)
[1] "integer"
Browse[2]> mat
[1] 2 4
C'est fini, nous avons bien localisé l'endroit où R change la matrice. Il reste à taper Q
puis undebug(gereMatrice) pour revenir à un comportement "normal" de R :
Browse[2]> Q
> undebug(gereMatrice)
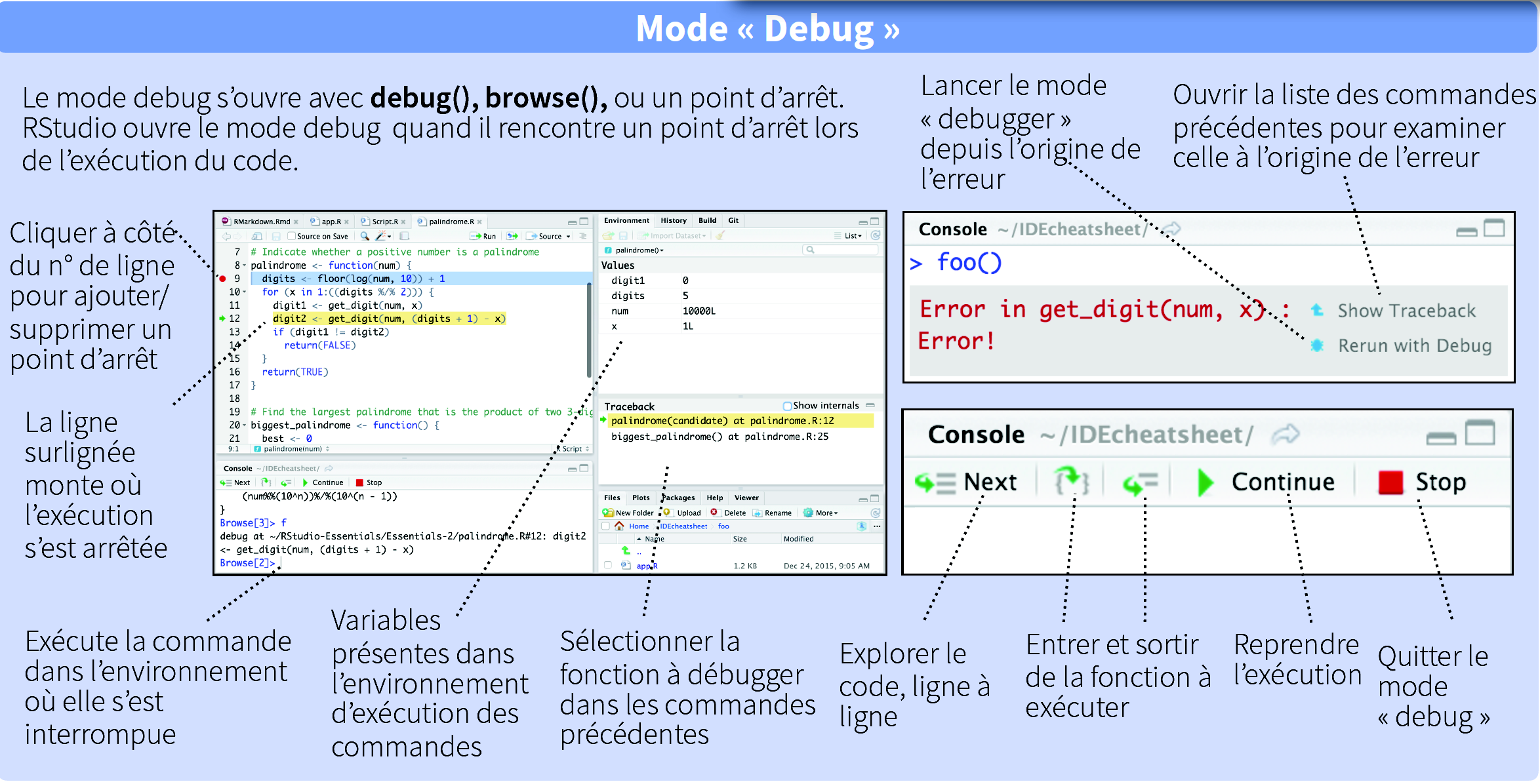
Il ne faut pas hésiter à recourir à la fonction debug() car c'est un outil très pratique
qui permet de localiser les erreurs. De plus, Rstudio
fournit un environnement adapté à debug() avec des boutons pour les actions
c, n... et le panneau de droite montre les variables locales...
A l'usage, c'est beaucoup plus complet et beaucoup plus pratique que le mode session de l'interface
standard de R.
L'image ci-dessous est extraite des cartes de référence de Rstudio accessibles à l'adresse cheatsheets.
4. Ecrire <<- n'est pas une faute de frappe
Il arrive parfois les programmes soient longs à s'exécuter et nous essaierons de gérer ce
problème dans la section suivante. Il arrive aussi qu'un programme s'exécute sans erreur apparente mais avec
un résultat faux au bout du compte. Pour peu qu'il y ait beaucoup de résultats intermédiaires, ou beaucoup
de variables et beaucoup de fonctions, la technique précédente avec debug() et browser est parfois un peu
"lourde" à utiliser, en particulier parce qu'il s'agit d'un environnement local. Une fois debug() quitté, les
variables locales utilisées par la fonction ont disparu.
R fournit avec l'instruction <<- un moyen intéressant de "sauvegarder" des variables.
Mais commençons par regarder cette disparition des variables.
Par défaut, toute variable dans une fonction est locale, ce qu'on peut voir avec le code suivant
pblv <- "non !" # pblv : Plus Belle La Vie
pourLesVieux <- function() {
pblv <- "OUI"
cat(" en local : ",pblv,"\n")
} # fin de fonction pourLesVieux
cat("global : ",pblv,"\n")
pourLesVieux()
cat("global : ",pblv,"\n")
dont l'exécution fournit :
global : non !
en local : OUI
global : non !
Une fonction l'exécution terminée, on n'a plus accès aux variables définies dans la fonction.
Si on écrit <<- au lieu de <-, R vient écrire la variable dans l'environnement
parent donc pour nous ici dans l'environnement global. Ainsi avec
pblv <- "non !" # pblv : Plus Belle La Vie
pourLesVieux <- function() {
pblv <<- "OUI" # AVEC DEUX SYMBOLES <
cat(" en local : ",pblv,"\n")
} # fin de fonction pourLesVieux
cat("global : ",pblv,"\n")
pourLesVieux()
cat("global : ",pblv,"\n")
on obtient :
global : non !
en local : OUI
global : OUI
Il est clair que pour notre exemple, cela ne sert pas à grand-chose. Pour un exemple en vraie grandeur,
utiliser <<- dans le corps de la fonction, ou même à l'intérieur de debug()
peut permettre de sauvegarder les "grosses" variables (listes nommées, data frame construits progressivement...)
dans différents états au cours de l'exécution afin de pouvoir comparer après coup les structures,
ou même simplement pour
documenter ce qui se passe en
début, en cours et en fin d'exécution...
5. Profilage
Une fois que les programmes sont justes et qu'il s'exécutent "bien", on peut vouloir
qu'ils s'exécutent "vite". Mais d'où vient la lenteur de notre code ?
Imaginons qu'on ait deux fonctions appelées à l'intérieur d'un programme. La lenteur
vient peut-être de la première fonction, ou de la deuxième ou peut-être même
des autres parties du programme. Pour tester cela, R fournit en standard plusieurs fonctions,
dont Rprof() et summaryRprof().
Voici comment les utiliser : on lance Rprof(), on exécute le code et ensuite
on demande à R via summaryRprof() d'afficher les résultats du profilage.
Imaginons par exemple qu'une fonction h()
appelle la fonction f()
et
la fonction g(), comme dans le code du fichier
lent01.r suivant
f <- function(x,n) {
for (nbfois in 1:n) {
x <- c(x,nbfois)
} # fin pour
return(x)
} # fin de fonction f
g <- function(x,n) {
for (nbfois in 1:n) {
x <- x + 1
} # fin pour
return(x)
} # fin de fonction g
h <- function(n) {
x <- 0
x <- f(x,n)
y <- 1
y <- g(y,n)
} # fin de fonction h
h(5*10**4)
Voici le code à utiliser pour analyser notre fonction h()
Rprof()
source("lent01.r",encoding="latin1")
Rprof(NULL)
summaryRprof()
Et son résultat sachant que les durées sont exprimées en secondes :
> summaryRprof()
$by.self
self.time self.pct total.time total.pct
"c" 5.00 98.43 5.00 98.43
"f" 0.06 1.18 5.06 99.61
"+" 0.02 0.39 0.02 0.39
$by.total
total.time total.pct self.time self.pct
"eval" 5.08 100.00 0.00 0.00
"h" 5.08 100.00 0.00 0.00
"source" 5.08 100.00 0.00 0.00
"withVisible" 5.08 100.00 0.00 0.00
"f" 5.06 99.61 0.06 1.18
"c" 5.00 98.43 5.00 98.43
"+" 0.02 0.39 0.02 0.39
"g" 0.02 0.39 0.00 0.00
$sample.interval
[1] 0.02
$sampling.time
[1] 5.08
Les quatre dernières lignes de l'affichage de $by.total
montrent clairement que les fonctions
f() et
c()
prennent beaucoup plus de temps à s'exécuter que les fonctions
+() et
g().
Pour tester plus finement du code, on peut utiliser le package nommé
microbenchmark. Par exemple, est-il plus rapide de générer un vecteur
de valeurs numériques suivant une loi uniforme ou une loi normale ?
Voici le code qui donne la réponse (il est plus lent de générer des données normales), avec son exécution :
> library("microbenchmark")
> n <- 3*10**5
> microbenchmark( a <- runif(n), b <- rnorm(n) )
Unit: milliseconds
expr min lq mean median uq max neval
a <- runif(n) 10.31694 10.32199 12.16642 10.32667 10.98350 59.37682 100
b <- rnorm(n) 25.70522 25.75042 27.25000 25.77834 27.35857 75.23661 100
De même, nous avions écrit qu'il vaut mieux éviter de réaliser
le même appel de fonction si les paramètres ne changent pas. Voici
un exemple chronométré de ce que cela signifie.
Soit le code R suivant à évaluer :
library("microbenchmark")
f <- function(v) {
return(list( sum(v==max(v)),which(v==max(v)) ))
} # fin de fonction f
g <- function(v) {
maxv <- max(v)
return(list( sum(v==maxv), which(v==maxv) ))
} # fin de fonction g
microbenchmark(
v <- runif(10**6),
f(v) ,
g(v)
) # fin de microbenchmark
Pour comprendre qu'il ne s'agit que d'une évaluation du temps passé, voici deux
exécutions de ce programme :
Unit: milliseconds
expr min lq mean median uq max neval cld
v <- runif(10^6) 34.39743 34.46519 40.33607 35.65167 36.15951 86.18001 100 c
f(v) 22.91168 24.65615 31.64044 24.83843 25.94697 73.81922 100 b
g(v) 19.31882 21.02488 26.43521 21.07547 22.05723 70.80157 100 a
Unit: milliseconds
expr min lq mean median uq max neval cld
v <- runif(10^6) 34.39903 35.06691 40.89298 36.01946 36.19025 85.32907 100 b
f(v) 22.92871 24.63554 30.57184 24.69739 25.35163 74.58290 100 a
g(v) 19.32942 21.01391 25.81828 21.06611 21.47420 71.03639 100 a
A la lecture de ces résultats, la fonction f est bien plus lente que la fonction g :
il ne faut donc pas calculer plusieurs fois
max(v) mais bien mettre cette valeur dans une variable comme maxv.
On peut aussi vouloir s'intéresser non pas aux fonctions mais aux lignes de code.
Le package lineprof permet un tel profilage et fournit un affichage adapté.
Attention : ce package n'est pas
disponible directement via le CRAN mais via github seulement.
Il utilise le package shiny pour un affichage interactif dans le navigateur.
Pour installer ce package lineprof, il faut
écrire (sous réserve, donc, que le package devtools soit installé au préalable) :
devtools::install_github("hadley/lineprof")
Admettons par exemple que l'on veuille analyser les lignes du code suivant :
lent <- function() {
print(date())
n <- 8*10**4
x <- c()
y <- 0
for (i in (1:n)) {
x <- c(x,i)
y <- y + 1
} # fin pour i
print(date())
} # fin de fonction lent
Pour cela, on écrit
library(lineprof) # chargement
profil <- lineprof( lent() ) # exécution
shine( profil ) # visualisation
Et on obtient dans le navigateur par défaut :
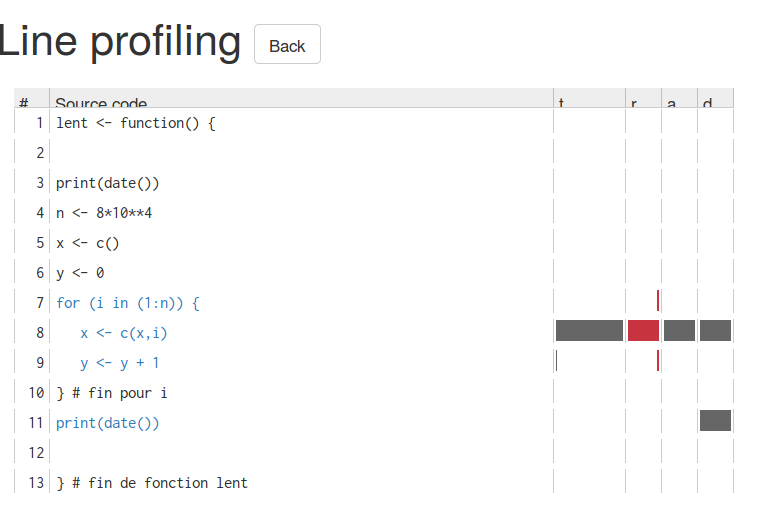
Il est facile de voir que la ligne 8 est la plus "consommatrice".
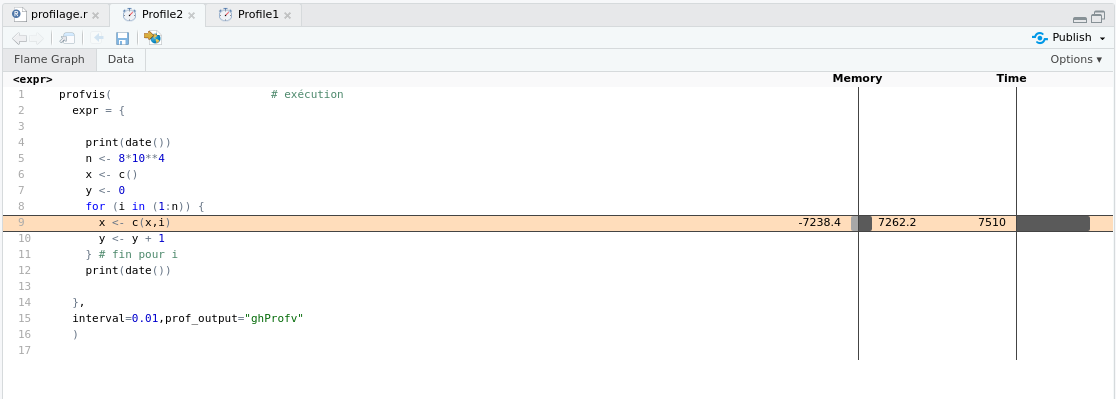
Depuis janvier 2019, il est conseillé d'utiliser profvis plutôt que lineprof. Voici comment on s'en sert, pour le même exemple :
library("profvis")
profvis(
expr = {
print(date())
n <- 8*10**4
x <- c()
y <- 0
for (i in (1:n)) {
x <- c(x,i)
y <- y + 1
} # fin pour i
print(date())
},
interval=0.01,prof_output="ghProfv"
) # fin de profvis
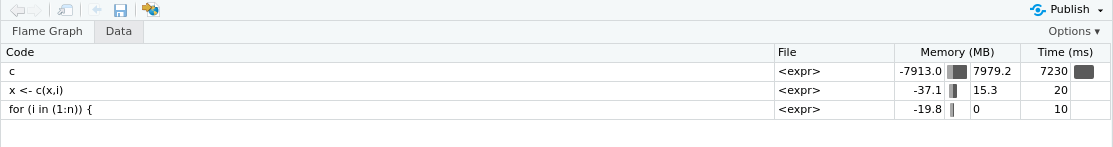
Ce qu'on obtient dans Rstudio est reproduit dans les deux images ci-dessous :
6. Optimisation
Maintenant que nous savons quelle partie de code est lente,
comment aller plus vite ?
C'est une question compliquée car il est possible que la méthode soit lente : si vous
avez décidé, par exemple, de passer en revue toutes les sous-chaines de caractères d'un génome humain, ce
sera forcément assez long, disons plusieurs dizaines de minutes sans doute.
Donc nous allons supposer que le code est "bon" mais lent. Une fois repérées les parties vraiment
lentes grâce à la section précédente, il faut essayer d'appliquer
quelques grands principes pour éviter qu'un code ne soit lent. Si ce n'est pas suffisant,
il faudra analyser si la lenteur vient du langage, en interne, ou si cela vient de notre façon de coder.
Tout d'abord, comme nous
venons de le voir, un premier grand principe est qu'il ne faut pas
effectuer deux fois le même calcul déterministe sur une structure si elle
ne change pas.
Donc si on utilise une boucle POUR, par exemple, il faut enlever tous les
calculs comme 3*h si h ne dépend pas de l'indice de boucle...
Ensuite, il faut essayer d'utiliser les fonctions vectorielles de R plutôt que
de réécrire soi-même les calculs.
Voici par exemple pourquoi il ne faut pas
réinventer la somme d'un vecteur :
somme1 <- function(v) {
return( sum(v) )
} # fin de fonction somme1
somme2 <- function(v) {
somme <- 0
for (element in v) {
somme <- somme + element
} # fin pour element
return( somme )
} # fin de fonction somme2
microbenchmark(
somme1(10**6),
somme2(10**6)
) # fin de microbenchmark
Unit: microseconds
expr min lq mean median uq max neval cld
somme1(10^6) 1.142 1.2790 1.93744 1.5270 1.6440 23.361 100 a
somme2(10^6) 1.313 1.5805 2.14648 1.7725 1.8815 20.343 100 a
Un deuxième grand principe est qu'il ne faut pas laisser R gérer la mémoire.
Malheureusement, ce n'est pas toujours facile de savoir quand R recopie les variables en mémoire.
Disons, pour simplifier, qu'il est prudent de prévoir au maximum les tailles des
tableaux résultats, de les initialiser et ensuite de les remplir.
Imaginons par exemple
que l'on veuille calculer n nombres "simples", par exemple tous les nombres de 1 à n.
Si vous écrivez cela en concaténant chaque nouveau nombre au vecteur précédent des résultats, soit le code
remplissage1 <- function( nbValeurs=8*10**4 ) {
vecRes <- c() # vecteur des résultats
for (ind in (1:nbValeurs)) {
vecRes <- c(vecRes,ind)
} # fin pour ind
return( vecRes )
} # fin de fonction remplissage1
alors R viendra de temps en temps ou peut-être même à chaque fois recopier le vecteur v
en mémoire. Il est beaucoup plus efficace de prévoir un vecteur de la "bonne taille" (voir l'exercice
3 associé à cette page qui explicite les stratégies possibles lorsqu'il n'est pas possible de prévoir avant l'exécution
la taille des résultats) et de le remplir au fur et à mesure, soit le code :
remplissage2 <- function( nbValeurs=8*10**4 ) {
# initialisation du vecteur des résultats avec la bonne taille
vecRes <- rep(0,nbValeurs)
# remplissage
for (ind in (1:nbValeurs)) {
vecRes[ind] <- ind
} # fin pour ind
return(vecRes)
} # fin de fonction remplissage2
Mais de toutes façons ce ne sera jamais aussi efficace que le code R natif et vectoriel :
remplissage3 <- function( nbValeurs=8*10**4 ) {
vecRes <- 1:nbValeurs
return( vecRes )
} # fin de fonction remplissage3
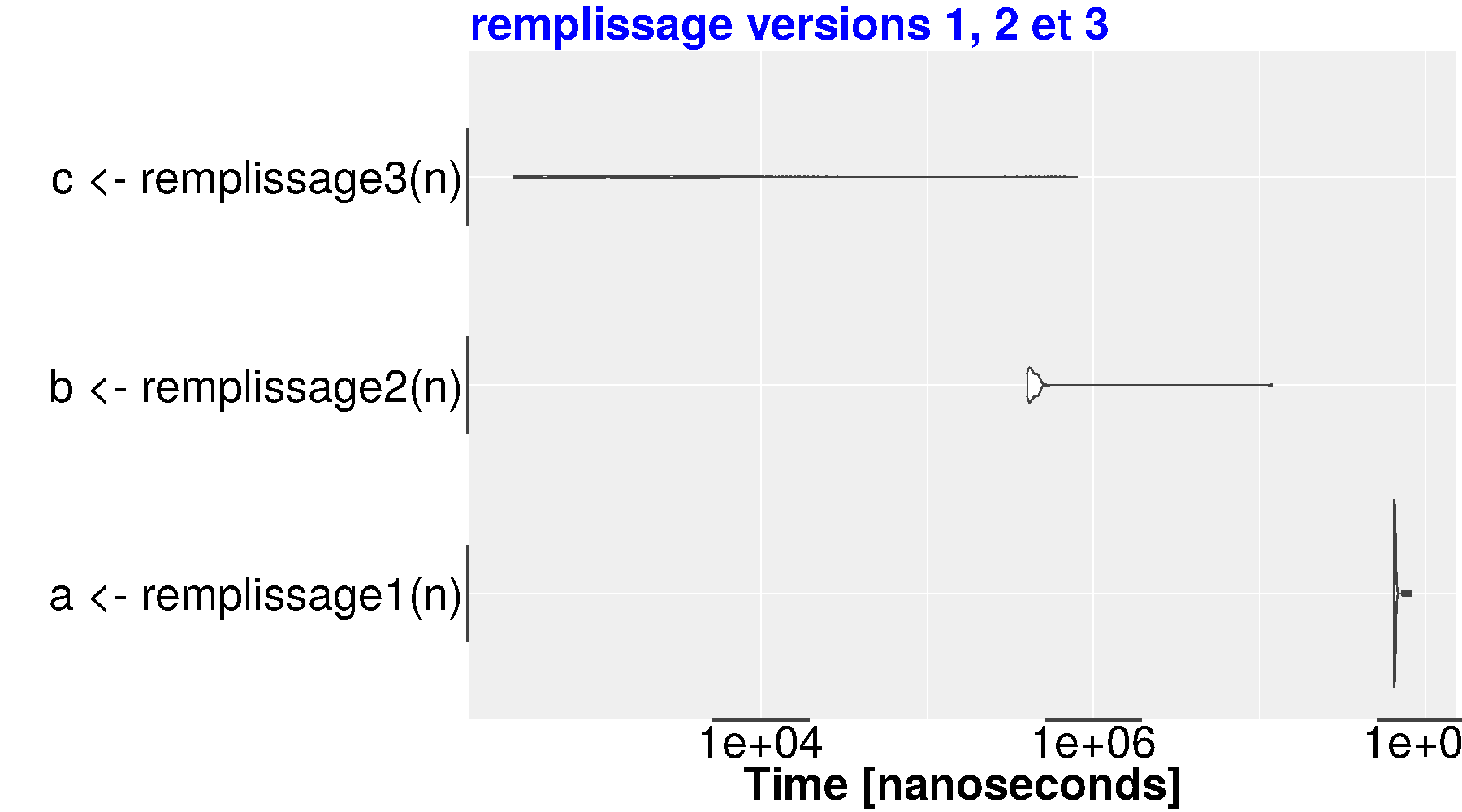
En voici la preuve, avec une machine qui a beaucoup de mémoire. Nous avons pris la précaution
de vérifier que l'ordre des instructions ne perturbe pas les résultats (à cause des
encombrements mémoire successifs) :
> library("microbenchmark")
> n <- 10**4
> microbenchmark(
a <- remplissage1(n),
b <- remplissage2(n),
c <- remplissage3(n)
) # fin de microbenchmark
Unit: microseconds
expr min lq mean median uq max neval cld
a <- remplissage1(n) 98655.609 101171.269 104653.99511 102550.045 103735.207 143625.852 100 c
b <- remplissage2(n) 6794.431 7388.097 8092.45409 7584.002 8710.362 11176.973 100 b
c <- remplissage3(n) 5.004 6.139 8.82518 9.639 10.759 14.907 100 a
> microbenchmark(
c <- remplissage3(n),
a <- remplissage1(n),
b <- remplissage2(n)
) # fin de microbenchmark
Unit: microseconds
expr min lq mean median uq max neval cld
c <- remplissage3(n) 4.952 6.2715 9.05478 9.7185 10.7445 20.37 100 a
a <- remplissage1(n) 101334.792 103374.7525 107689.51062 104629.8870 106394.4925 150000.07 100 c
b <- remplissage2(n) 6680.088 7267.6015 7902.64836 7575.8575 8368.8110 11169.16 100 b
Il est possible de visualiser via ggplot les profils d'exécution :
library(microbenchmark)
n <- 10**4
resSort <- microbenchmark(
a <- remplissage1(n),
b <- remplissage2(n),
c <- remplissage3(n)
) # fin de microbenchmark
library(ggplot2)
cairo_ps("remplissage.eps",pointsize=20,fallback_resolution=600,width=18,height=10)
autoplot(resSort) +
ggtitle("remplissage versions 1, 2 et 3") +
theme(plot.title = element_text(size=40, face="bold",colour="blue")) +
theme(axis.title = element_text(size=40, face="bold")) +
theme(axis.ticks = element_line(size=40)) +
theme(axis.text = element_text(size=40,colour="black"))
dev.off()
7. Options d'exécution
Les codes R précédents ont été exécutés via Rstudio. Il existe une autre alternative : la ligne de commandes. On utilise pour cela soit R soit Rscript en console, dans un terminal. On dispose alors de nombreuses options, pour gérer l'exécution, l'environnement. Voici l'aide la commande R :
$gh> R --help
Usage: R [options] [< infile] [> outfile]
or: R CMD command [arguments]
Start R, a system for statistical computation and graphics, with the
specified options, or invoke an R tool via the 'R CMD' interface.
Options:
-h, --help Print short help message and exit
--version Print version info and exit
--encoding=ENC Specify encoding to be used for stdin
--encoding ENC
RHOME Print path to R home directory and exit
--save Do save workspace at the end of the session
--no-save Don't save it
--no-environ Don't read the site and user environment files
--no-site-file Don't read the site-wide Rprofile
--no-init-file Don't read the user R profile
--restore Do restore previously saved objects at startup
--no-restore-data Don't restore previously saved objects
--no-restore-history Don't restore the R history file
--no-restore Don't restore anything
--vanilla Combine --no-save, --no-restore, --no-site-file,
--no-init-file and --no-environ
--no-readline Don't use readline for command-line editing
--max-ppsize=N Set max size of protect stack to N
--min-nsize=N Set min number of fixed size obj's ("cons cells") to N
--min-vsize=N Set vector heap minimum to N bytes; '4M' = 4 MegaB
-q, --quiet Don't print startup message
--silent Same as --quiet
--slave Make R run as quietly as possible
--interactive Force an interactive session
--verbose Print more information about progress
-d, --debugger=NAME Run R through debugger NAME
--debugger-args=ARGS Pass ARGS as arguments to the debugger
-g TYPE, --gui=TYPE Use TYPE as GUI; possible values are 'X11' (default)
and 'Tk'.
--arch=NAME Specify a sub-architecture
--args Skip the rest of the command line
-f FILE, --file=FILE Take input from 'FILE'
-e EXPR Execute 'EXPR' and exit
FILE may contain spaces but not shell metacharacters.
Commands:
BATCH Run R in batch mode
COMPILE Compile files for use with R
SHLIB Build shared library for dynamic loading
INSTALL Install add-on packages
REMOVE Remove add-on packages
build Build add-on packages
check Check add-on packages
LINK Front-end for creating executable programs
Rprof Post-process R profiling files
Rdconv Convert Rd format to various other formats
Rd2pdf Convert Rd format to PDF
Rd2txt Convert Rd format to pretty text
Stangle Extract S/R code from Sweave documentation
Sweave Process Sweave documentation
Rdiff Diff R output ignoring headers etc
config Obtain configuration information about R
javareconf Update the Java configuration variables
rtags Create Emacs-style tag files from C, R, and Rd files
Please use 'R CMD command --help' to obtain further information about
the usage of 'command'.
Options --arch, --no-environ, --no-init-file, --no-site-file and --vanilla
can be placed between R and CMD, to apply to R processes run by 'command'
Report bugs at <https://bugs.R-project.org>.
Et celle de la commande Rscript :
$gh> Rscript
Usage: /path/to/Rscript [--options] [-e expr [-e expr2 ...] | file] [args]
--options accepted are
--help Print usage and exit
--version Print version and exit
--verbose Print information on progress
--default-packages=list
Where 'list' is a comma-separated set
of package names, or 'NULL'
or options to R, in addition to --slave --no-restore, such as
--save Do save workspace at the end of the session
--no-environ Don't read the site and user environment files
--no-site-file Don't read the site-wide Rprofile
--no-init-file Don't read the user R profile
--restore Do restore previously saved objects at startup
--vanilla Combine --no-save, --no-restore, --no-site-file
--no-init-file and --no-environ
'file' may contain spaces but not shell metacharacters
Expressions (one or more '-e <expr>') may be used *instead* of 'file'
See also ?Rscript from within R
On poura consulter le tp3 de notre cours DECRA afin de voir comment tester et passer les paramètres à R et Rscript.
Exercices : énoncés solutions [Retour à la page principale du cours]
|  Retour à la page principale de
(gH)
Retour à la page principale de
(gH)




