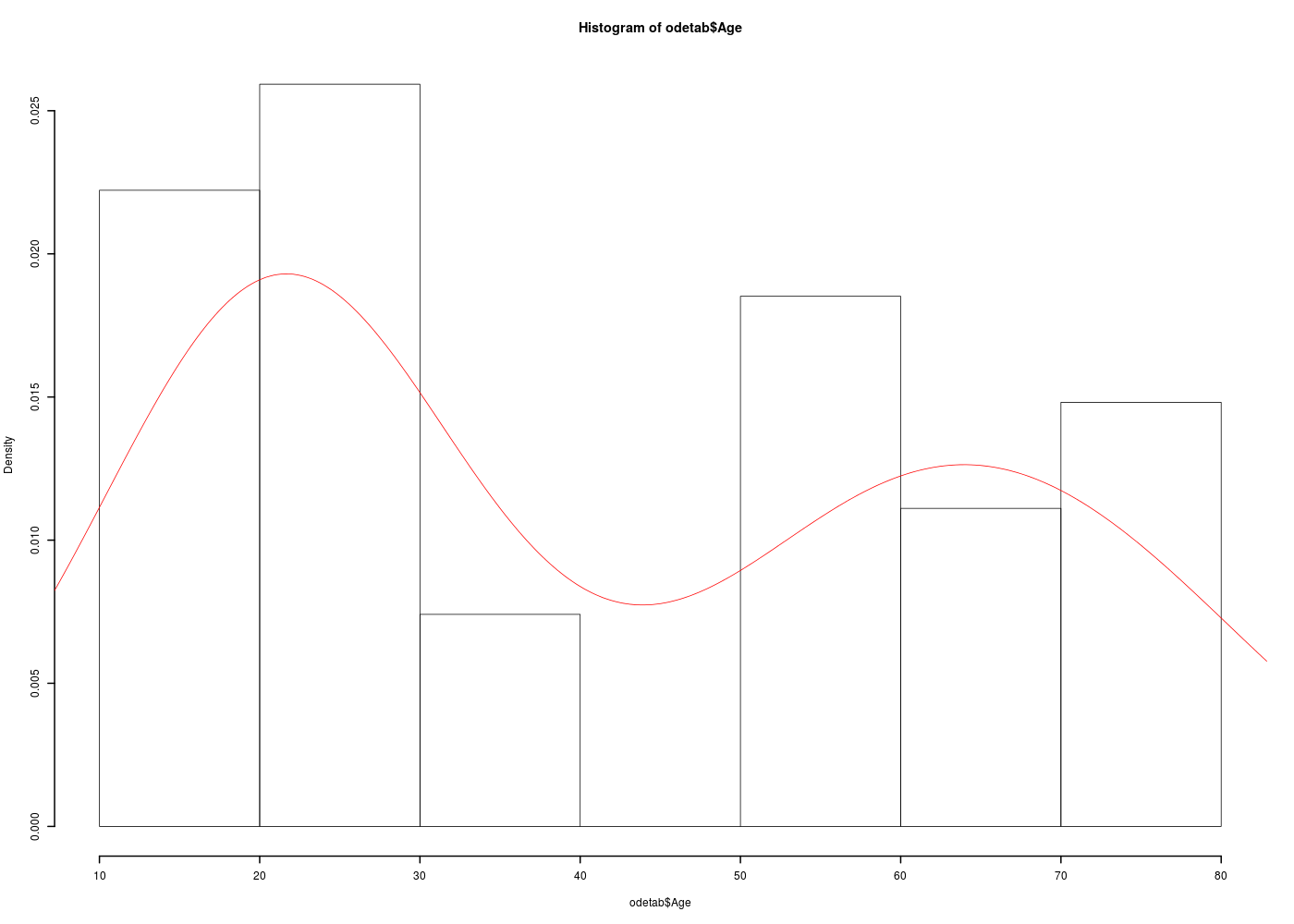
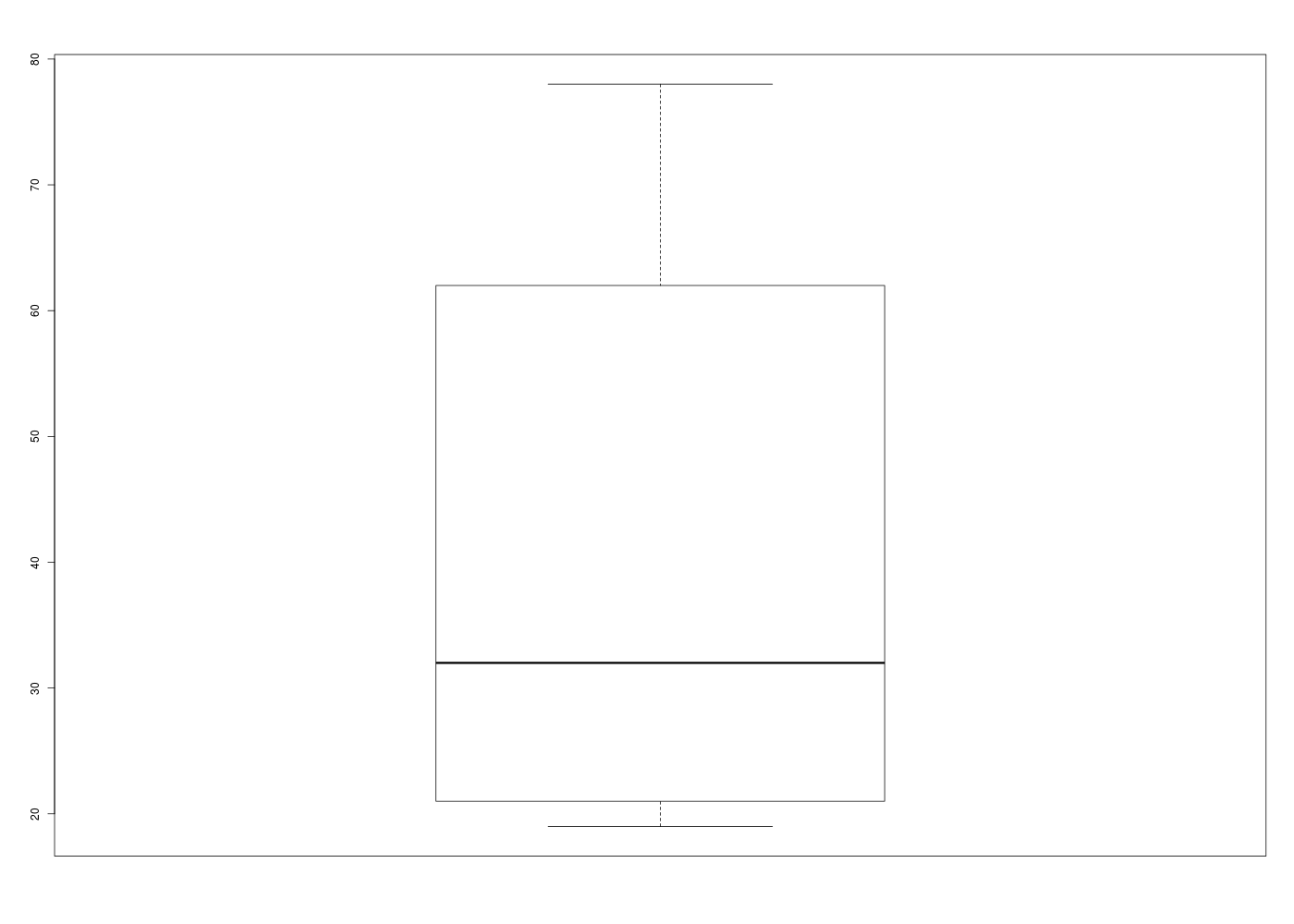
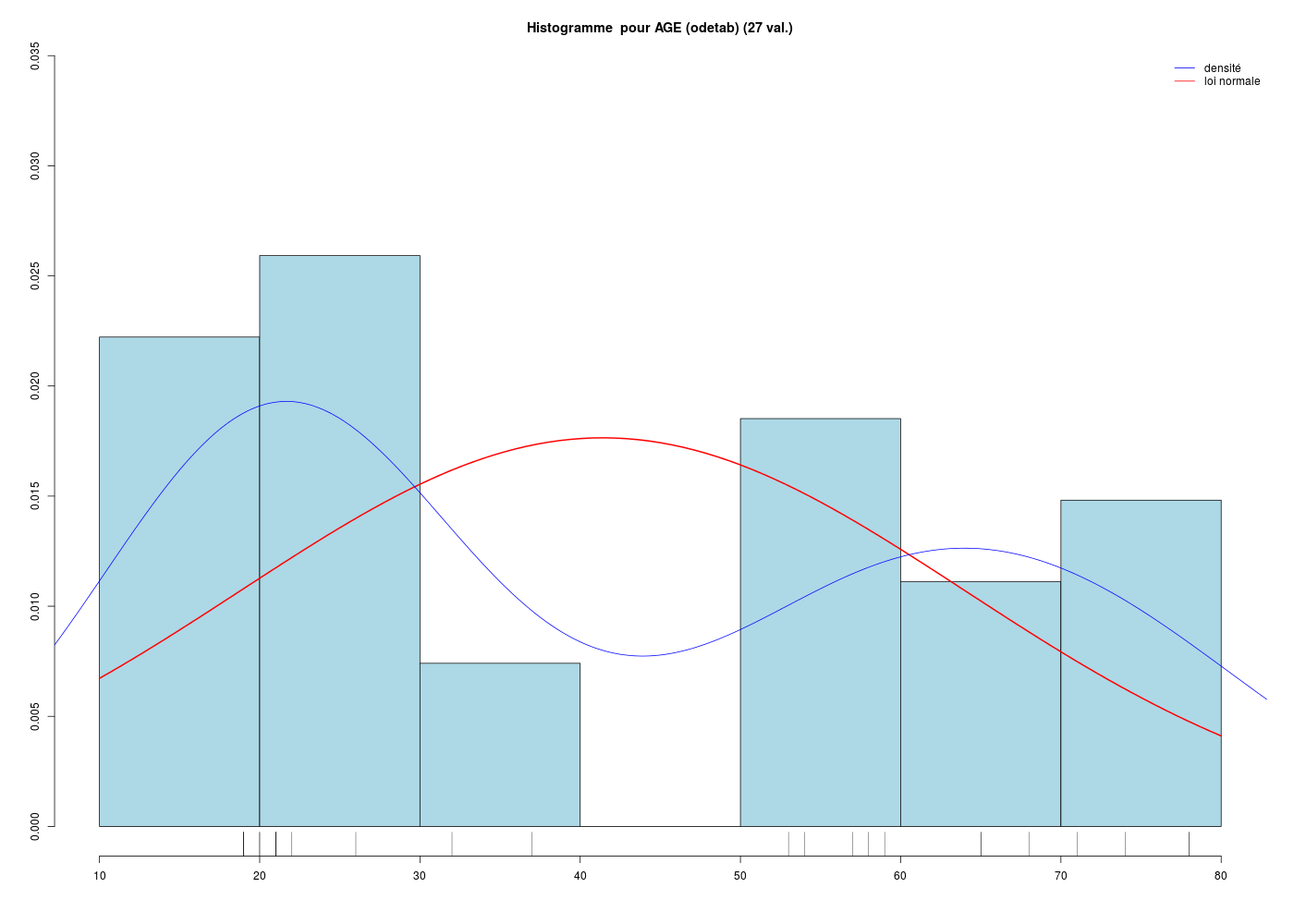
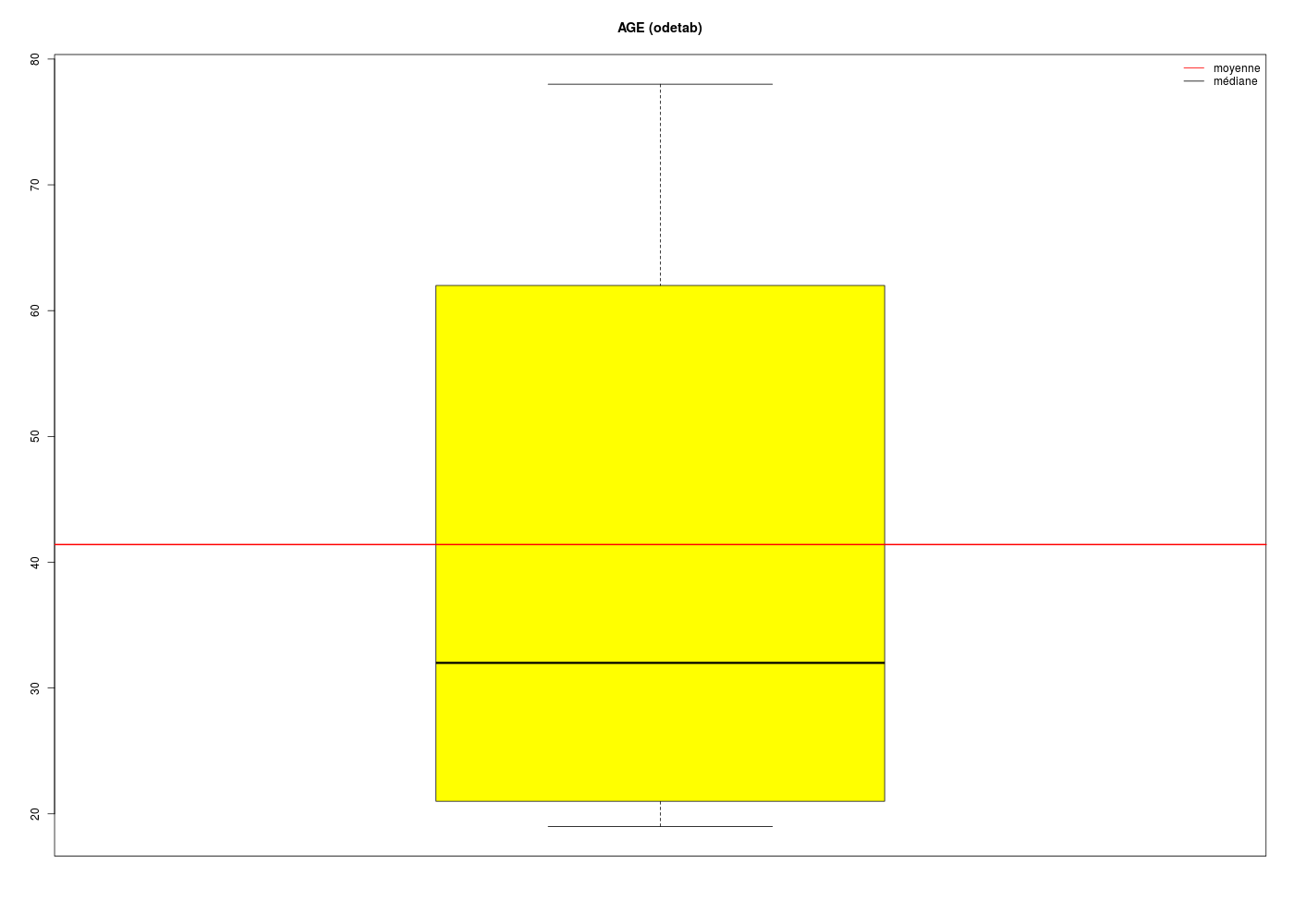
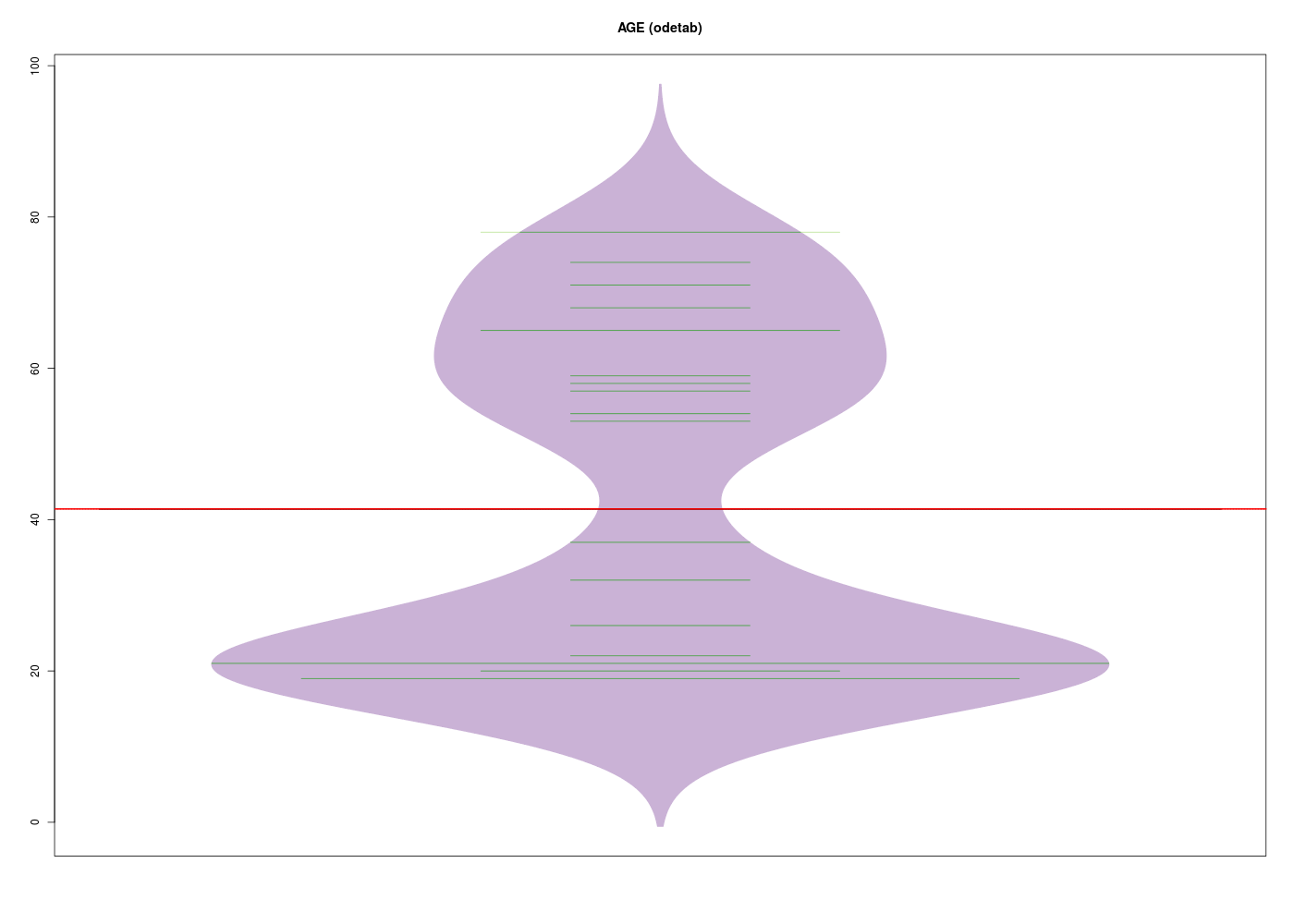
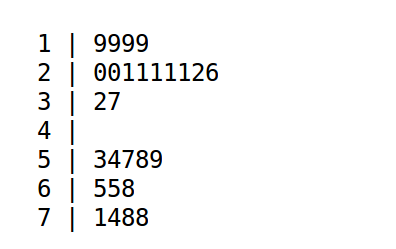
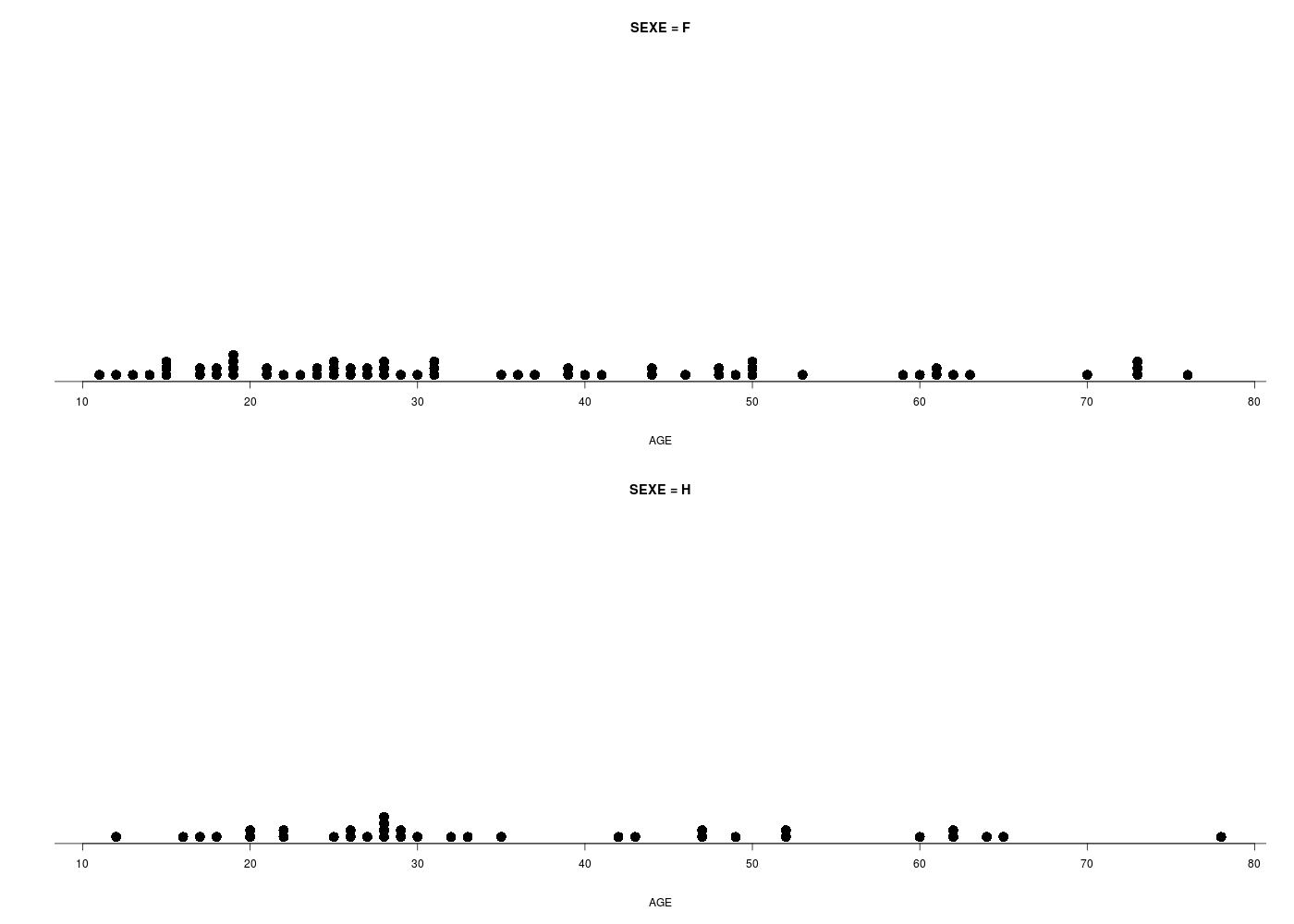
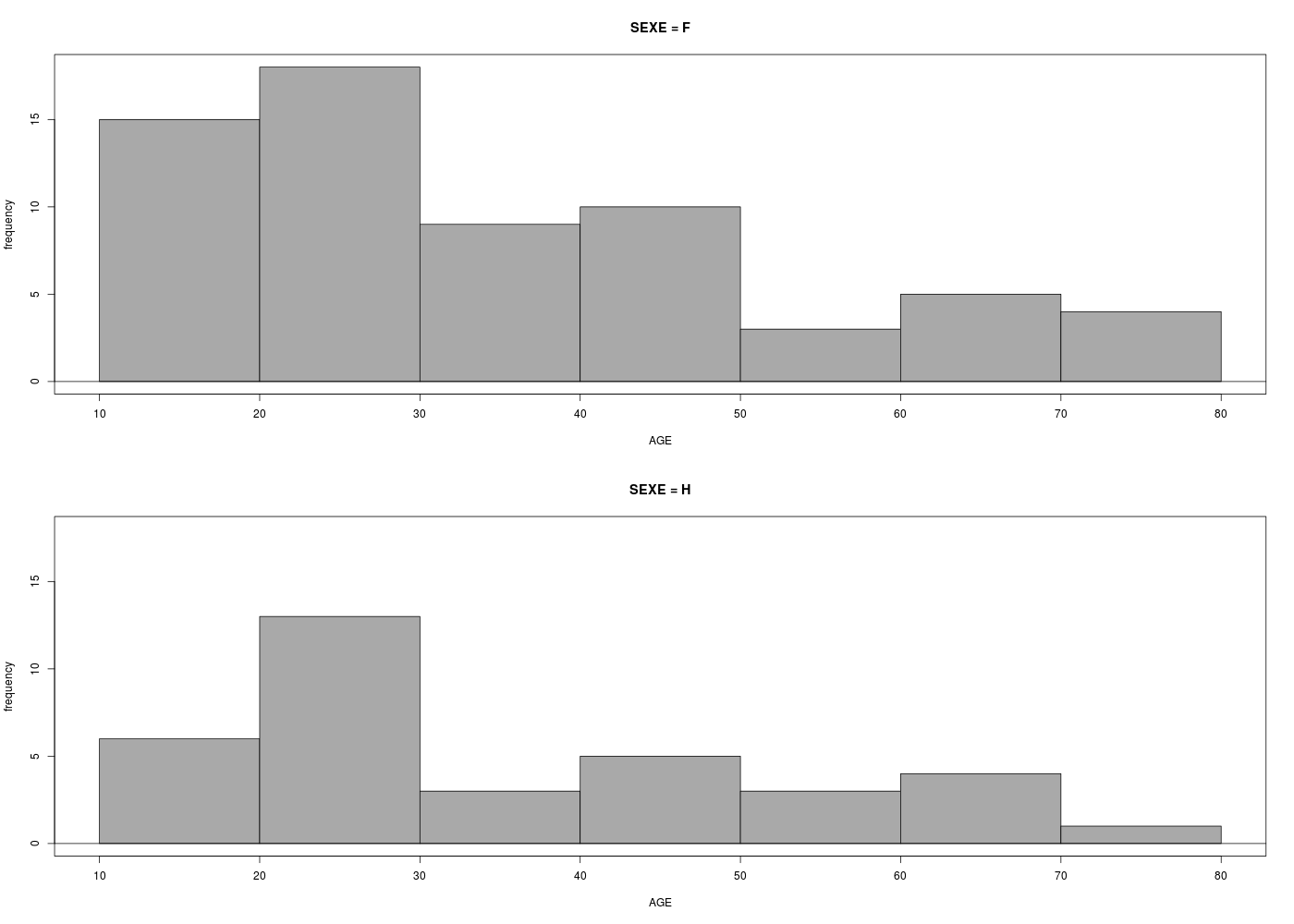
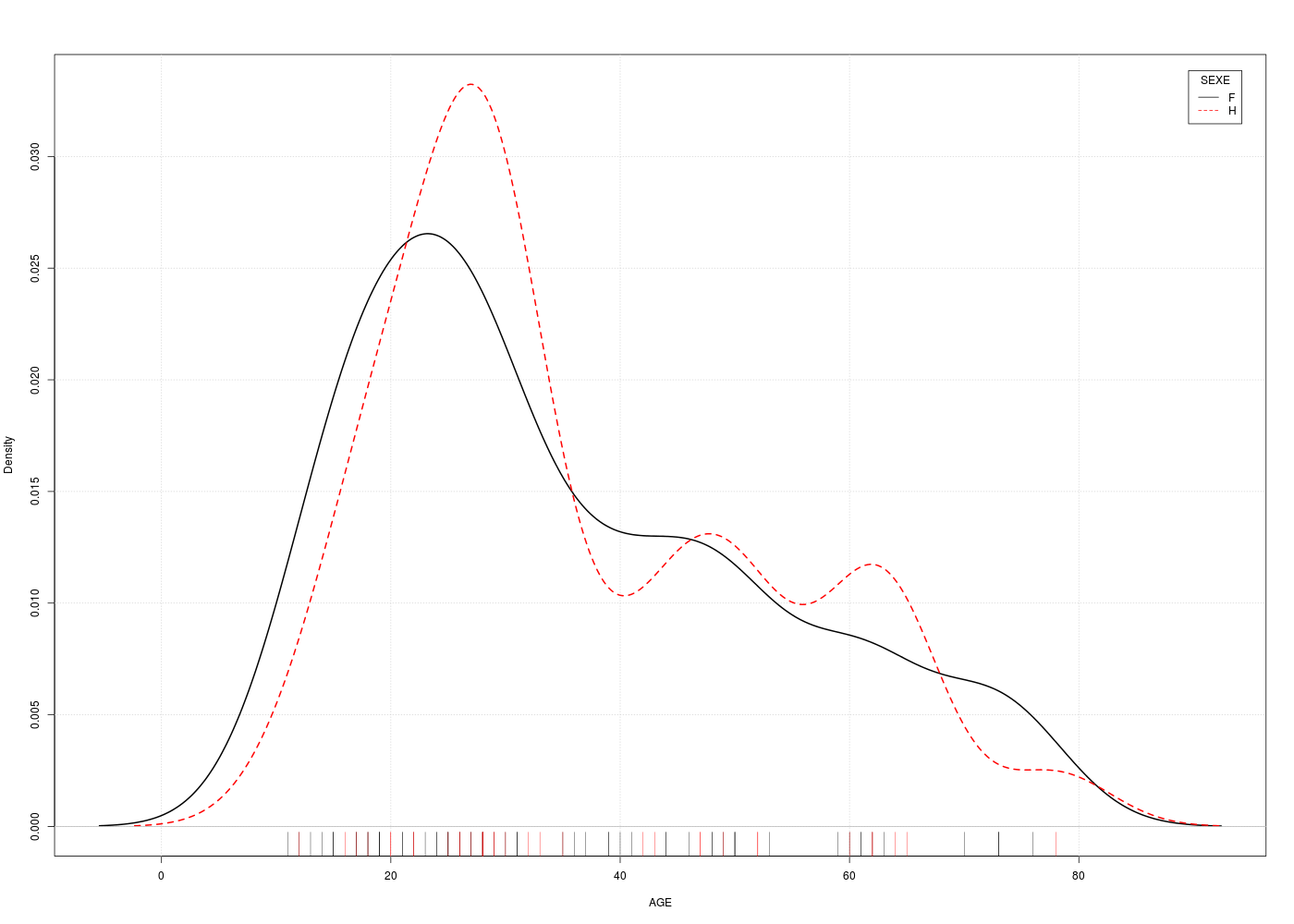
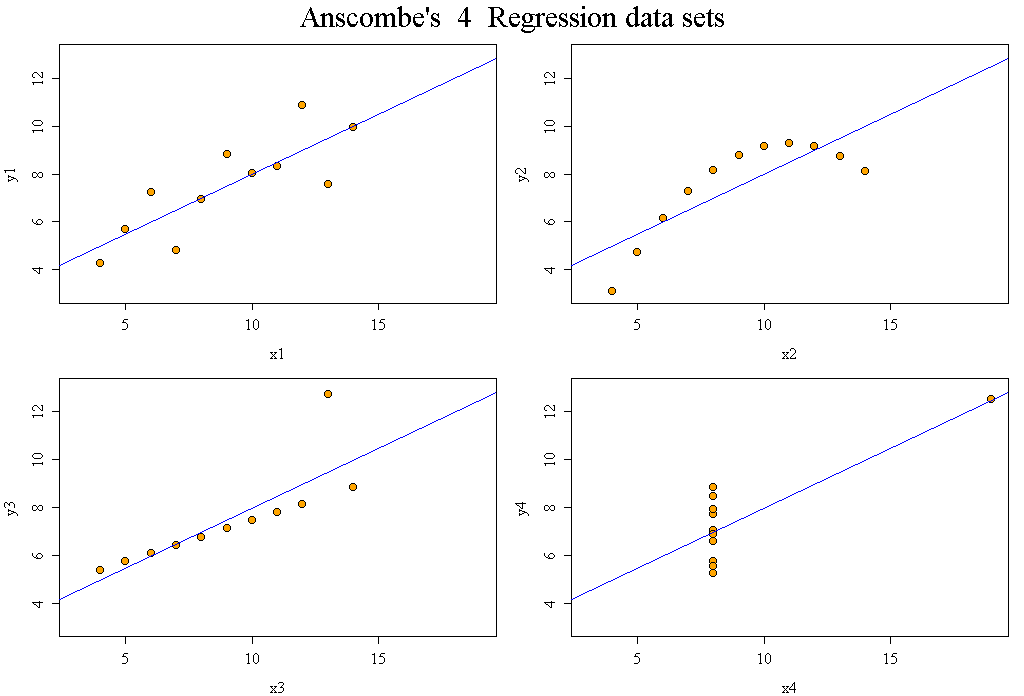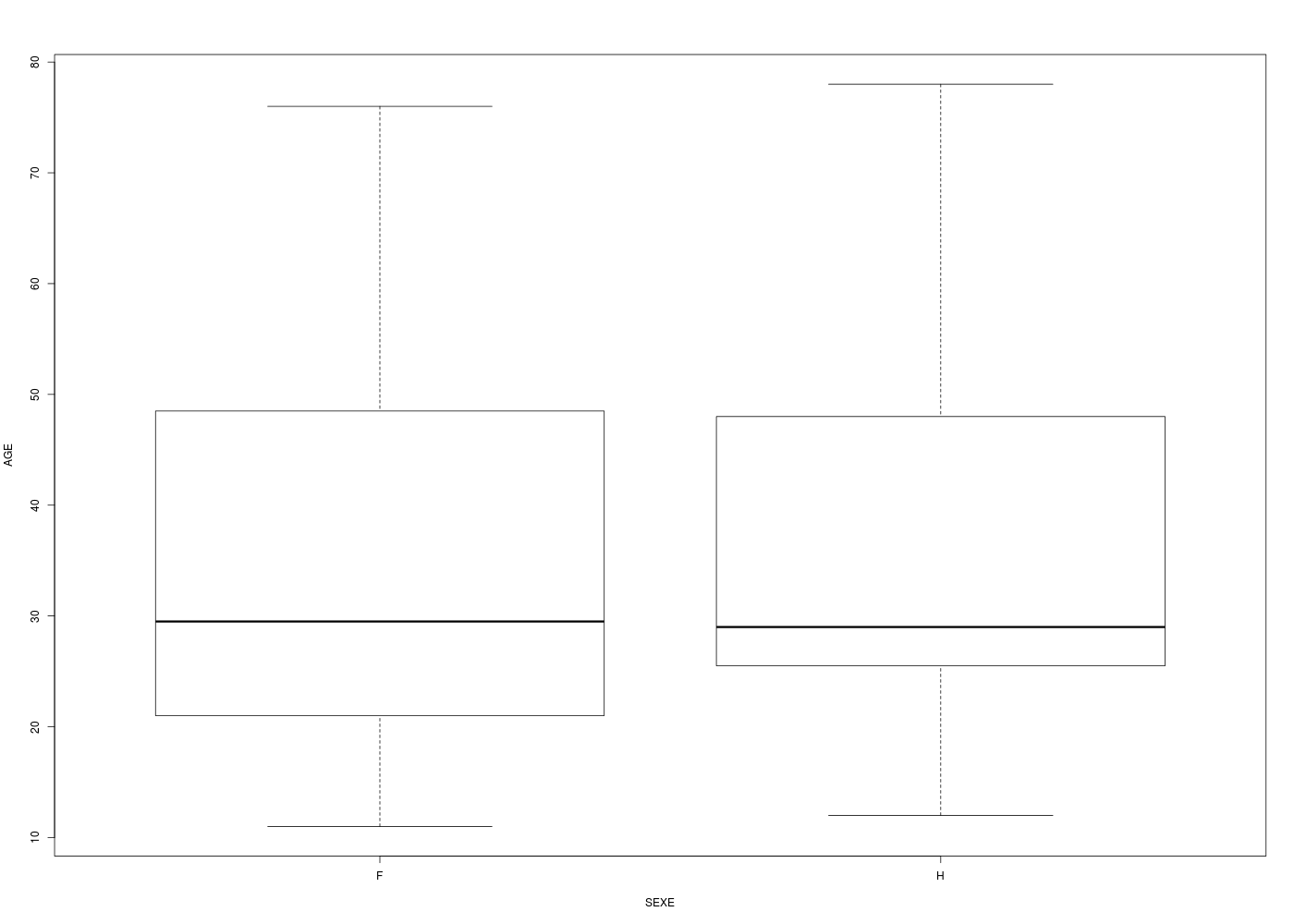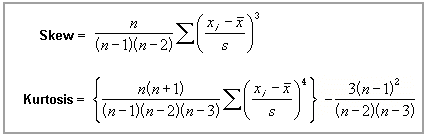Représenter graphiquement la variable AGE pour les données odetab. La distribution est-elle unimodale (une bosse) ou bimodale (deux bosses) ?
Que peut-on en déduire sur le calcul de la médiane ou de la moyenne pour ces données d'age ?
Un graphique comme l'histogramme de classes (avec l'estimation de la densité sous-jacente) montre qu'il y a deux bosses, de même qu'un diagramme en tiges et feuilles. Par contre une boite à moustaches classique ne peut pas le montrer :
Graphiques R standards :
Avec les fonctions de statgh.r il y a plus de couleurs et surtout, il y a une fonction beanplotQT qui utilise la fonction beanplot() du package éponyme qui montre bien ces deux bosses.
Voici les graphiques associés (nous avons aussi ajouté un tracé en tige et feuilles) :
L'ensemble du code R est le suivant :
## representation graphique de l'age, DONNEES ODETAB
# lecture du fichier Excel
library("gdata")
odetab <- read.xls("odetabExtrait1.xls")
# tracé standard
hist(odetab$Age,probability=TRUE,lwd=2)
lines(density(odetab$Age),col="red")
# boxplot standard
boxplot(odetab$Age)
# tracé avec les fonctions de statgh.r
boxplotQT("AGE (odetab)",odetab$Age,"ans")
histoQT("AGE (odetab)",odetab$Age,"ans")
# utilisation d'un beanplot
beanplotQT("AGE (odetab)",odetab$Age)
# tracé en tige et feuilles
stem(odetab$Age)
La conséquence de la séparation des données en deux sous-groupes très distincts est que le calcul de la moyenne ou de la médiane n'est pas pertinent ici puisqu'il fournit un seul résumé central inadapté moy=41 ans, med=42 ans, min=19, max=78 alors qu'il y a clairement deux groupes, un groupe de «jeunes» age<40, n=15, moy=22, med=21, min=19, max=37 et un groupes de «vieux» age>40, n=12, moy=65, med=65, min=53, max=78.
Ce genre de situation est classique et normale quand on réalise une étude dite cas-témoins : on choisit des témoins (sans maladie) et des cas (avec maladie) afin d'essayer de caractériser la pathologie. En effet, une étude détaillée de la variable d'intérêt "SOURD" (nom de variable maladroit) montre que les "jeunes" sont bien entendant ou normo-entendant alors que les "vieux" sont mal entendant. Revoir si nécessaire l'exercice 5 de la séance 3 à ce sujet.
On s'intéresse à des valeurs d'AGE pour des personnes. Peut-on commenter directement les résultats numériques ci-dessous :
Variable AGE, dossier HERS
n=2763 moy=66.65 med=67 ect=6.65 iqr=10 cdv=10% iqrr=15% min=44 max=79 pdv=35
Variable AGE, dossier ELF
n=99 moy=35.83 med=29 ect=17.55 iqr=26.5 cdv=49% iqrr=91% min=11 max=78 pdv=67
Variable AGE, dossier TRIOLA
n=80 moy=34.35 med=32 ect=13.18 idr=18.75 cdv=38% iqrr=59% min=12 max=73 pdv=61
Variable AGE, dossier ODETAB
n=27 moy=41.41 med=32 ect=22.61 iqr=41 cdv=54% iqrr=128% min=19 max=78 pdv=59
Variable AGE, dossier FIBROSE
n=1011 moy=45.42 med=43.3 ect=12.50 iqr=17.25 cdv=28% iqrr=40% min=8.3 max=79.0 pdv=7.07
Peut-on penser que, pour une petite série de données, connaitre la moyenne et l'écart-type permet de se faire une bonne idée des données ? Par exemple, comment sont distribuées les données avec les valeurs suivantes :
Taille 11 valeurs
Moyenne 7.5 UI
Ecart-type 2.0 UI
Coef. de variation 26 %
Hélas, on a cru longtemps que ces informations étaient suffisantes pour décire les variables QT, mais on sait aujourd'ui qu'en fait elles peuvent recouvrir des réalités différentes. Sans aucune autre information et donc sans savoir le contexte de l'étude et de la collecte des données, on ne peut dire que des choses très générales. Ainsi les données HERS et FIBROSE correspondent à des grands échantillons, les données ELF et TRIOLA ont une taille d'échantillon raisonnable pour "observer des phénomènes", les données ODETAB constituent un petit échantillon.
Par comparaison entre ces résultats, seule la distribution de l'age pour les données HERS parait très homogène (cdv 10 %), celle pour les données FIBROSE est "assez homogène" (cdv 28 %). A l'opposé, les données ELF et ODETAB semblent les plus "dispersées" (cdv respectifs 49 et 54 %, iqrr 91 et 128 %).
Si on connait le domaine, on peut sans doute aller plus loin comme l'age 8.3 sans doute incorrect comme valeur minimale pour une étude sur la fibrose hépatique, un age minimal très faible aussi pour les données ELF et TRIOLA...
Par contre la présence de valeurs après la virgule dans l'age (données FIBROSE) n'est sans doute pas une erreur mais le résultat d'un calcul de l'age à partir de la date de naissance.
Même pour pour une petite série de données on ne peut pas savoir grand chose au vu seul de la moyenne et de l'écart-type. Les valeurs fournies ici sont celles du fameux exemple des quartets d'Anscombe, données reproduites ci-dessous.
ID X Y1 Y2 Y3 X4 Y4
a 4 4.26 3.10 5.39 19 12.50
b 5 5.68 4.74 5.73 8 6.89
c 6 7.24 6.13 6.08 8 5.25
d 7 4.82 7.26 6.42 8 7.91
e 8 6.95 8.14 6.77 8 5.76
f 9 8.81 8.77 7.11 8 8.84
g 10 8.04 9.14 7.46 8 6.58
h 11 8.33 9.26 7.81 8 8.47
i 12 10.84 9.13 8.15 8 5.56
j 13 7.58 8.74 12.74 8 7.71
k 14 9.96 8.10 8.84 8 7.04
Ces données d'Anscombe correspondent à 4 jeux de données qui donnent les mêmes résultats numériques de régression linéaire simple alors que les données sont très différentes. Il faut donc toujours utiliser des représentations graphiques au lieu de se baser uniquement sur les résultats chiffrés.
# lecture des données
qansc <- lit.dar("http://www.info.univ-angers.fr/~gh/Datasets/anscombe.dar")
# résumés "à l'aveugle"
cats("les 4 colonnes semblent peu différentes, en résumé")
cols <- paste("Y",1:4,sep="")
mdr <- matrix(nrow=2,ncol=length(cols))
dy <- qansc[,cols]
mdr[1,] <- colMeans( dy )
mdr[2,] <- apply(X=dy,FUN=sd,MARGIN=2)
colnames(mdr) <- cols
row.names(mdr) <- c("Moyennes","Ecarts-type")
print(mdr)
les 4 colonnes semblent peu différentes, en résumé
==================================================
Y1 Y2 Y3 Y4
Moyennes 7.500909 7.500909 7.500000 7.500909
Ecarts-type 2.031568 2.031657 2.030424 2.030579
On consultera la page du wiki Anscombe pour plus d'information et on pourra lire la page d'aide associée au jeu de données anscombe du package datasets, aide disponible par help(anscombe) sous R, le tracé étant affiché par example(anscombe).
Il est très classique d'avoir à effectuer des calculs sur des variables QT restreintes à des sous-groupes définis par des variables QL, comme l'étude de l'age par sexe, ou le taux de réussite à un test en fonction du degré de pathologie. Comment réaliser cela avec R et Rstudio ?
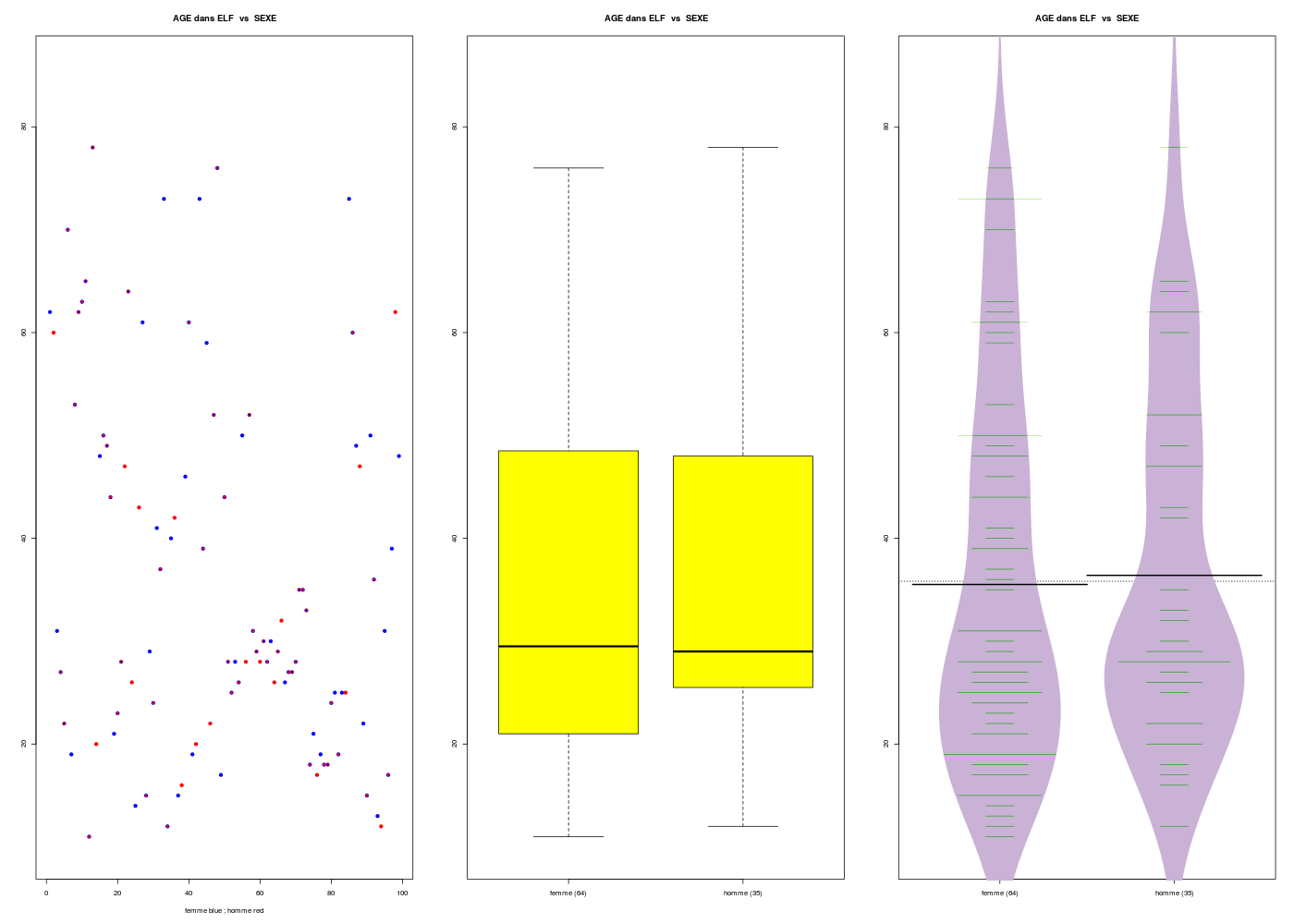
Application : étudier l'AGE en fonction du SEXE dans le dossier ELF.
Nous avions déjà vu comment faire à l'exercice 3 de la séance 2 : on utilise en général les fonctions lapply() et sapply(), voire tapply().
Notre fonction decritQTparFacteur réalise à la fois les calculs et les graphiques pour ce type d'analyse :
## CALCULS ET GRAPHIQUES PAR SOUS-GROUPES
## avec des fonctions de statgh.r
# lecture des données
library(gdata)
elf <- read.xls("elfOrthoQL.xls")
levels(elf$SEXE) <- c("Femme","Homme")
# analyse par sous-groupe
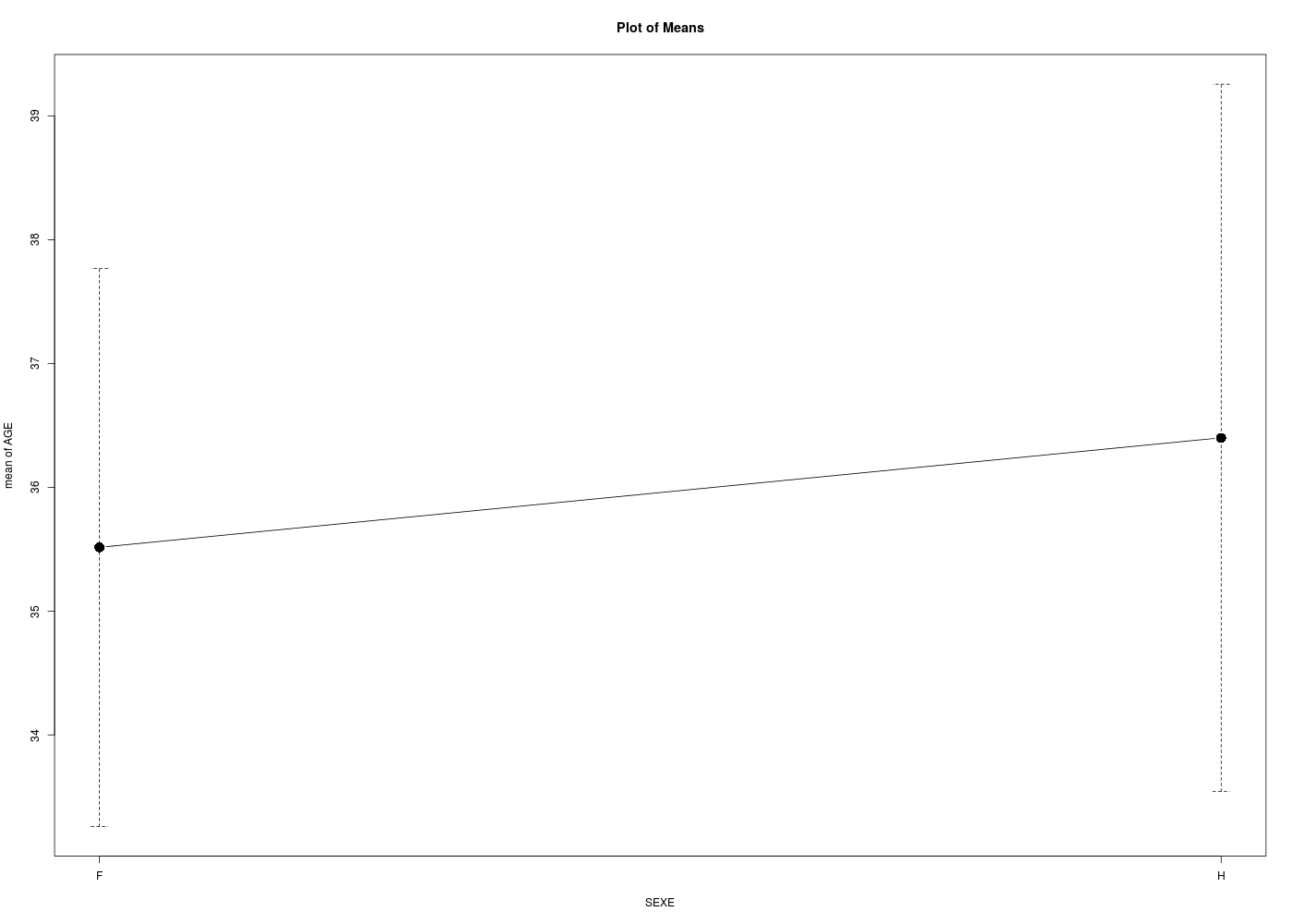
decritQTparFacteur("AGE dans ELF",elf$AGE,"ans","SEXE",as.numeric(elf$SEXE),"femme homme",TRUE)
VARIABLE QT AGE dans ELF ,unit=ans
VARIABLE QL SEXE labels : femme homme
N Moy Unite Ect Cdv Q1 Med Q3 EIQ Min Max
Global 99 35.828 ans 17.464 49 22.000 29.000 48.500 26.500 11.000 78.000
femme 64 35.516 ans 17.886 50 21.000 29.500 48.250 27.250 11.000 76.000
homme 35 36.400 ans 16.650 46 25.500 29.000 48.000 22.500 12.000 78.000
Analysis of Variance Table
Response: nomVarQT
Df Sum Sq Mean Sq F value Pr(>F)
ql 1 17.7 17.696 0.0569 0.812
Residuals 97 30176.4 311.097
ANOVA : there is no significant diffence at the level 0.05 for AGE dans ELF SEXE
Kruskal-Wallis rank sum test
data: nomVarQT by ql
Kruskal-Wallis chi-squared = 0.3016, df = 1, p-value = 0.5829
KRUSKAL-WALLIS : there is no significant diffence at the level 0.05 for AGE dans ELF SEXE
Avec Rstudio, pour les calculs, on peut utiliser le menu Statistiques, sous-menu Moyennes, sous-sous-menu ANOVA à un facteur.
Voici le code R produit :
library(survival, pos=17)
library(MASS, pos=17)
library(TH.data, pos=17)
library(multcomp, pos=17)
library(abind, pos=22)
AnovaModel.1 <- aov(AGE ~ SEXE, data=elf)
summary(AnovaModel.1)
with(elf, numSummary(AGE, groups=SEXE, statistics=c("mean", "sd")))
oneway.test(AGE ~ SEXE, data=elf) # Welch test
Les résultats sont :
Rcmdr> AnovaModel.1 <- aov(AGE ~ SEXE, data=elf)
Rcmdr> summary(AnovaModel.1)
Df Sum Sq Mean Sq F value Pr(>F)
SEXE 1 18 17.7 0.057 0.812
Residuals 97 30176 311.1
Rcmdr> with(elf, numSummary(AGE, groups=SEXE, statistics=c("mean", "sd")))
mean sd data:n
Femme 35.51562 18.02731 64
Homme 36.40000 16.89274 35
RcmdrMsg: [3] AVIS: Le facteur a moins de 3 niveaux ; Les comparaisons multiples
RcmdrMsg+ sont omises.
Rcmdr> oneway.test(AGE ~ SEXE, data=elf) # Welch test
One-way analysis of means (not assuming equal variances)
data: AGE and SEXE
F = 0.059112, num df = 1.00, denom df = 74.04, p-value = 0.8086
Pour les graphiques avec Rstudio, voir la réponse à l'exercice suivant.
En plus de calculer, il est souvent bon de représenter graphiquement les données, y compris par sous-groupe. Quelles sont les possibilités offertes par R et Rstudio dans ce domaine ?
Application : essayer de trouver des représentations adaptées et coloriées pour l'AGE en fonction du SEXE dans le dossier ELF ou pour les données RONFLE.
Rstudio sait tracer de nombreuses représentations par facteur, via le menu Graphes comme :
-
le tracé en point (dotplot) ;
-
l'histogramme ;
-
l'estimation de densité ;
-
le tracé en tige et feuilles dos à dos ;
-
la boite de dispersion (boite à moustaches) ;
-
le tracé des moyennes.
Voici le code R issu des clics dans ce menu
## tracés issus de library(Rcdmr)
## pour le dossier ELF nommé ici eo
with(eo, Dotplot(AGE, by=SEXE, bin=FALSE))
with(eo, Hist(AGE, groups=SEXE, scale="frequency", breaks="Sturges", col="darkgray"))
densityPlot(AGE~SEXE, data=eo, bw="SJ", adjust=1, kernel="gaussian")
library(tcltk, pos=16)
library(aplpack, pos=16)
with(eo, stem.leaf.backback(AGE[SEXE == "F"], AGE[SEXE == "H"], na.rm=TRUE))
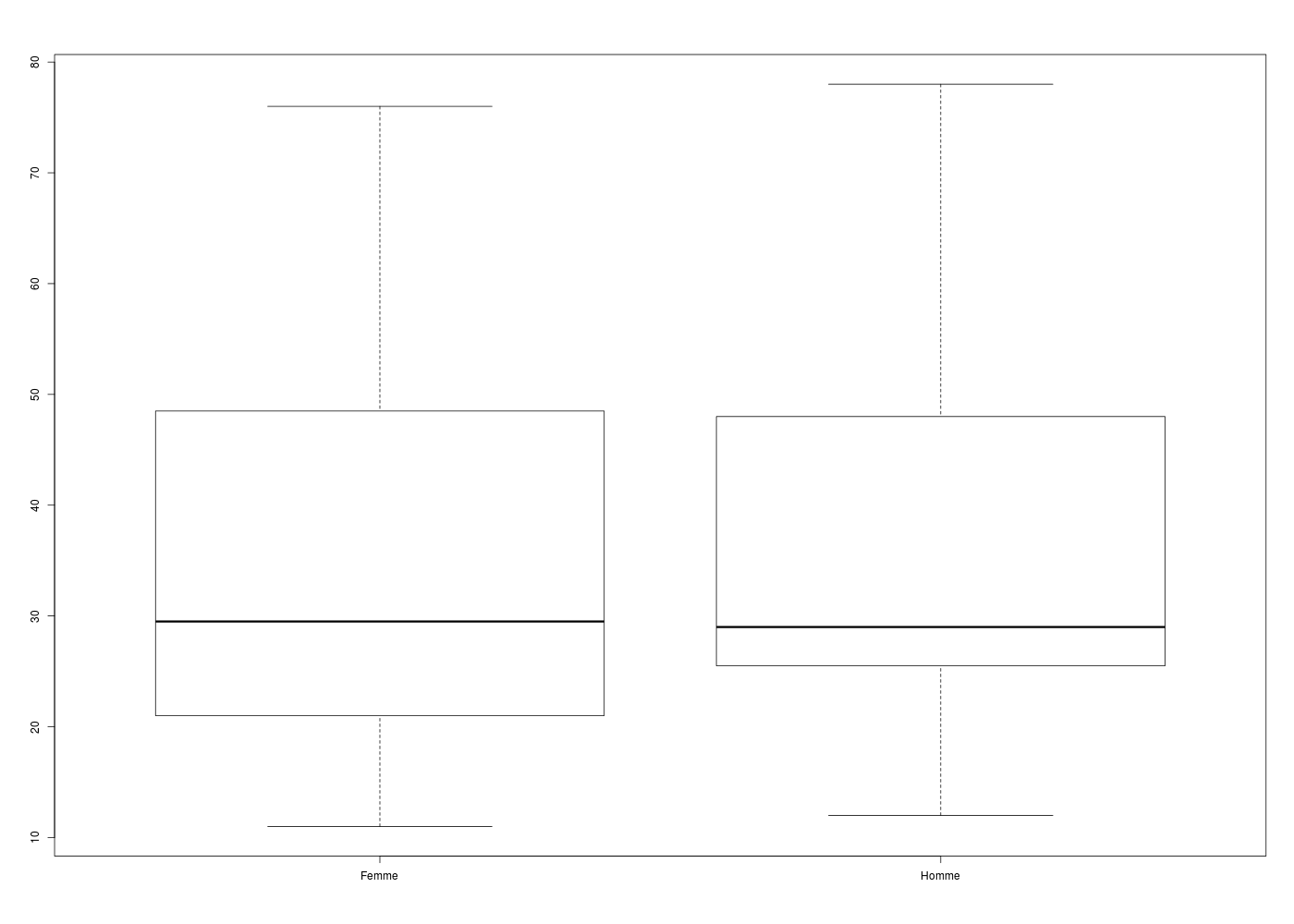
Boxplot(AGE~SEXE, data=eo, id.method="y")
with(eo, plotMeans(AGE, SEXE, error.bars="se", connect=TRUE))
Graphiques générés :
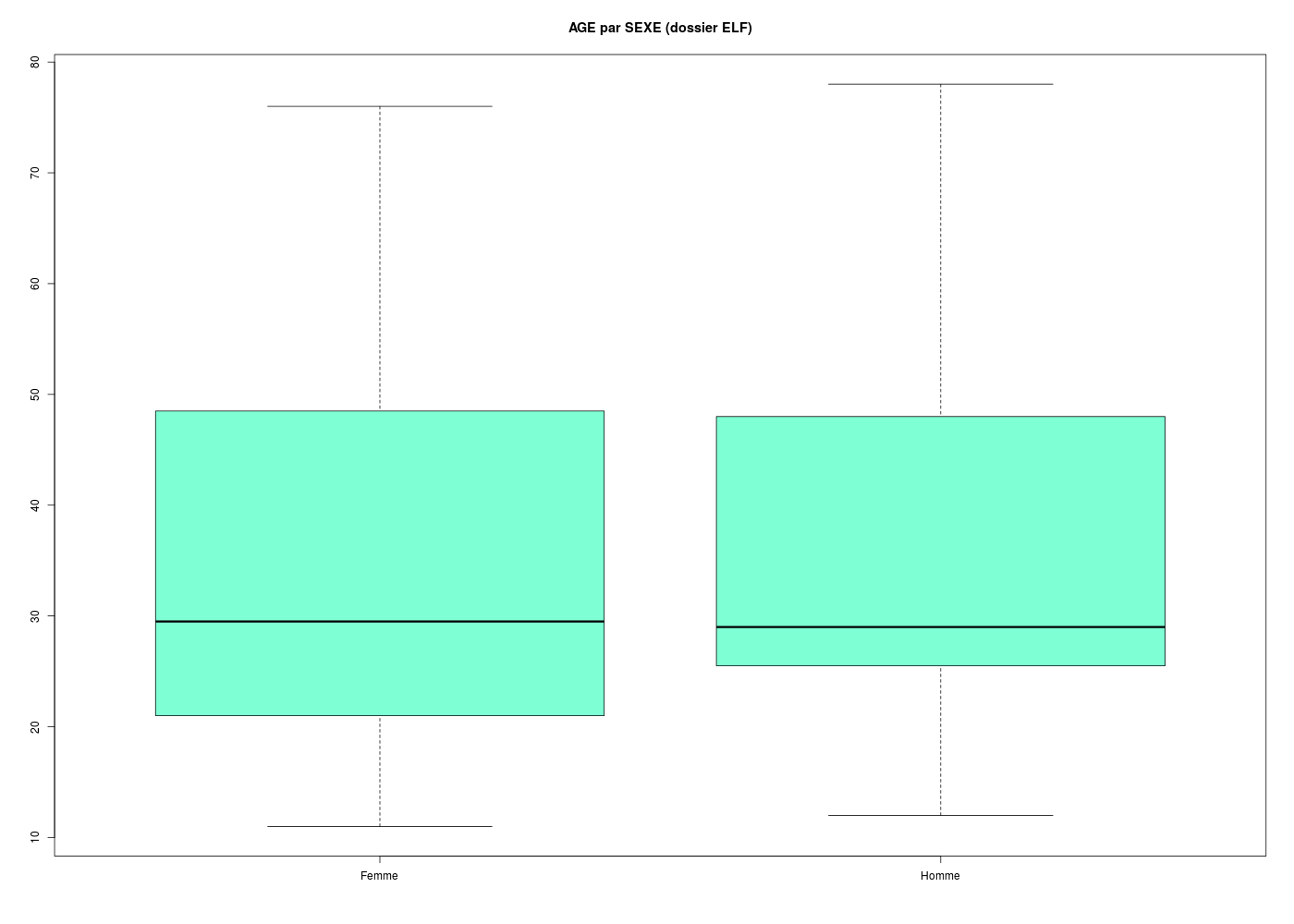
Mettre de la couleur avec R n'est pas spécialement difficile. Pour un certain nombre de fonctions, le paramètre col permet de spécifier la ou les couleurs.
## boxplots sans et avec couleurs
## pour les données AGE et SEXE du dossier ELF
# lecture des données
library("gdata")
elf <- read.xls("elfOrthoQL.xls")
levels(elf$SEXE) <- c("Femme","Homme")
# boxplot sans couleur ni titre
# (et noms explicites de variables maladroits)
boxplot(elf$AGE ~ elf$SEXE)
# boxplot avec titre et couleur
# via with() les noms de variables sont "propres"
with(elf,boxplot(AGE ~ SEXE,col="aquamarine",main="AGE par SEXE (dossier ELF)"))
Pour connaitre le noms des couleurs reconnues par R, on peut écrire demo("colors") mais le plus simple est de regarder ce que renvoie la fonction colors() du package grDevices.
> head(colors(),n=50)
[1] "white" "aliceblue" "antiquewhite" "antiquewhite1" "antiquewhite2"
[6] "antiquewhite3" "antiquewhite4" "aquamarine" "aquamarine1" "aquamarine2"
[11] "aquamarine3" "aquamarine4" "azure" "azure1" "azure2"
[16] "azure3" "azure4" "beige" "bisque" "bisque1"
[21] "bisque2" "bisque3" "bisque4" "black" "blanchedalmond"
[26] "blue" "blue1" "blue2" "blue3" "blue4"
[31] "blueviolet" "brown" "brown1" "brown2" "brown3"
[36] "brown4" "burlywood" "burlywood1" "burlywood2" "burlywood3"
[41] "burlywood4" "cadetblue" "cadetblue1" "cadetblue2" "cadetblue3"
[46] "cadetblue4" "chartreuse" "chartreuse1" "chartreuse2" "chartreuse3"
Pour tracer des points de couleur en fonction des modalités d'une variable qualitative, c'est souvent la fonction ifelse() qui est utilisée :
## tracé sans et avec couleurs
## pour les données AGE, PODS, SEXE, RONFLE et TABA du dossier RONFLE
# lecture des données sur internet
library("gdata")
url <- "http://forge.info.univ-angers.fr/~gh/wstat/Ortho/ronfle01.xls"
ronfle <- read.xls(url)
ronfle$SEXE <- ifelse(ronfle$SEXE==0,"Homme","Femme")
# transform() est plus agréable à utiliser pour ce
# type de recodage :
ronfle <- transform(ronfle,
SEXE =ifelse(SEXE==0,"Homme","Femme"),
RONFLE =ifelse(RONFLE==0,"non-ronfleur","ronfleur"),
TABA =ifelse(TABA==0,"non-fumeur","fumeur")
) # fin de transform
# tracé dans sans couleur ni titre
# (et noms explicites de variables maladroits)
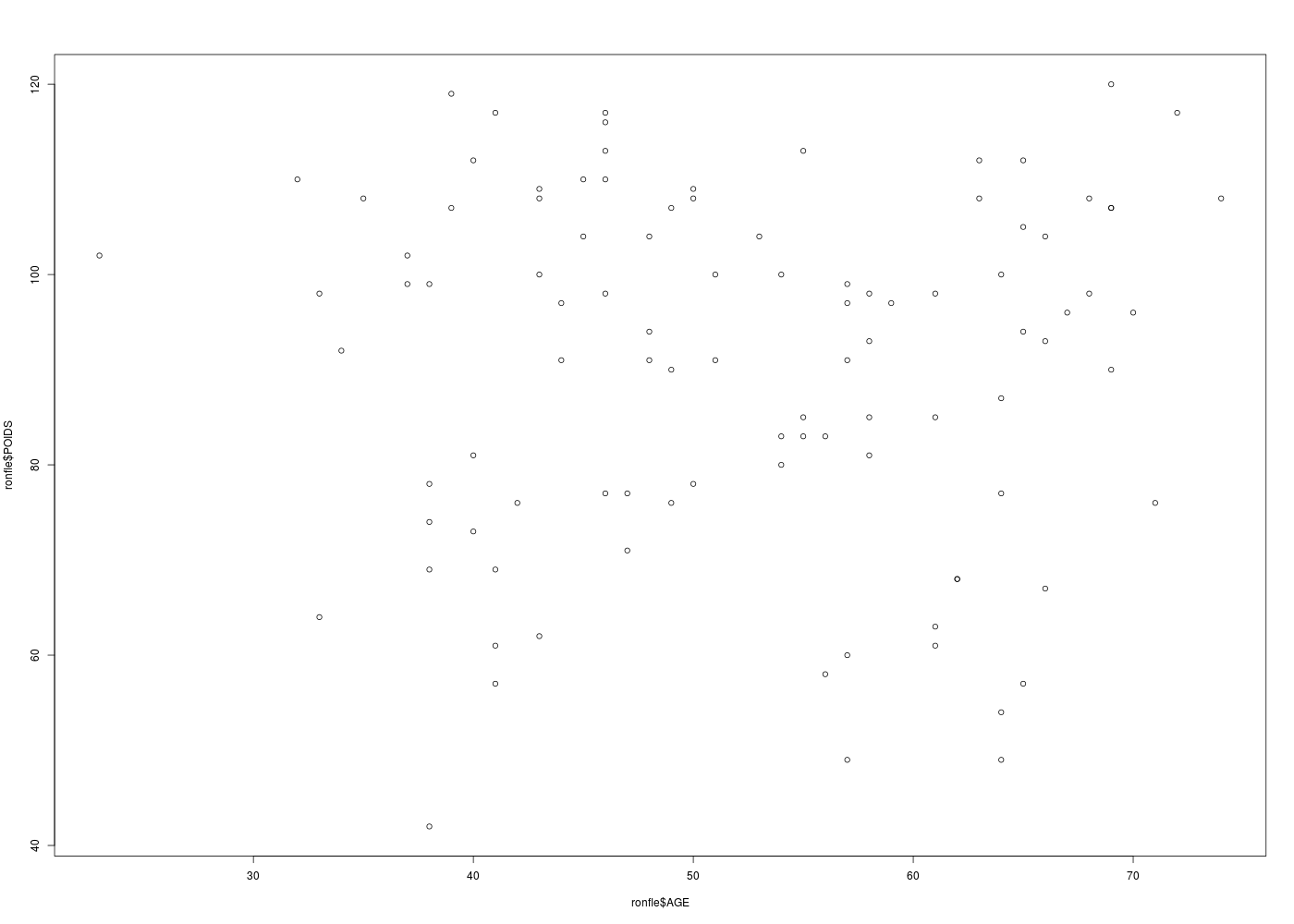
plot(ronfle$POIDS ~ ronfle$AGE)
# tracés avec titre et couleur
# via with() les noms de variables sont "propres"
colSexe <- ifelse(ronfle$SEXE=="Homme","blue","red")
colRonfle <- ifelse(ronfle$RONFLE=="non-ronfleur","blue","red")
colTaba <- ifelse(ronfle$TABA=="non-fumeur","blue","red")
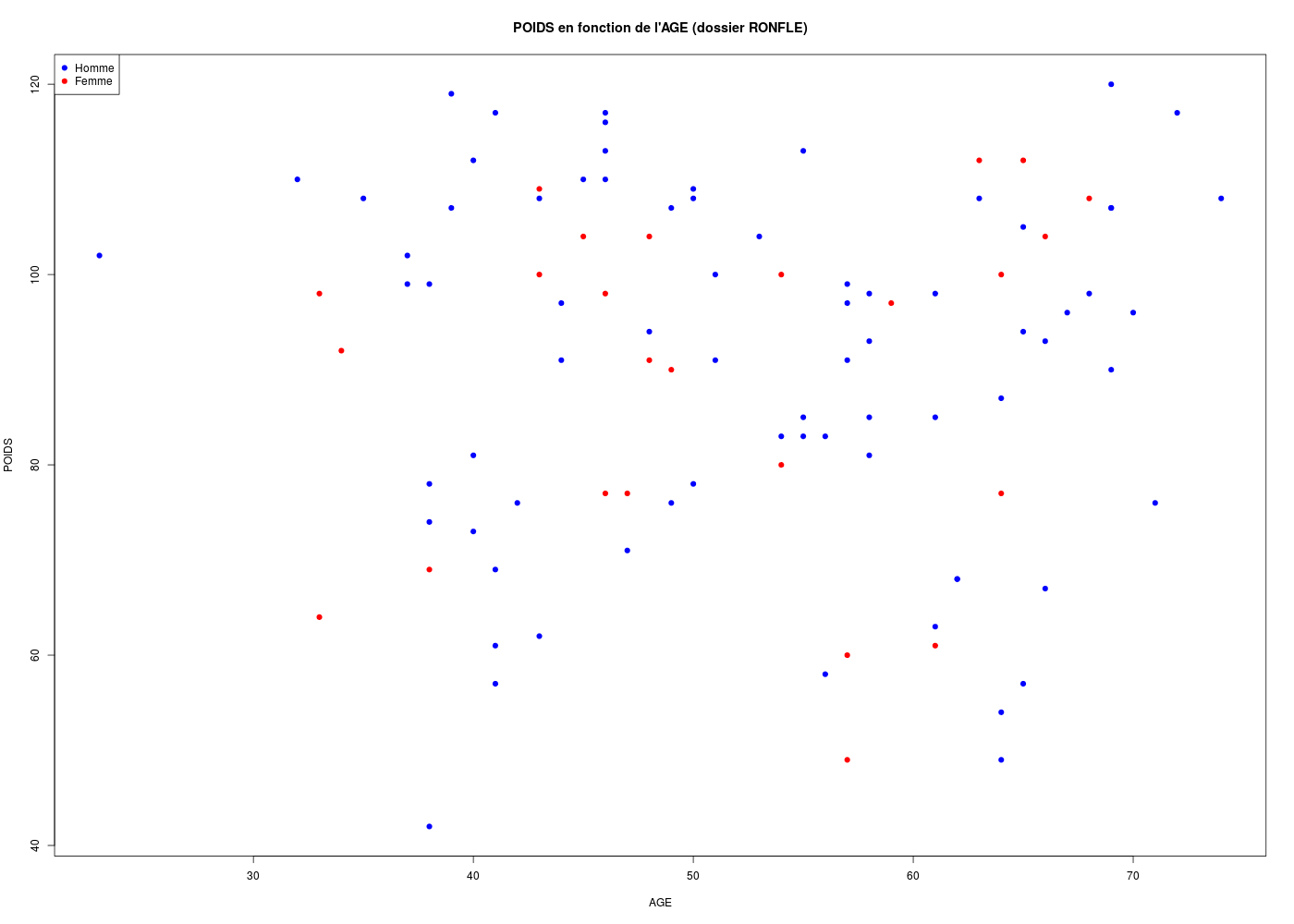
with(ronfle,plot(POIDS ~ AGE,col=colSexe,main="POIDS en fonction de l'AGE (dossier RONFLE)",pch=19))
legend(
x="topleft",
legend=c("Homme","Femme"),
col=c("blue","red"),
pch=19
) # fin de légende
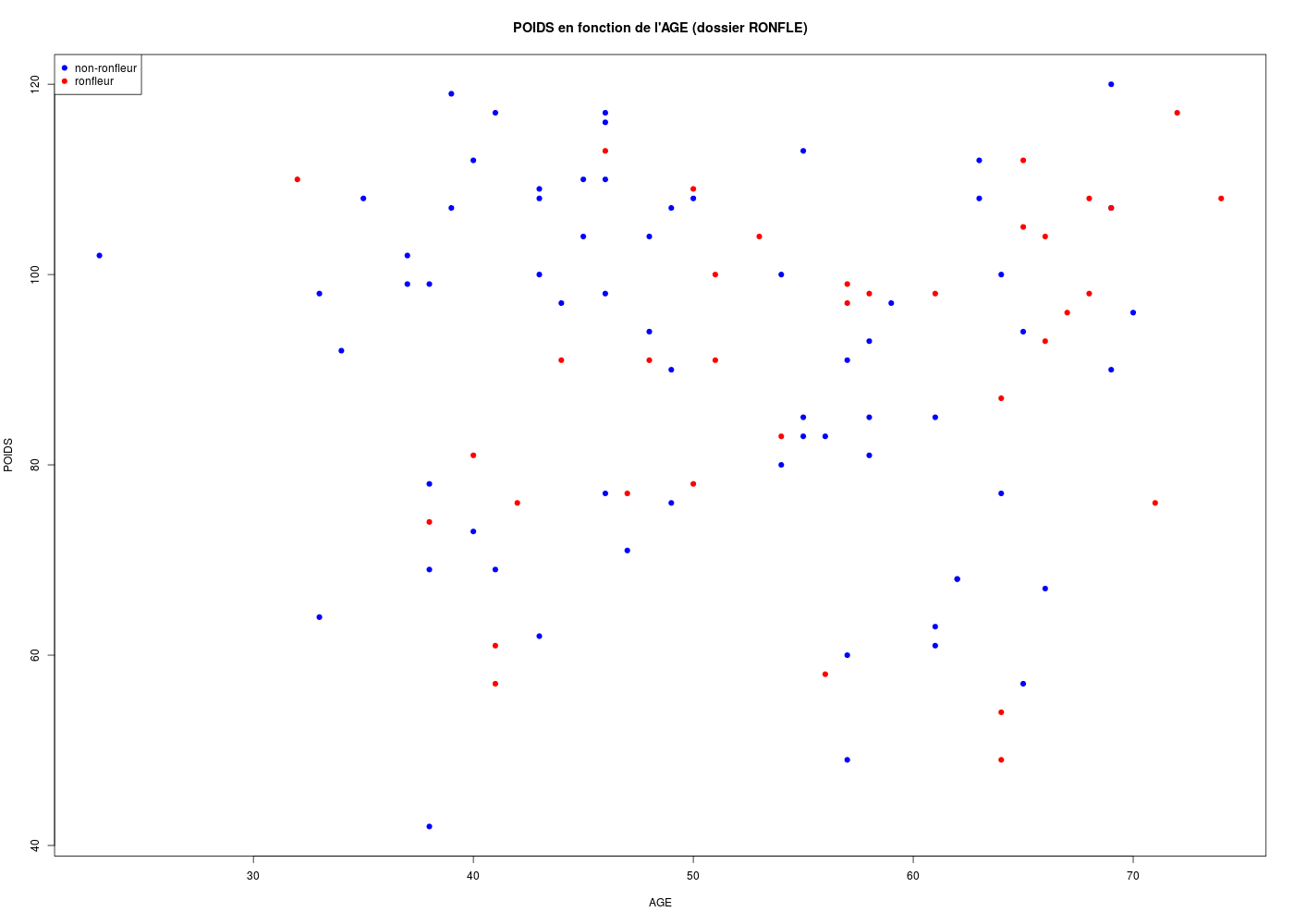
with(ronfle,plot(POIDS ~ AGE,col=colRonfle,main="POIDS en fonction de l'AGE (dossier RONFLE)",pch=19))
legend(
x="topleft",
legend=c("non-ronfleur","ronfleur"),
col=c("blue","red"),
pch=19
) # fin de légende
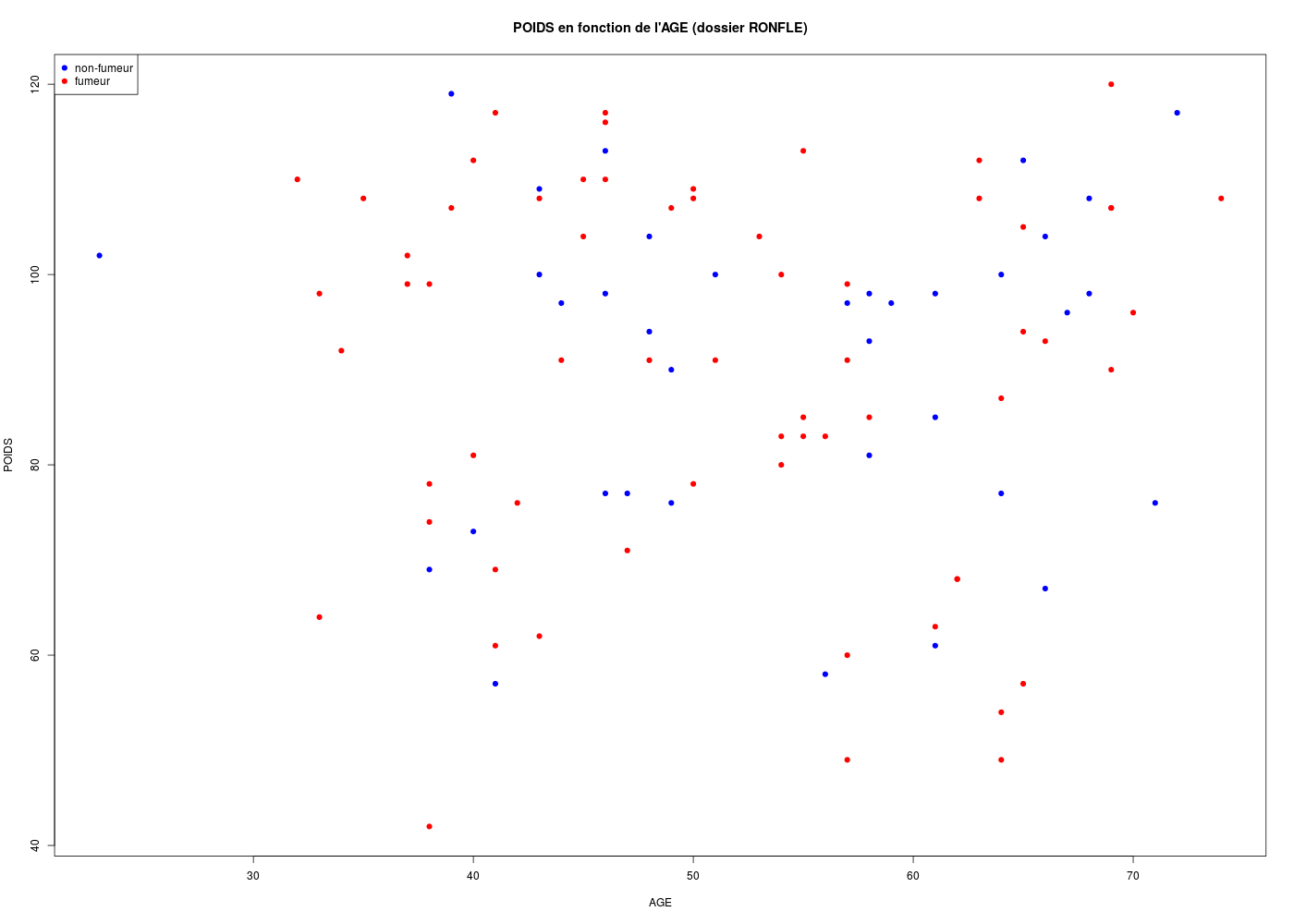
with(ronfle,plot(POIDS ~ AGE,col=colTaba,main="POIDS en fonction de l'AGE (dossier RONFLE)",pch=19))
legend(
x="topleft",
legend=c("non-fumeur","fumeur"),
col=c("blue","red"),
pch=19
) # fin de légende
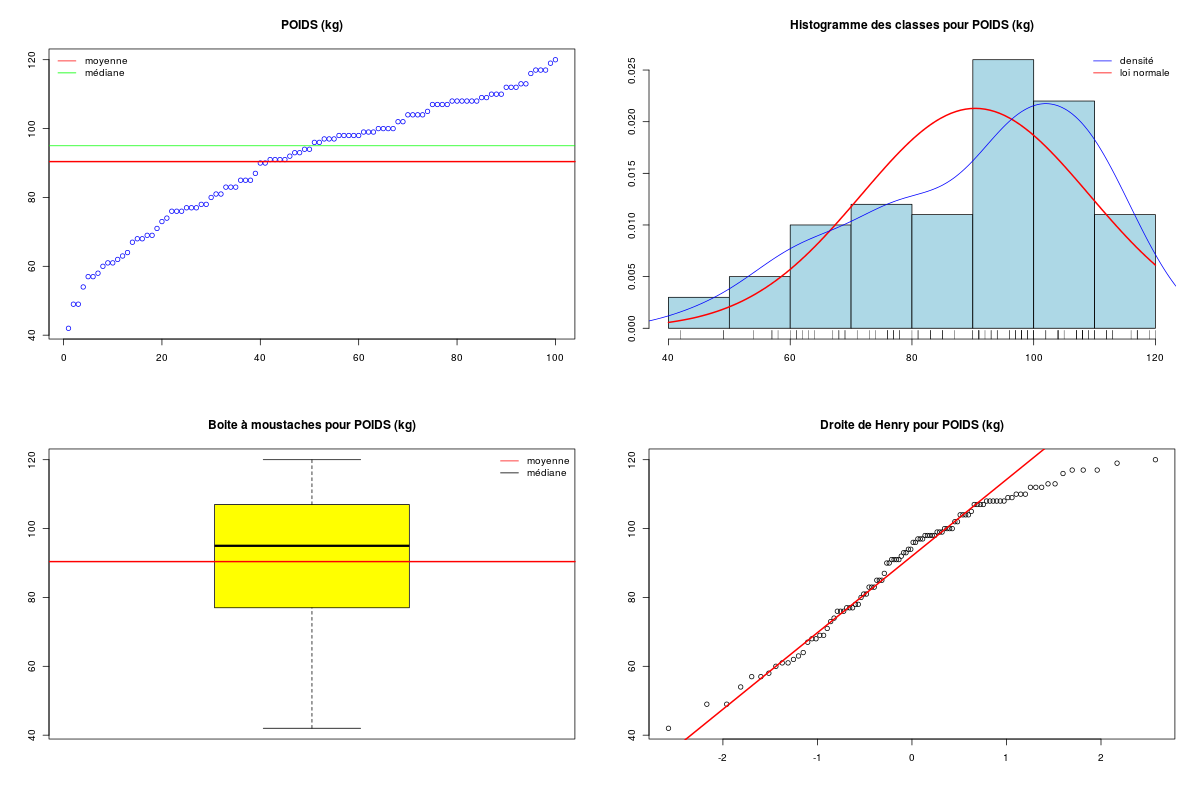
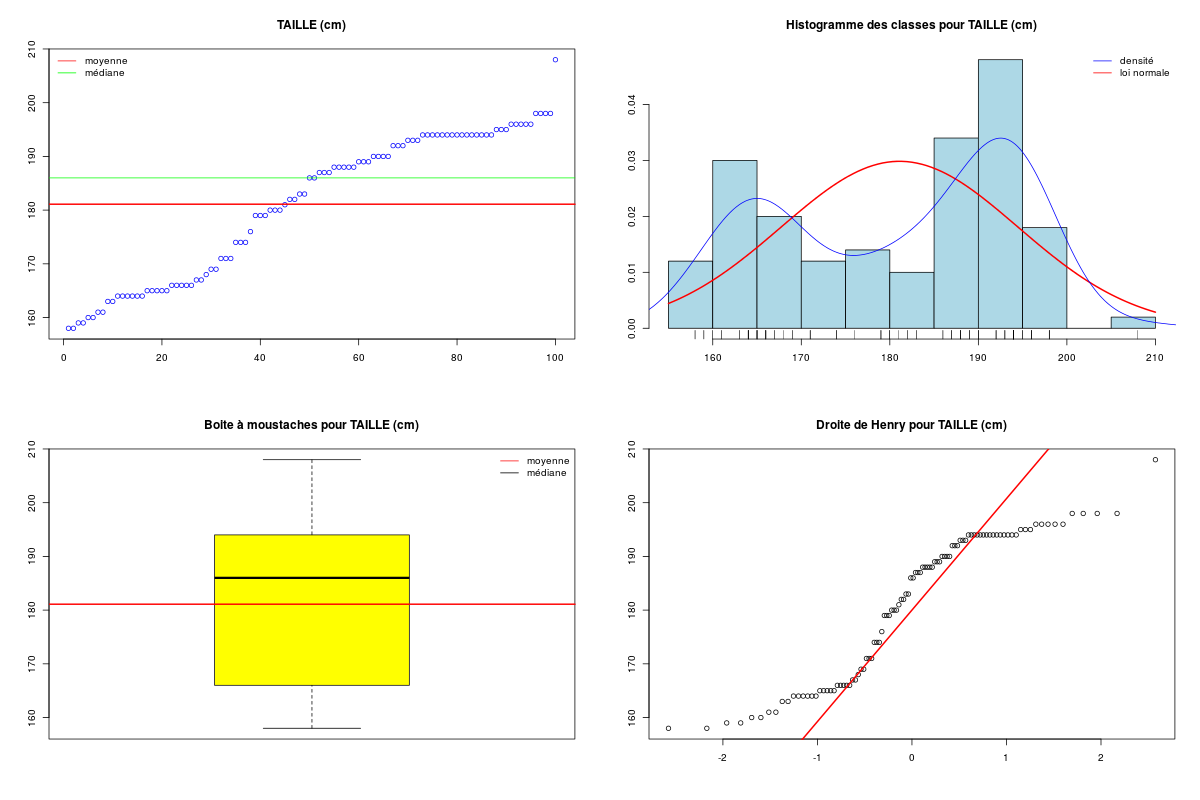
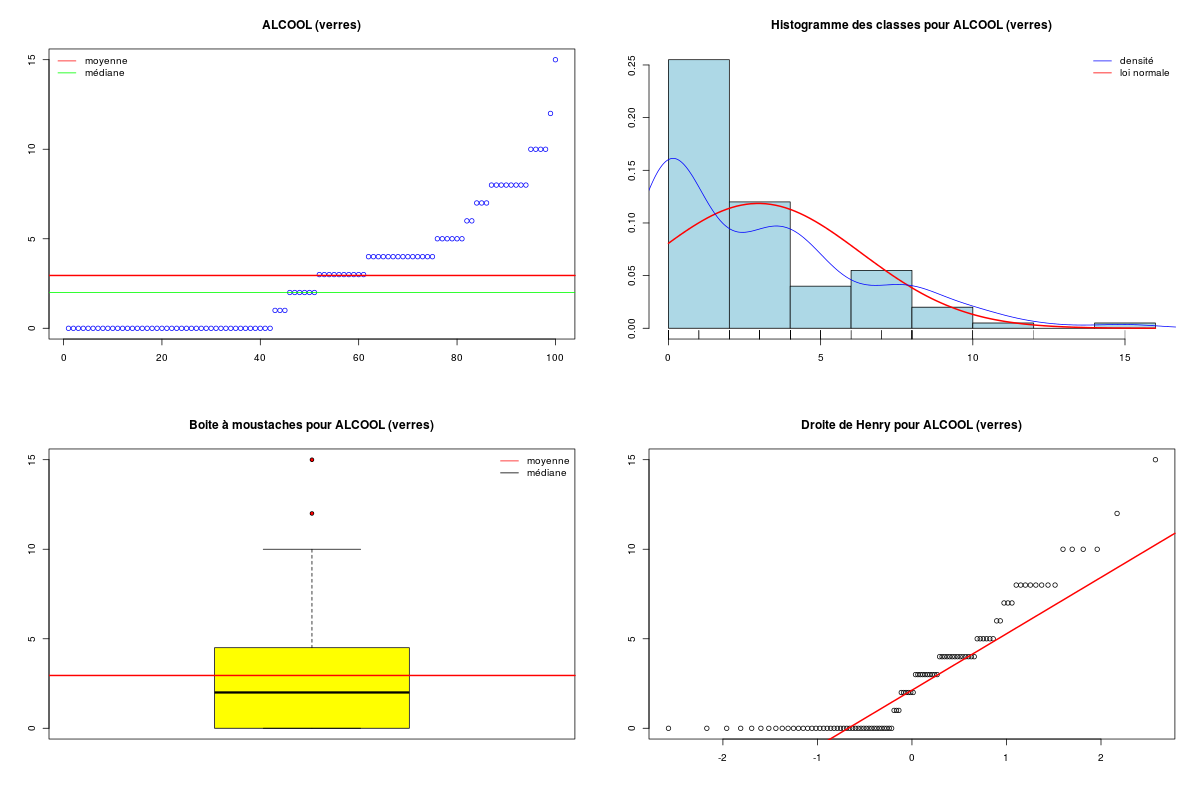
Décrire conjointement les variables QT du dossier RONFLE.
Une fois les données QT sélectionnées, toutes les analyses peuvent être réalisées avec notre fonction allQTdf. Le code R à saisir est donc très court mais il peut produire plusieurs centaines de lignes de résultats et de nombreux graphiques.
## Analyse conjointe et régression linéaire
## pour les données (QT) AGE, POIDS, TAILLE, ALCOOL du dossier RONFLE
# lecture des données sur internet
library("gdata")
url <- "http://forge.info.univ-angers.fr/~gh/wstat/Ortho/ronfle01.xls"
ronfle <- read.xls(url)
# on se restreint aux QT
ronfleQT <- ronfle[,(2:5)] # dans cet ordre : AGE, POIDS, TAILLE, ALCOOL
unites <- c("ans","kg","cm","verres")
allQTdf(ronfleQT,nomU=unites,details=TRUE,univar=TRUE)
Voici les 10 premières lignes de données (il y en a 100 en tout)
AGE POIDS TAILLE ALCOOL
1 47 71 158 0
2 56 58 164 7
3 46 116 208 3
4 70 96 186 3
5 51 91 195 2
6 46 98 188 0
7 40 112 193 5
8 46 77 165 0
9 49 76 164 0
10 39 119 196 3
Description des 4 variables statistiques par cdv décroissant
Num Nom Taille Moyenne Unite Ecart-type Coef. de var. Minimum Maximum
4 ALCOOL 100 2.950 verres 3.365 114.06 % 0.000 15.000
1 AGE 100 52.270 ans 11.397 21.80 % 23.000 74.000
2 POIDS 100 90.410 kg 18.736 20.72 % 42.000 120.000
3 TAILLE 100 181.100 cm 13.366 7.38 % 158.000 208.000
Par ordre d'entrée
Num Nom Taille Moyenne Unite Ecart-type Coef. de var. Minimum Maximum
1 AGE 100 52.270 ans 11.397 21.80 % 23.000 74.000
2 POIDS 100 90.410 kg 18.736 20.72 % 42.000 120.000
3 TAILLE 100 181.100 cm 13.366 7.38 % 158.000 208.000
4 ALCOOL 100 2.950 verres 3.365 114.06 % 0.000 15.000
Par moyenne décroissante
Num Nom Taille Moyenne Unite Ecart-type Coef. de var. Minimum Maximum
3 TAILLE 100 181.100 cm 13.366 7.38 % 158.000 208.000
2 POIDS 100 90.410 kg 18.736 20.72 % 42.000 120.000
1 AGE 100 52.270 ans 11.397 21.80 % 23.000 74.000
4 ALCOOL 100 2.950 verres 3.365 114.06 % 0.000 15.000
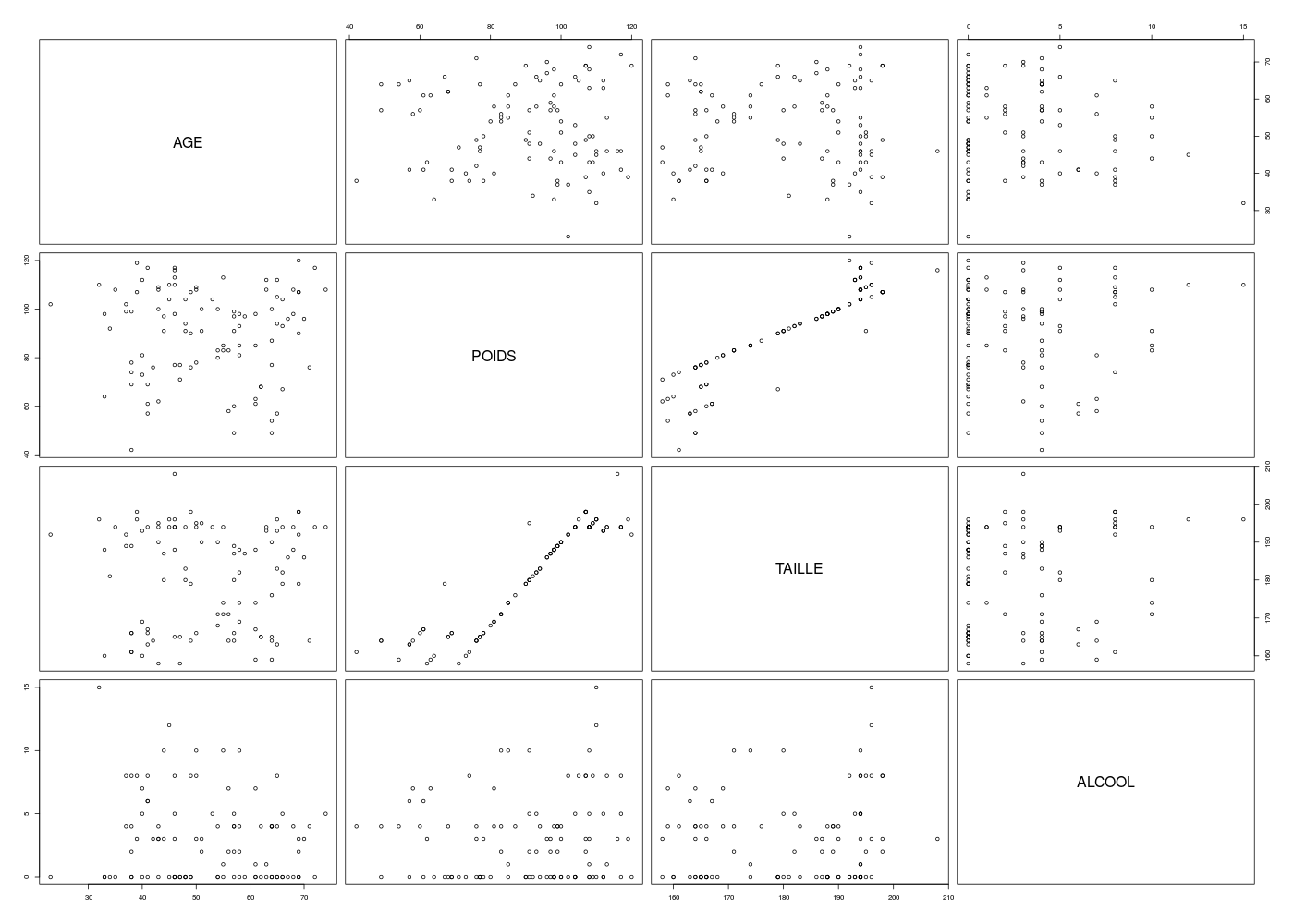
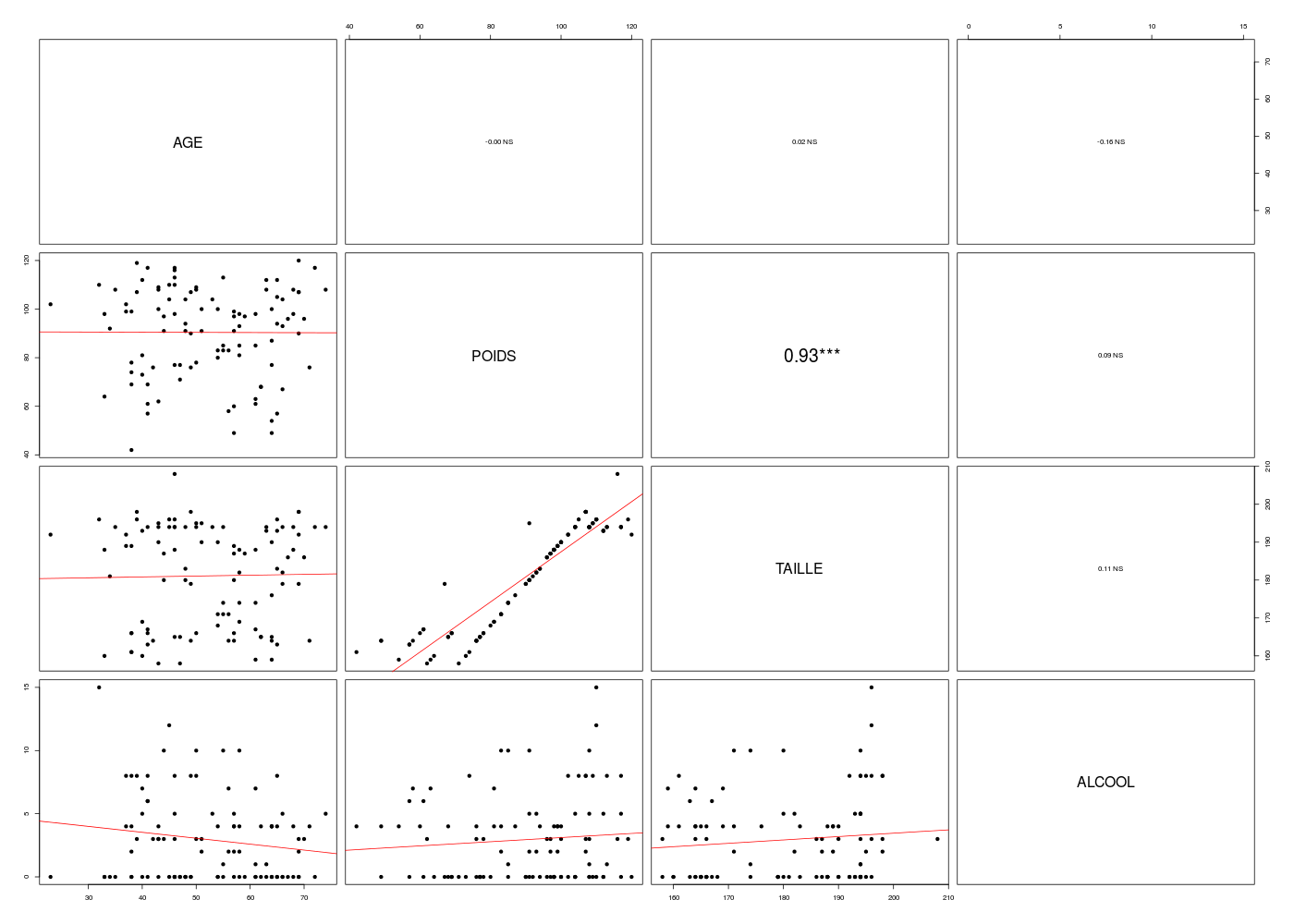
Matrice des corrélations au sens de Pearson pour 100 lignes et 4 colonnes
AGE POIDS TAILLE ALCOOL
AGE 1.000
POIDS -0.004 1.000
TAILLE 0.019 0.927 1.000
ALCOOL -0.159 0.092 0.106 1.000
Meilleure corrélation 0.9269744 pour TAILLE et POIDS p-value 1.61e-43
Formules POIDS = 1.299 * TAILLE -144.905
et TAILLE = 0.661 * POIDS + 121.311
Coefficients de corrélation par ordre décroissant
0.927 p-value 0.0000 pour TAILLE et POIDS : TAILLE = 0.661 x POIDS + 121.311
-0.159 p-value 0.1147 pour ALCOOL et AGE : ALCOOL = -0.047 x AGE + 5.400
0.106 p-value 0.2923 pour ALCOOL et TAILLE : ALCOOL = 0.027 x TAILLE - 1.898
0.092 p-value 0.3652 pour ALCOOL et POIDS : ALCOOL = 0.016 x POIDS + 1.464
0.019 p-value 0.8482 pour TAILLE et AGE : TAILLE = 0.023 x AGE + 179.912
-0.004 p-value 0.9687 pour POIDS et AGE : POIDS = -0.007 x AGE + 90.752
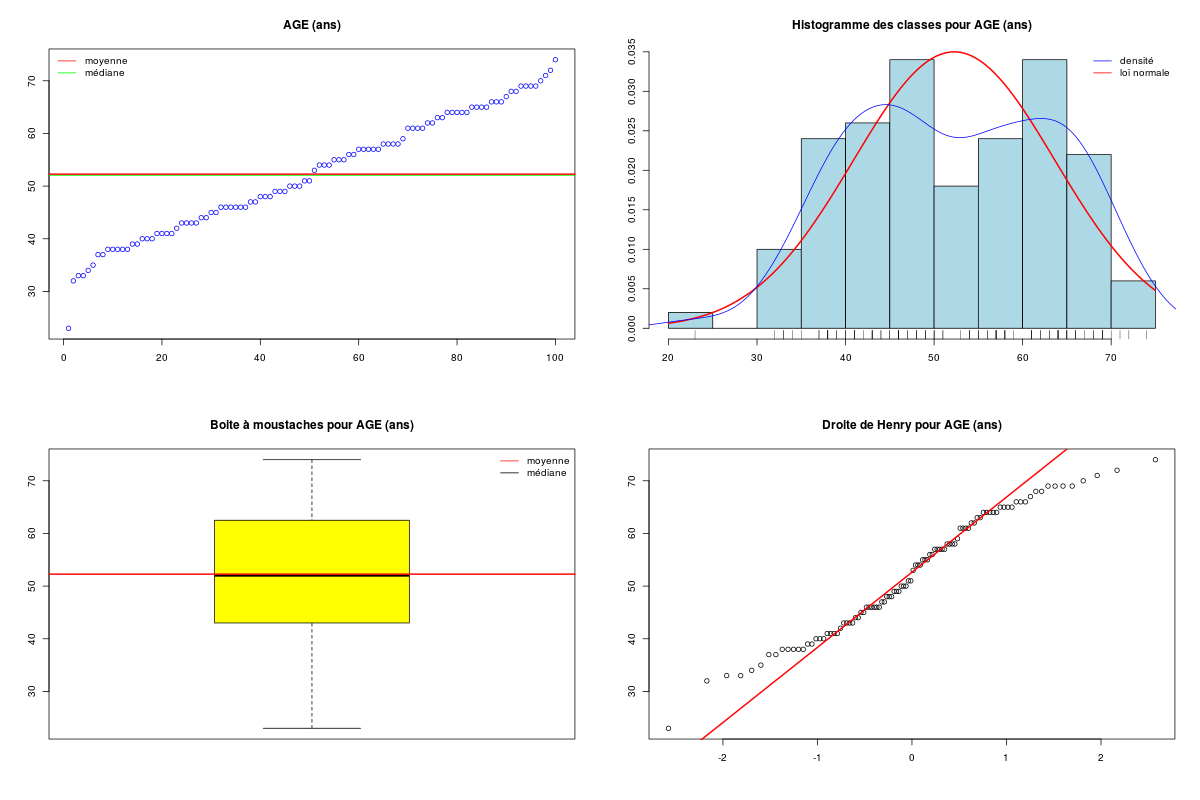
DESCRIPTION STATISTIQUE DE LA VARIABLE AGE
Taille 100 individus
Moyenne 52.2700 ans
Ecart-type 11.3972 ans
Coef. de variation 22 %
1er Quartile 43.0000 ans
Mediane 52.0000 ans
3eme Quartile 62.2500 ans
iqr absolu 19.2500 ans
iqr relatif 37.0000 %
Minimum 23.000 ans
Maximum 74.000 ans
Tracé tige et feuilles
The decimal point is 1 digit(s) to the right of the |
2 | 3
3 | 23345778888899
4 | 000111123333445566666677888999
5 | 000113444555667777788889
6 | 111122334444455556667889999
7 | 0124
DESCRIPTION STATISTIQUE DE LA VARIABLE POIDS
Taille 100 individus
Moyenne 90.4100 kg
Ecart-type 18.7359 kg
Coef. de variation 21 %
1er Quartile 77.0000 kg
Mediane 95.0000 kg
3eme Quartile 107.0000 kg
iqr absolu 30.0000 kg
iqr relatif 32.0000 %
Minimum 42.000 kg
Maximum 120.000 kg
Tracé tige et feuilles
The decimal point is 1 digit(s) to the right of the |
4 | 299
5 | 4778
6 | 01123478899
7 | 13466677788
8 | 0113335557
9 | 001111233446677788888999
10 | 00002244445777788888899
11 | 0002223367779
12 | 0
DESCRIPTION STATISTIQUE DE LA VARIABLE TAILLE
Taille 100 individus
Moyenne 181.1000 cm
Ecart-type 13.3662 cm
Coef. de variation 7 %
1er Quartile 166.0000 cm
Mediane 186.0000 cm
3eme Quartile 194.0000 cm
iqr absolu 28.0000 cm
iqr relatif 15.0000 %
Minimum 158.000 cm
Maximum 208.000 cm
Tracé tige et feuilles
The decimal point is 1 digit(s) to the right of the |
15 | 8899
16 | 001133444444555556666677899
17 | 1114446999
18 | 000122336677788888999
19 | 0000222333444444444444444555666668888
20 | 8
DESCRIPTION STATISTIQUE DE LA VARIABLE ALCOOL
Taille 100 individus
Moyenne 2.9500 verres
Ecart-type 3.3646 verres
Coef. de variation 113 %
1er Quartile 0.0000 verres
Mediane 2.0000 verres
3eme Quartile 4.2500 verres
iqr absolu 4.2500 verres
iqr relatif 212.0000 %
Minimum 0.000 verres
Maximum 15.000 verres
Tracé tige et feuilles
The decimal point is at the |
0 | 000000000000000000000000000000000000000000000
2 | 0000000000000000
4 | 00000000000000000000
6 | 00000
8 | 00000000
10 | 0000
12 | 0
14 | 0
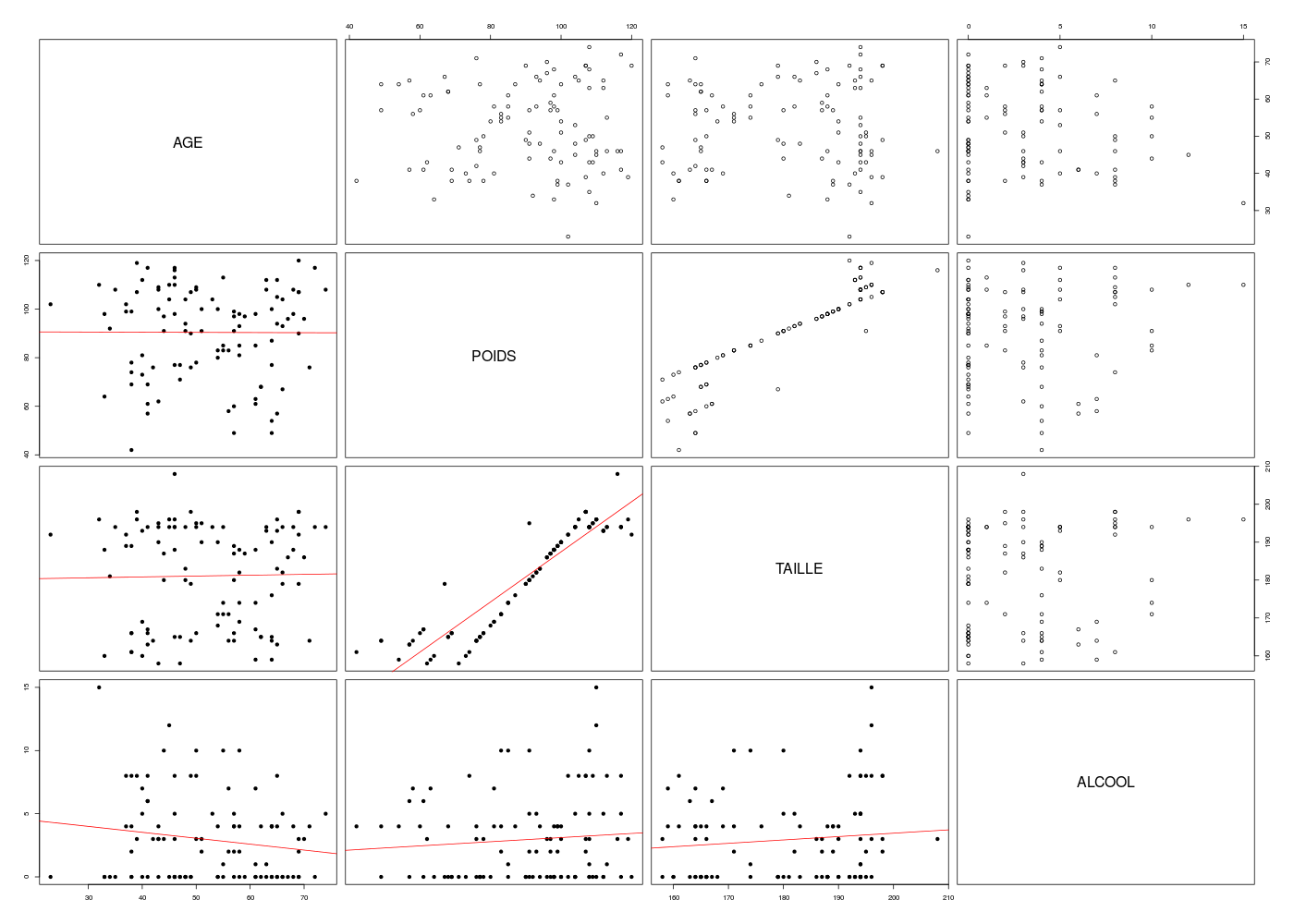
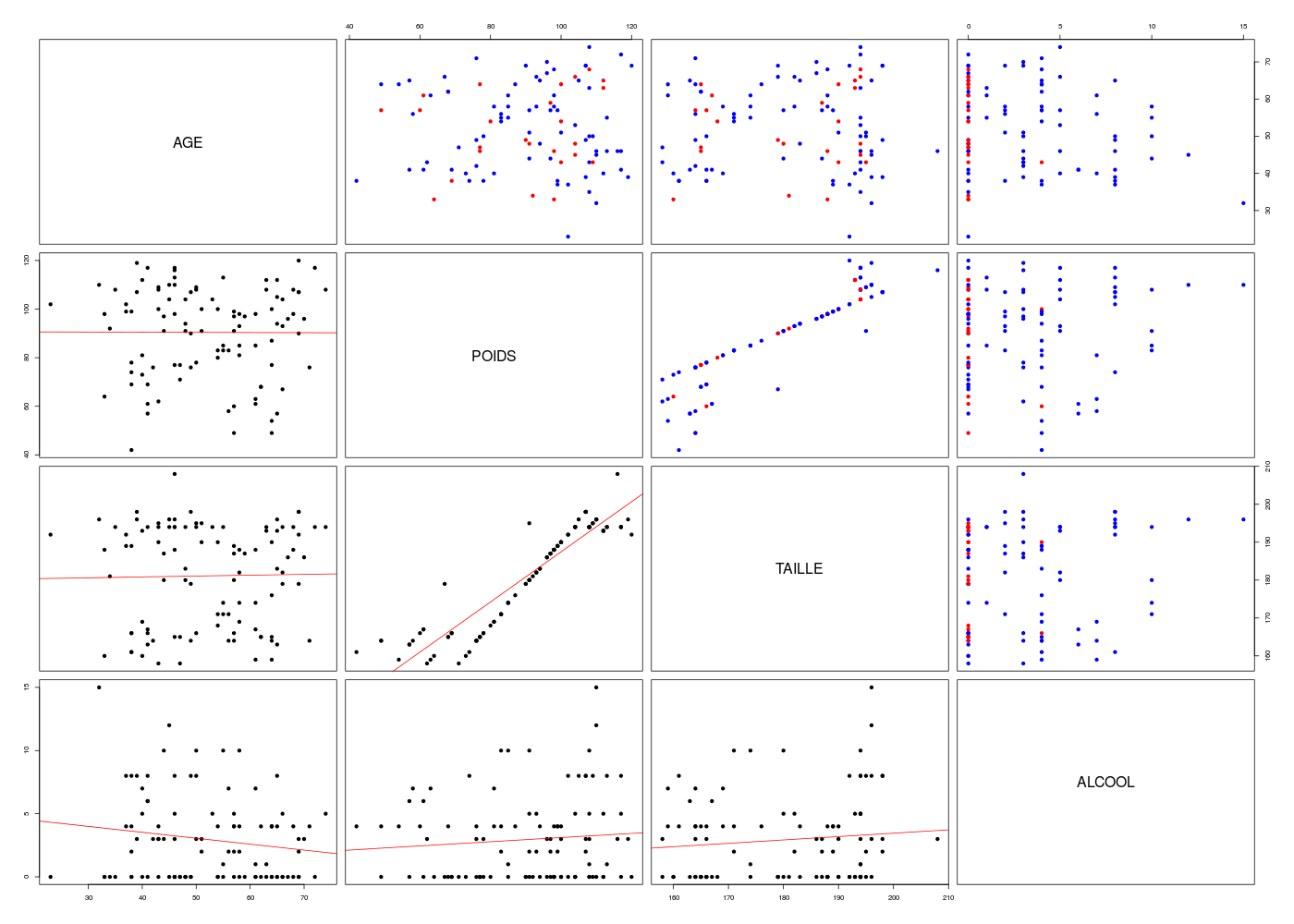
Pour bien décrire statistiquement les corrélations possibles au niveau de ces données QT, un simple pairs(ronfleQT) n'est sans doute pas suffisant. Avec notre fonction pairsi, les tendances sont plus visibles et on peut même voir les sous-groupes :
## tracé par paires des données QT du dossier RONFLE
# tracé standard de R
pairs(ronfleQT)
# notre tracé amélioré
pairsi(ronfleQT,opt=1)
pairsi(ronfleQT,opt=2)
couleurs <- ifelse(ronfle$SEXE==0,"blue","red")
pairsi(ronfleQT,opt=1,col=couleurs,pch=19)
Une fois tous ces calculs et graphiques tracés, la «vraie» analyse statistique peut commencer...
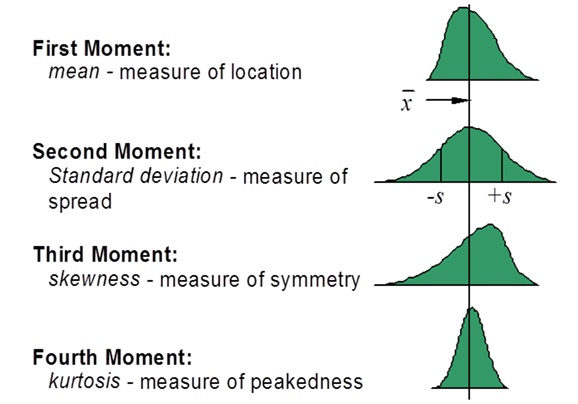
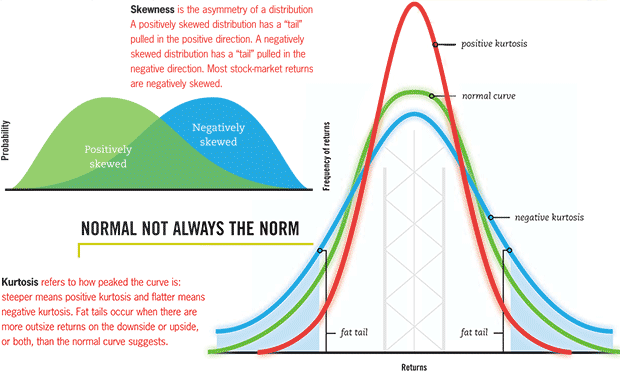
Lorsqu'on décrit numériquement une variable quantitative, certains auteurs utilisent les deux indicateurs numériques skewness et kurtosis. Comment les calcule-t-on et à quoi servent-t-ils ?
Ces termes techniques sont des indicateurs descriptifs de comparaison à la loi normale, définis par les formules suivantes :
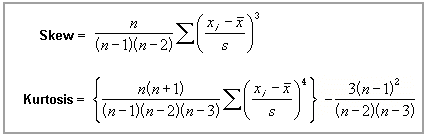
On leur associe des qualificatifs comme leptokurtique, mésokurtique, platikurtique... Chaque terme a une entrée wiki : skewness ; kurtosis. En France, on parle volontiers de coefficient de dissymétrie au lieu de skewness et de coefficient d'aplatissement au lieu de kurtosis.
retour au plan de cours
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)