XML en M1 (2018), université d'Angers
T.P. numéro 2
Table des matières cliquable
1. Compter des éléments ou des attributs dans un document XML
2. Expressions XPATH
3. Comprendre la transformation vide
4. Réaliser une transformation minimale avec XSL
5. Une transformation XSL élémentaire
6. Une deuxième transformation XSL
7. Transformations XSL avancées et statistiques
Il est possible d'afficher toutes les solutions via ?solutions=1 et de les masquer via ?solutions=0 .
On s'intéresse au fichier leadb880.xml suivant qui contient des données pour des protéines LEA :
Après avoir décrit la structure du fichier, donner une grammaire DTD minimale et raisonnable pour modéliser le contenu du fichier, puis compter le nombre de lignes, le nombre de lignes vides, d'élements et d'attributs. Vous pouvez utiliser tout outil logiciel qui vous parait adapté. Comment trouver le nombre d'éléments distincts ?
Visiblement l'élément racine du fichier se nomme proteins et il ne contient que des éléments nommés protein. Chaque élément protein contient trois éléments, à savoir accession, class et fasta. De plus seul cet élément fasta possède un attribut nommé length.
Une grammaire raisonnable pour ces données est fournie par le fichier suivant nommé leadb.dtd.
Grammaire leadb.dtd :
<?xml encoding="UTF-8"?>
<!ELEMENT proteins (protein)+ >
<!ELEMENT protein (accession,class,fasta) >
<!ELEMENT accession (#PCDATA) >
<!ELEMENT class (#PCDATA) >
<!ELEMENT fasta (#PCDATA) >
<!ATTLIST fasta xmlns CDATA #FIXED '' length CDATA #REQUIRED>
Avec la commande xmlstarlet el -u on dispose de la structure des données en termes d'éléments :
xmlstarlet el -u leadb880.xml
proteins
proteins/protein
proteins/protein/accession
proteins/protein/class
proteins/protein/fasta
Il ne manque donc que la ligne proteins/protein/fasta/@length pour compléter cette description des éléments et attributs.
On peut facilement obtenir le nombre de lignes avec la commande système nommée wc. Pour le nombre de lignes vides, chainer grep et wc est sans doute un bon choix. Pour le nombre d'élements distincts et la structure, xmlstarlet et trang sont des outils biens adaptés. Enfin, pour le nombre d'éléments en tout, on peut chainer xmlstarlet et wc alors que pour le nombre d'attributs en tout, il faut chainer xmlstarlet, grep et wc. Voici le détail des commandes associées :
wc -l leadb880.xml # nombre de lignes (6163)
grep "^$" leadb880.xml | wc -l # nombre de lignes vides (881)
xmlstarlet el leadb880.xml | wc -l # nombre d'éléments (3521)
xmlstarlet el -u leadb880.xml | wc -l # nombre d'éléments distincts (5)
xmlstarlet el -a leadb880.xml | wc -l # nombre d'éléments ou attributs (4401)
xmlstarlet el -a leadb880.xml | grep "/@" | wc -l # nombre d'attributs (880)
xmlstarlet el -a leadb880.xml | grep "@" | wc -l # idem
(trang leadb880.xml leadb_tmp.dtd ; cat leadb_tmp.dtd) # production de la grammaire dtd
(trang leadb880.xml leadb_tmp.xsd ; cat leadb_tmp.xsd) # production de la grammaire xsd
On s'intéresse ici au document films2.xml qui contient des films et des artistes.
En voici la grammaire DTD :
<!ELEMENT FILMSETARTISTES (FILMS,ARTISTES)>
<!ELEMENT FILMS (FILM)+>
<!ELEMENT FILM (TITRE,GENRE,PAYS,MES,ROLES,RESUME?) >
<!ELEMENT TITRE (#PCDATA) >
<!ELEMENT GENRE (#PCDATA) >
<!ELEMENT PAYS (#PCDATA) >
<!ELEMENT MES EMPTY >
<!ELEMENT ROLES (ROLE)* >
<!ELEMENT RESUME (#PCDATA) >
<!ELEMENT ROLE (PRENOM,NOM,INTITULE) >
<!ELEMENT PRENOM (#PCDATA) >
<!ELEMENT INTITULE (#PCDATA) >
<!ELEMENT NOM (#PCDATA) >
<!ELEMENT ARTISTES (ARTISTE)+ >
<!ELEMENT ARTISTE (ARTNOM,ARTPRENOM,ANNEENAISS?) >
<!ELEMENT ARTNOM (#PCDATA) >
<!ELEMENT ARTPRENOM (#PCDATA) >
<!ELEMENT ANNEENAISS (#PCDATA) >
<!ATTLIST ARTISTE xmlns CDATA #FIXED '' id CDATA #REQUIRED >
<!ATTLIST FILM xmlns CDATA #FIXED '' Annee CDATA #REQUIRED >
<!ATTLIST MES xmlns CDATA #FIXED '' idref CDATA #REQUIRED >
On fournit aussi la structure explicite du fichier (éléments seulement) obtenue à l'aide de la commande xmlstarlet el -u films2.xml :
FILMSETARTISTES
FILMSETARTISTES/ARTISTES
FILMSETARTISTES/ARTISTES/ARTISTE
FILMSETARTISTES/ARTISTES/ARTISTE/ANNEENAISS
FILMSETARTISTES/ARTISTES/ARTISTE/ARTNOM
FILMSETARTISTES/ARTISTES/ARTISTE/ARTPRENOM
FILMSETARTISTES/FILMS
FILMSETARTISTES/FILMS/FILM
FILMSETARTISTES/FILMS/FILM/GENRE
FILMSETARTISTES/FILMS/FILM/MES
FILMSETARTISTES/FILMS/FILM/PAYS
FILMSETARTISTES/FILMS/FILM/RESUME
FILMSETARTISTES/FILMS/FILM/ROLES
FILMSETARTISTES/FILMS/FILM/ROLES/ROLE
FILMSETARTISTES/FILMS/FILM/ROLES/ROLE/INTITULE
FILMSETARTISTES/FILMS/FILM/ROLES/ROLE/NOM
FILMSETARTISTES/FILMS/FILM/ROLES/ROLE/PRENOM
FILMSETARTISTES/FILMS/FILM/TITRE
Trouver comment on peut produire la liste des attributs uniques du fichier, ce que ne sait pas faire xmlstarlet. Voici ce qu'on s'attend à voir :
FILMSETARTISTES/ARTISTES/ARTISTE/@id
FILMSETARTISTES/FILMS/FILM/@Annee
FILMSETARTISTES/FILMS/FILM/MES/@idref
A l'aide de xmllint en mode «shell», essayer ensuite de répondre aux questions suivantes :
-
Quels sont tous les titres de films ?
-
Quel est le titre du neuvième film ?
-
En quelle année est sorti le film Blade Runner ?
-
Quel en est le metteur en scène ?
-
Combien y a-t-il de films ?
-
Quels éléments contiennent le texte "Bruce" ?
Essayer enfin à l'aide de PHP via le module SimpleXML d'afficher tous les titres de films avec leur numéro, comme ci-dessous :
Voici tous les titres de films
1. Vertigo
2. Alien
3. Titanic
4. Sacrifice
5. Volte/Face
6. Sleepy Hollow
7. American Beauty
8. Impitoyable
9. Gladiator
10. Blade Runner
11. Piège de cristal
12. 58 minutes pour vivre
13. Van Gogh
14. Seven
15. L'armée des douze singes
16. Le nom de la rose
17. Pulp fiction
18. Mary à tout prix
19. Terminator
20. Les dents de la mer
21. Le silence des agneaux
22. Le prince d'Egypte
23. Godzilla
24. Matrix
25. Mission: Impossible
26. Kagemusha
27. Les pleins pouvoirs
28. Le gendarme et les extra-terrestres
29. Les frères pétards
30. Le monde perdu
31. Rain Man
32. Top Gun
33. Les bronzés font du ski
34. MICROCOSMOS
35. Psychose
36. Le retour du Jedi
37. Les oiseaux
38. Reservoir dogs
39. Eyes Wide Shut
40. Shining
41. Pas de printemps pour Marnie
42. Fenêtre sur cour
43. La mort aux trousses
44. Jeanne d'Arc
45. Le cinquième élément
46. Léon
47. Nikita
48. Le grand bleu
Les lignes suivantes complétent la description des éléments uniques en chainant xmlstarlet, grep et sort :
xmlstarlet el -a films2.xml | grep "/@" | sort -u .
Voici les instructions à exécuter dans le shell de XMLLINT pour répondre aux questions posées :
# lancer xmllint en mode shell sur le fichier des films
xmllint --shell films2.xml
# tous les titres de films
ls //TITRE
# le titre du neuvième film (on trouve Gladiator)
# on aurait pu écrire en ligne de commandes
# grep TITRE films2.xml | head -n 9 | tail -n 1
ls //FILM[position()=9]/TITRE # plus "propre" que ls //FILM[9]/TITRE
# année de sortie du film Blade Runner (on trouve 1982)
ls //FILM[TITRE="Blade Runner"]/@Annee
## metteur en scène du film Blade Runner (Ridley SCOTT)
# solution 1, en deux temps : numéro du metteur en scène (on trouve 4)
ls //FILM[TITRE="Blade Runner"]//MES/@idref
# artiste associé
ls //ARTISTE[@id="4"]/ARTNOM # Scott
ls //ARTISTE[@id="4"]/ARTPRENOM # Ridley
# solution 2, tout en une seule fois
ls //ARTISTE[@id=//FILM[TITRE="Blade Runner"]//MES/@idref]/ARTNOM
# nombre de films (réponse 48)
xpath count(//FILM)
# quels éléments contiennent le texte Bruce (5 élements PRENOM et un élément ARTPRENOM)
grep Bruce
Pour les titres de films en PHP, une fois la documentation de SimpleXML parcourue, les exemples standards suffisent largement pour écrire le code suivant qui fournit l'affichage demandé :
<?php
echo "Voici tous les titres de films \n" ;
$nomFicXml = "films2.xml" ;
$films = simplexml_load_file($nomFicXml) ;
/* ce n'est pas exactement comme
$films = new SimpleXMLElement(file_get_contents($nomFicXml)) ;
*/
$nbFilms = 0 ;
foreach ($films->xpath('//TITRE') as $titre) {
$nbFilms++ ;
echo sprintf("%3d",$nbFilms).". " ;
echo $titre, PHP_EOL;
} // fin de pour chaque
?>
Que produit la transformation vide définie dans le fichier vide.xsl si on l'applique aux fichiers eau.xml, eau2.xml, trajets.xml et firefox.svg du T.P. 1 ?
<xsl:stylesheet xmlns:xsl='http://www.w3.org/1999/XSL/Transform' version="1.0">
<xsl:output method="text" encoding="UTF-8" />
</xsl:stylesheet>
Quelle est, pour ces fichiers, la différence avec la transformation minimaliste nommée minimaliste.xsl ?
<xsl:stylesheet xmlns:xsl='http://www.w3.org/1999/XSL/Transform' version="1.0">
<xsl:output method="text" encoding="UTF-8" />
<xsl:template match="/"><xsl:apply-templates /></xsl:template>
</xsl:stylesheet>
La transformation vide affiche les contenus textuels des éléments et ignore les attributs. Pour le fichier eau.xml on récupère donc trois lignes pratiquement vides (en fait on récupère les espaces d'indentation et les sauts de ligne, ce qui fait qu'à l'écran on ne voit rien. Pour le fichier eau2.xml on récupère les noms et les nombres d'atomes. Au niveau de trajets.xml, le type de train, le nom des gares de départ et d'arrivée sont affichés.
Pour la dessin du renard, il y a beaucoup de lignes vides affichées, ce qui fait qu'on risque de manquer le début de l'affichage. Heureusement, grep est notre ami ! On récupère donc comme seules lignes de texte le contenu des éléments title et desc.
$gh> xsltproc vide.xsl eau.xml # (3 lignes presque vides)
$gh> xsltproc vide.xsl eau2.xml
Oxygene
1
Hydrogene
2
$gh> xsltproc vide.xsl trajets.xml
TGV
Angers
Paris Montparnasse
Micheline
Troyes
Dijon
$gh> xsltproc vide.xsl firefox.svg # on ne voit rien
$gh> xsltproc vide.xsl firefox.svg | wc -l # 422 lignes, quand même !
$gh> xsltproc vide.xsl firefox.svg | grep "[a-z]" | wc -l # 2 lignes non vides
Il n'y a en principe aucune différence entre les transformations vide.xsl et minimaliste.xsl parce que la transformation vide applique toutes les règles par défaut à partir de la racine.
Que faut-il ajouter à la transformation minimaliste définie dans le fichier minimaliste.xsl pour qu'elle n'affiche que les noms d'atome si on l'applique au fichier eau2.xml ?
Une première solution consiste à définir deux règles, l'une pour l'élément nom et l'autre pour l'élément nombre. Pour nom on veut afficher le contenu alors que pour nombre on ne veut rien faire (puisqu'on ne veut pas afficher). Voici donc la solution dans mineau2.xsl :
<xsl:stylesheet xmlns:xsl='http://www.w3.org/1999/XSL/Transform' version="1.0">
<xsl:output method="text" encoding="UTF-8" />
<xsl:template match="/"><xsl:apply-templates /></xsl:template>
<xsl:template match="nom"><xsl:value-of select="." /></xsl:template>
<xsl:template match="nombre" />
</xsl:stylesheet>
Une meilleure solution est sans doute de ne demander que de déclencher les règles pour les éléments noms, soit le code du fichier mineau3.xsl :
<xsl:stylesheet xmlns:xsl='http://www.w3.org/1999/XSL/Transform' version="1.0">
<xsl:output method="text" encoding="UTF-8" />
<xsl:template match="/"><xsl:apply-templates select="//nom" /></xsl:template>
<xsl:template match="nom"><xsl:value-of select="." /><xsl:text> </xsl:text></xsl:template>
</xsl:stylesheet>
On voudrait compter le nombre de protéines du fichier leadb880.xml et vérifier que chaque protéine a bien un attribut length. Trouver une solution en ligne de commandes puis à l'aide d'une transformation XSL. Comment peut-on trouver la plus petite longueur de protéine et la plus grande via XSL ?
Ecrire ensuite un script PHP qui trouve la plus petite longueur de protéine et la plus grande.
Une protéine correspond à un élément protein donc il suffit de compter les chaines <protein> pour les dénombrer. L'attribut length, lui, peut se repérer grâce à l'expression <fasta length=. Dans les deux cas, on trouve 880 lignes correspondantes, mais cela ne prouve sans doute pas que chaque protéine a bien un attribut length dans un sous-élément fasta sauf à valider le fichier pour la grammaire leadb.dtd de la question 1 (ou à savoir bien compter, car 3521 = 1 + 4x880). Par contre rechercher toutes les expressions proteins/protein/fasta/@length dans la sortie de xmlstarlet el -a peut le prouver. Voici les commandes à utiliser :
grep "<protein>" leadb880.xml | wc -l # 880 protéines
grep "<fasta length=" leadb880.xml | wc -l # 880 longueurs
xmlstarlet el -a leadb880.xml | grep "proteins/protein/fasta/@length" | wc -l # 880 longueurs
Pour effectuer une transformation XSL qui réalise les mêmes comptages, il suffit de mettre une seule règle sur la racine du document et d'utiliser la fonction XPATH nommée count().
Transformation proteines.xsl :
<?xml version="1.0" encoding="ISO-8859-1" ?>
<xsl:stylesheet version="1.0" xmlns:xsl="http://www.w3.org/1999/XSL/Transform">
<xsl:output method="text" />
<xsl:template match="/">
Il y a <xsl:value-of select="count(//protein)" /> protéines
et <xsl:value-of select="count(//protein/fasta/@length)" /> longueurs.
</xsl:template>
</xsl:stylesheet>
Pour exécuter cette transformation, on peut utiliser xsltproc :
$gh> xsltproc proteines.xsl leadb880.xml
Il y a 880 protéines
et 880 longueurs.
mais ce n'est pas le seul choix possible. Par exemple on peut utiliser xmlstarlet :
$gh> xmlstarlet tr proteines.xsl leadb880.xml
Il y a 880 protéines
et 880 longueurs.
On peut aussi mettre l'appel du fichier XSL dans l'en-tête du fichier XML et faire exécuter la transformation par le navigateur, comme ici.
Pour trouver la longueur minimale et la longueur maximale, on peut se contenter de trier par ordre croissant ou décroissant et de garder la première valeur, soit le code XSL suivant :
<xsl:stylesheet version="1.0" xmlns:xsl="http://www.w3.org/1999/XSL/Transform">
<xsl:variable name="lngMin">
<xsl:for-each select="//@length">
<xsl:sort select="." data-type="number" order="ascending"/>
<xsl:if test="position() = 1"><xsl:value-of select="."/></xsl:if>
</xsl:for-each>
</xsl:variable>
<xsl:variable name="lngMax">
<xsl:for-each select="//@length">
<xsl:sort select="." data-type="number" order="descending"/>
<xsl:if test="position() = 1"><xsl:value-of select="."/></xsl:if>
</xsl:for-each>
</xsl:variable>
<xsl:template match="/">
Longueur minimale <xsl:value-of select="$lngMin" /> aa.
Longueur maximale <xsl:value-of select="$lngMax" /> aa.
</xsl:template>
</xsl:stylesheet>
dont le rendu est
# résultat de xsltproc prot_max.xsl leadb880.xml
Longueur minimale 66 aa.
Longueur maximale 843 aa.
Il n'y a aucune difficulté à utiliser de nouveau SimpleXML pour trouver en PHP la plus petite longueur et la plus grande longueur de protéine et la plus grande. Il faut juste ne pas oublier de convertir le texte issu de XPATH via la fonction intval() sous peine de trouver 110 aa à la fois comme plus petite valeur et plus grande valeur.
<?php
echo "Longueurs de protéines. \n" ;
$nomFicXml = "leadb880.xml" ;
$films = simplexml_load_file($nomFicXml) ;
$minLng = 10**6 ;
$maxLng = 0 ;
foreach ($films->xpath('//@length') as $lng) {
$lng = intval($lng) ;
if ($lng<$minLng) { $minLng = $lng ; } ;
if ($lng>$maxLng) { $maxLng = $lng ; } ;
} // fin de pour chaque
echo " longueur minimale $minLng aa, longueur maximale $maxLng aa.\n\n" ;
?>
Dans le document films2.xml, combien y a-t-il de films ? Et d'artistes ? Combien de références pour combien de metteurs en scène ? On écrira une transformation XSL qui affichera ces résultats en mode texte qu'on exécutera avec xmlstarlet avant de modifier le document pour avoir un rendu dans une page Web valide pour la grammaire XHTML Strict.
Remarque : on pourra utiliser le fichier stdWeb2.xsl qui contient des sous-programmes XSL adaptés à la production de pages Web.
Fichier stdWeb2.xsl
Compter avec XSL se fait à l'aide de la fonction count() dans un attribut select d'un élément xsl:value-of. Encore faut-il trouver les noeuds correspondants. Pour les films et les artistes, c'est assez simple car les éléments FILM et ARTISTE identifient de façon unique ce qu'on cherche donc il suffit de compter //FILM et //ARTISTE.
Pour trouver les metteurs en scène, il faut retenir les attributs id de ARTISTE qui correspondent à un attribut idref de MES, soit le filtre ARTISTE[@id=//MES/@idref] -- bien noter le double slash devant MES. Nous avons rajouté dans la solution des xsl:text pour rendre les affichages plus lisibles et nous avons mis le nom des metteurs en majuscules (avec des indications de solution XSL 2) :
<?xml version="1.0" encoding="ISO-8859-1" ?>
<xsl:stylesheet version="1.0" xmlns:xsl="http://www.w3.org/1999/XSL/Transform">
<xsl:output method="text" />
<!-- fichier nbfa1.xsl, s'applique à films2.xml -->
<xsl:template match="/">
<xsl:text>Il y a </xsl:text>
<xsl:value-of select="count(//FILM)" />
<xsl:text> films dans films2.xml</xsl:text>
<xsl:text> et </xsl:text>
<xsl:value-of select="count(//ARTISTE)" />
<xsl:text> artistes.
</xsl:text>
<xsl:value-of select="count(//MES)" />
<xsl:text> références de metteurs en scènes sont utilisées pour </xsl:text>
<!-- ceci est du XSL 2 :
<xsl:value-of select="count(distinct-values(//MES))" />
-->
<xsl:value-of select="count(//ARTISTE[./@id=//MES/@idref])" />
<xsl:text> metteurs en scène distincts.
</xsl:text>
</xsl:template>
<!-- fin de document -->
</xsl:stylesheet>
$gh> xmlstarlet tr nbfa1.xsl films2.xml
Il y a 48 films dans films2.xml et 117 artistes.
48 références de metteurs en scènes sont utilisées pour 32 metteurs en scène distincts.
Produire une page Web résultat est un peu plus compliqué car il faut aussi produire du code HTML ce qui signifie qu'il faut sans doute utiliser deux espaces de noms distincts.
Voici la solution :
<?xml version="1.0" encoding="ISO-8859-1" ?>
<xsl:stylesheet xmlns:xsl='http://www.w3.org/1999/XSL/Transform' version="1.0">
<xsl:import href="stdWeb2.xsl" />
<xsl:output method="xml" encoding="ISO-8859-1"/>
<!-- fichier films2.xsl, s'applique à films2.xml -->
<xsl:template match="/">
<xsl:call-template name="debutPage">
<xsl:with-param name="leTitre">Films et artistes avec table des matières</xsl:with-param>
<xsl:with-param name="h1redite">no</xsl:with-param>
</xsl:call-template>
<xsl:call-template name="debutSection" />
<blockquote>
<h1>Comptages dans le fichier <a href="films2.xml">films2.xml</a></h1>
<p>
Il y a <b><xsl:value-of select="count(//FILM)" /></b> films dans ce fichier
et <strong><xsl:value-of select="count(//ARTISTE)" /></strong> artistes.
</p>
<p>
<span class="grougef"><xsl:value-of select="count(//MES)" /></span>
références de metteurs en scènes sont utilisées pour
<!-- ceci est du XSL 2 :
<xsl:value-of select="count(distinct-values(//MES))" />
-->
<span class="gbleuf"><xsl:value-of select="count(//ARTISTE[./@id=//MES/@idref])" /></span>
metteurs en scène distincts.
</p>
</blockquote>
<xsl:call-template name="finSection" />
<xsl:call-template name="finPage" />
</xsl:template>
<!-- fin de document -->
</xsl:stylesheet>
Le rendu correspondant est ici.
On voudrait effectuer des calculs statistiques et tracer des graphiques pour étudier statistiquement la longueur des protéines du fichier leadb880.xml. Que peut-on calculer et tracer via XSL ?
Est-ce qu'écrire un script PHP est ici adapté ?
XSL ne peut pas grand chose pour nous ici car XSL est conçu pour transformer du texte, pas pour calculer. PHP n'est pas vraiment non plus prévu pour réaliser des traitements statistiques. Par contre XSL peut nous aider à produire un fichier CSV utilisable par Excel, le logiciel R ou tout autre logiciel statistique. Pour cela, il suffit d'exporter le nom de chaque protéine, sa classe et sa longueur. C'est ce que fait la transformation suivante :
<?xml version="1.0" encoding="ISO-8859-1" ?>
<xsl:stylesheet version="1.0" xmlns:xsl="http://www.w3.org/1999/XSL/Transform">
<xsl:output method="text" />
<xsl:template match="/">
<xsl:text>Protein ; Classe ; Longueur</xsl:text>
<xsl:apply-templates match="protein" />
</xsl:template>
<xsl:template match="protein">
<xsl:value-of select="accession" />;<xsl:value-of select="class" />;<xsl:value-of select="fasta/@length" />
</xsl:template>
</xsl:stylesheet>
On pourra vérifier que les données obtenues par la commande xsltproc prot_export.xsl leadb880.xml | grep -v "^$" | head soit le fichier prot_export.csv est utilisable par Microsoft Office Excel ou par Libre Office Calc :
On peut alors analyser ces données via le script R suivant
## lecture du fichier de longueurs de protéines et analyse univariée
source("http://forge.info.univ-angers.fr/~gh/wstat/statgh.r",encoding="latin1")
prot <- read.csv("prot_export.csv",sep=";",head=TRUE)
gr("protLng.png")
decritQT("Longueur",prot$Longueur,"aa",TRUE)
dev.off()
qui produit les résultats ci-dessous
DESCRIPTION STATISTIQUE DE LA VARIABLE Longueur
Taille 880 individus
Moyenne 193.4989 aa
Ecart-type 102.3758 aa
Coef. de variation 53 %
1er Quartile 130.0000 aa
Mediane 167.0000 aa
3eme Quartile 229.0000 aa
iqr absolu 99.0000 aa
iqr relatif 59.0000 %
Minimum 66.000 aa
Maximum 843.000 aa
Tracé tige et feuilles
The decimal point is 2 digit(s) to the right of the |
0 | 77777888888899999999999999999999999999999999999999999999999999999999
1 | 00000000000000000000000000000000000000000000000000000000000111111111+142
1 | 55555555555555555555555555555555555555555555555555555555555555555566+177
2 | 00000000000000000000000000000000000000000111111111111111111112222222+64
2 | 55555555555555555555666666666666666666666666666666777777777788888888
3 | 00000000000111111222222222222333333333344444444
3 | 5555555566667889999
4 | 0111223
4 | 556677777889
5 | 00124
5 | 566888
6 | 1234
6 |
7 | 344
7 |
8 | 24
ainsi que le graphique suivant :
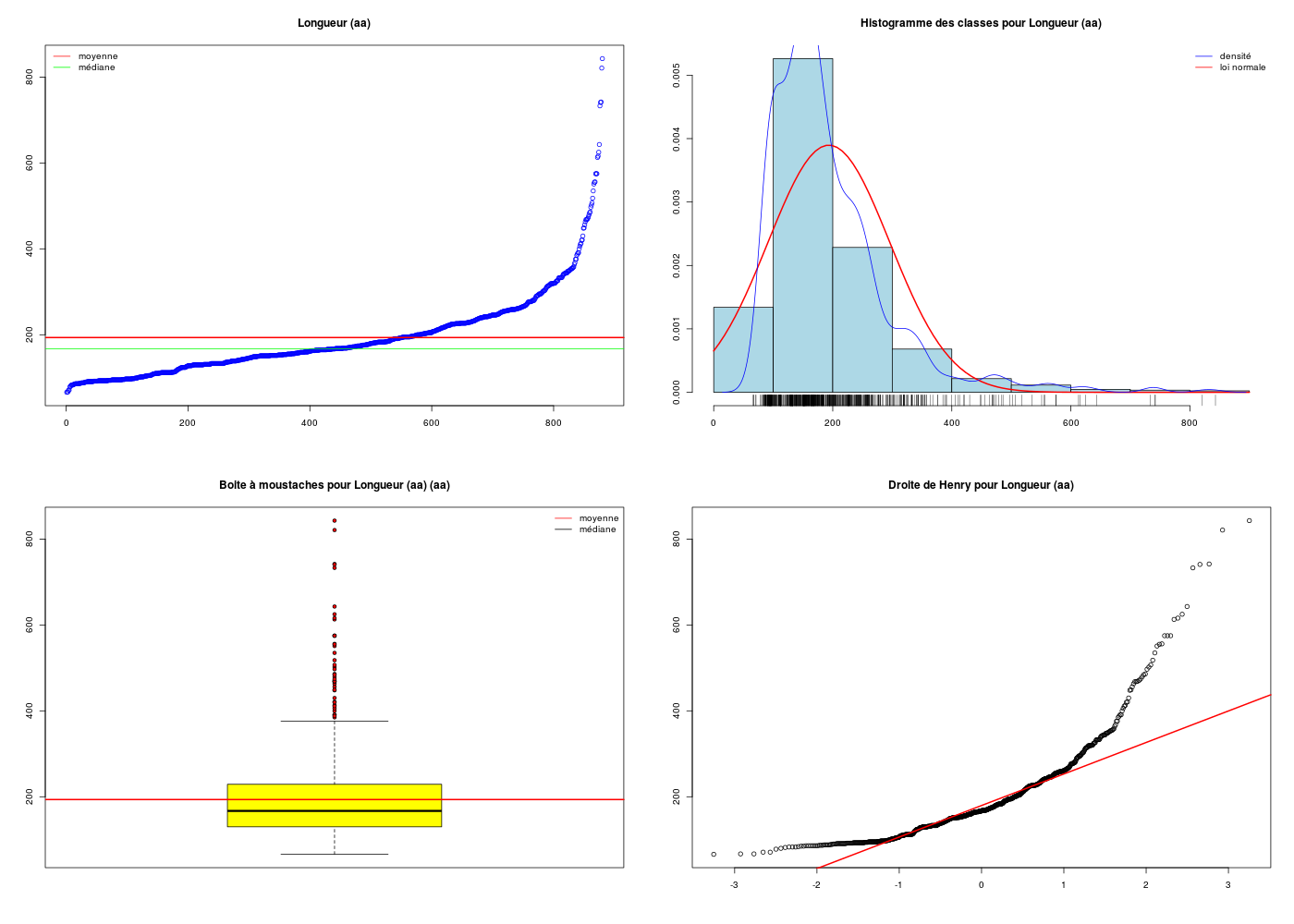
Nous laissons le soin au lecteur de rédiger le commentaire statistique associé. Le script R utilise notre fonction decritQT() décrite ici.
Questions sans réponse affichée dans le navigateur (donc venez en TP !)
-
Pourquoi est-ce difficile de construire une grammaire XSD fine et précise pour les protéines LEA si on n'est pas biologiste ?
-
Comment faire pour trouver les attributs uniques si on ne connait pas l'option -u de la commande sort ?
-
Peut-on compter des "non-éléments" ou des éléments sans attributs ? Par exemple, pour films2.xml combien manque-t-il de résumés ? et de dates de naissance ?
-
Pourquoi faut-il compter les attributs avec /@ plutôt qu'avec seulement @ ?
Code-source php de cette page.
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)
