Révision R à l'I.N.R.A., novembre 2014
Le but de ces deux demi-journées de révision est d'installer R, Rstudio et Rcmdr sur les portables de l'INRA
puis de vérifier que vous arrivez à réaliser les premières manipulations et calculs statistiques simples sur vos données à vous.
On trouvera donc ici à la fois des considérations conceptuelles et des informations techniques, le tout devrait permettre de savoir réaliser concrétement des petites analyses et des tracés graphiques simples tout en sachant s'adapter et progresser seul(e). La programmation R ne sera pas abordée mais on montrera comment écrire et exécuter des petits scripts (ensembles de commandes).
Pour que cette page soit facile à lire et à relire, tout est présenté sous forme de questions et réponses.
Table des matières cliquable
1. Installation de R
2. Installation de Rstudio
3. Installation de Rcmdr
4. Lecture de fichiers Excel
5. Utilisation de l'aide
6. Gestion de packages
7. Calculs en colonnes
8. Réalisation de graphiques
9. Lecture de fichiers-texte
10. Création de variables
11. Fusion de données
12. Calculs par sous-groupes
13. Automatisation de commandes
Il est possible d'afficher toutes les solutions via ?solutions=1 et de les masquer avec ?solutions=0.
1. Installation de R
Comment installer R sur un portable ?
Pour installer R sur un portable, comme sur un ordinateur fixe, il suffit de se rendre sur le site officiel nommé CRAN et de télécharger l'exécutable correspondant au système d'exploitation souhaité (Windows, MacOs, Unix). Pour Linux (Ubuntu), une page dédiée explique comment installer R via sudo apt-get. Pour Windows, l'exécutable est à l'adresse http://cran.r-project.org/bin/windows/base/.
La version courante en octobre 2014 est R 3.1 et l'exécutable fait environ 54 Mo.
Sous Windows, une fois R installé, on dispose d'une icone comme celle ci-dessous pour l'exécuter :

2. Installation de Rstudio
Faut-il installer Rstudio ? Si oui, comment faire pour l'installer ?
A notre avis, il faut installer Rstudio car l'interface standard de R est bien trop minimale. Là encore, il y a juste un exécutable à télécharger et à exécuter pour installer Rstudio à l'adresse http://www.rstudio.com/.
La version courante en octobre 2014 est Rstudio 0.98 et l'exécutable fait environ 52 Mo.
Une fois Rstudio installé, on dispose d'une icone comme celle ci-dessous pour l'exécuter :

Pour les utilisateurs de Windows, si l'icone de Rstudio n'est pas sur le bureau, il suffit de créer un raccourci vers Démarrer/Programmes/Rstudio/Rstudio.exe pour l'obtenir.
3. Installation de Rcmdr
Faut-il installer Rcmdr ? Si oui, comment faire pour l'installer ?
Et comment l'utiliser ensuite ? Faut-il l'utiliser dans RStudio ou en dehors de RStudio ?
A notre avis, il faut installer Rcmdr quand on débute avec R car cela fournit des menus cliquables, ce qui est bien pratique et rassurant. Par contre, au bout d'un moment, si on veut réaliser des manipulations plus sophistiquées et pour profiter de toutes les options et subtilités de R, il faut apprendre à s'en passer.
Rcmdr est un package. Il faut l'installer à partir de R (ou Rstudio). En cas de souci, il y a une page Web pour son installation mais elle est en anglais, ce qui peut en déranger certain(e)s. En fait, il suffit juste d'exécuter l'instruction install.packages("Rcmdr",dependencies=TRUE) dans une session R. Il est possible que R demande le nom d'un serveur-miroir de R, comme dans la copie-d'écran ci-dessous :
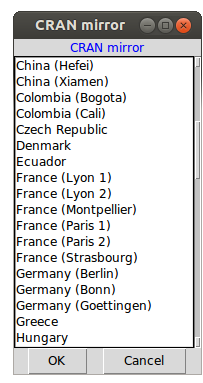 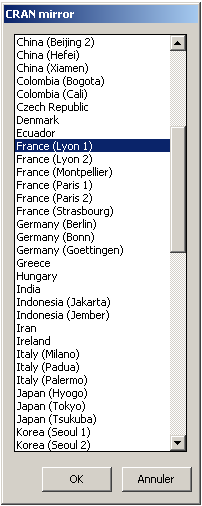
Le plus simple alors est sans doute de choisir un miroir français à Lyon.
Il est possible de définir ce miroir directement dans la commande d'installation, par exemple en écrivant :
install.packages("Rcmdr",dependencies=TRUE,repos="http://cran.univ-lyon1.fr/")
Cette installation de package se fait une fois pour toutes. Le package Rcmdr une fois installé est disponible pour chaque session de R, il suffit juste de le charger via la commande
library("Rcmdr")
On doit alors voir le menu de Rcmdr apparaitre, qui ressemble à

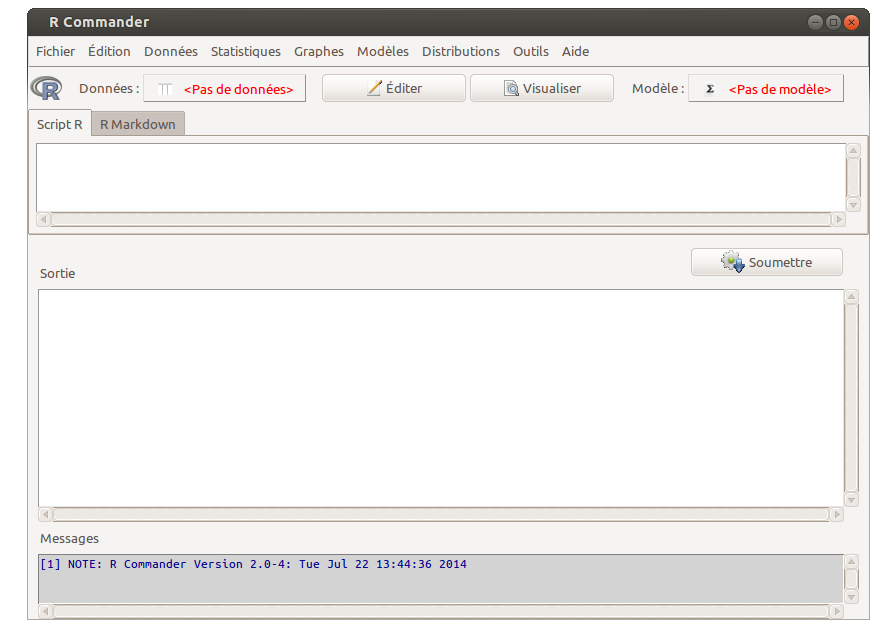
En ce qui concerne l'utilisation de Rcmdr, on peut l'utiliser seul avec l'interface standard de R ou l'utiliser dans Rstudio, c'est au choix.
Si on dispose d'un portable avec un petit écran, utiliser Rcmdr directement sous R est sans doute suffisant et plus agréable à utiliser.
4. Lecture de fichiers Excel
Comment lire le fichier Excel exemple1.xls qui ne contient qu'une feuille de calcul ?
Comment lire la deuxième feuille de calcul du fichier Excel exemple2.xls ?
On essaiera de réaliser les lectures avec et sans Rcmdr, dans l'ordre qui vous parait le plus simple.
Est-ce plus compliqué avec des fichiers .xlsx comme exemple3.xlsx et exemple4.xlsx ?
4.1 Lecture de fichiers XLS avec le package gdata
Pour lire un fichier Excel de type xls la fonction read.xls() du package gdata est suffisante.
Voici le code à écrire :
# chargement du package gdata
library(gdata)
# lecture du fichier Excel qui ne contient qu'une seule feuille de calcul
xmp1 <- read.xls("exemple1.xls",encoding="latin1",row.names=1)
# taille des données
cat(" dimensions (lignes,colonnes) : ",dims(xmp1),"\n")
# affichage du début des données
cat("voici les premières lignes\n")
print(head(xmp1))
# affichage de la fin des données
cat("voici les dernières lignes\n")
print(tail(xmp1))
# nature des colonnes
cat("voici les colonnes et leur nature\n")
print(cbind(sapply(X = xmp1,FUN = class)))
Et son résultat :
dimensions (lignes,colonnes) : 20 x 8
voici les premières lignes
CODESEXE AGE PROF CODEETUD REGI USAG SEXE ETUDES
M001 1 62 1 2 2 3 Femme Bepc
M002 0 60 9 3 4 1 Homme Bac
M003 1 31 9 4 4 1 Femme Supérieur
M004 1 27 8 4 1 1 Femme Supérieur
M005 0 22 8 4 1 2 Homme Supérieur
M006 1 70 4 1 1 1 Femme Primaire
voici les dernières lignes
CODESEXE AGE PROF CODEETUD REGI USAG SEXE ETUDES
M015 1 48 1 2 3 1 Femme Bepc
M016 1 50 1 1 4 2 Femme Primaire
M017 0 49 7 4 4 1 Homme Supérieur
M018 1 44 1 1 2 1 Femme Primaire
M019 1 21 8 4 2 2 Femme Supérieur
M100 1 48 9 4 2 0 Femme Supérieur
voici les colonnes et leur nature
CODESEXE "integer"
AGE "integer"
PROF "integer"
CODEETUD "integer"
REGI "integer"
USAG "integer"
SEXE "factor"
ETUDES "factor"
Si malheureusement au lieu de la lecture du fichier Excel vous voyez un message d'erreur comme
Erreur dans library("gdata") : aucun package nommé 'gdata' n'est trouvé.
Cela signifie que le package gdata n'est pas installé. Comme pour le package Rcmdr il suffit de l'installer avec la commande :
install.packages("gdata",dependencies=TRUE,repos="http://cran.univ-lyon1.fr/")
Si par contre le message d'erreur est
unable to locate valid Perl interpreter
perl executable not found
cela signifie que le langage et logiciel Perl n'est pas installé sur votre machine, ce qui est fréquent. Et comme le package gdata utilise Perl pour lire les fichiers Excel, il y a un souci. Nous vous conseillons pour résoudre ce problème de télécharger la version gratuite nommée Active Perl à l'adresse http://www.activestate.com/activeperl. Une fois le fichier téléchargé (environ 26 Mo) et exécuté, il faut fermer Rstudio et le relancer avant de pouvoir profiter de Perl dans Rstudio. Vous n'avez bien sûr absolument pas besoin de connaitre Perl, il doit simplement être installé.
Il y a des options de lecture comme verbose et stringsAsFactors dans la fonction read.xls() qui sont parfois utiles. Voici comment les utiliser :
# chargement du package gdata
library(gdata)
# lecture du fichier Excel qui ne contient qu'une seule feuille de calcul
# on utilise les options verbose et stringsAsFactors
xmp1opt <- read.xls(
xls="exemple1.xls",
encoding="latin1",
row.names=1,
verbose=TRUE,
stringsAsFactors=FALSE
) # fin de read.xls
# taille des données
cat(" dimensions (lignes,colonnes) : ",dims(xmp1opt),"\n")
# affichage du début des données
cat("voici les premières lignes\n")
print(head(xmp1opt))
# affichage de la fin des données
cat("voici les dernières lignes\n")
print(tail(xmp1opt))
# nature des colonnes
cat("voici les colonnes et leur nature\n")
print(cbind(sapply(X = xmp1opt,FUN = class)))
Et leur résultat :
Using perl at C:\Perl\bin\perl.exe
Using perl at C:\Perl\bin\perl.exe
Converting xls file
“exemple1.xls”
to csv file
“C:\DOCUME~1\ADMINI~1\LOCALS~1\Temp\Rtmp2XuhM8\filec4c7dad1e9f.csv”
...
Executing '
"C:\Perl\bin\perl.exe"
"C:/Program Files/R/R-3.1.1/library/gdata/perl/xls2csv.pl"
"exemple1.xls"
"C:\DOCUME~1\ADMINI~1\LOCALS~1\Temp\Rtmp2XuhM8\filec4c7dad1e9f.csv" "1"
'...
Loading 'exemple1.xls'...
Done.
Orignal Filename: exemple1.xls
Number of Sheets: 1
Writing sheet number 1 ('elf_xls') to file 'C:\DOCUME~1\ADMINI~1\LOCALS~1\Temp\Rtmp2XuhM8\filec4c7dad1e9f.csv'
Minrow=0 Maxrow=20 Mincol=0 Maxcol=8
(Ignored 0 blank lines.)
0
Done.
Reading csv file “C:\DOCUME~1\ADMINI~1\LOCALS~1\Temp\Rtmp2XuhM8\filec4c7dad1e9f.csv” ...
Done.
dimensions (lignes,colonnes) : 20 x 8
voici les premières lignes
CODESEXE AGE PROF CODEETUD REGI USAG SEXE ETUDES
M001 1 62 1 2 2 3 Femme Bepc
M002 0 60 9 3 4 1 Homme Bac
M003 1 31 9 4 4 1 Femme Supérieur
M004 1 27 8 4 1 1 Femme Supérieur
M005 0 22 8 4 1 2 Homme Supérieur
M006 1 70 4 1 1 1 Femme Primaire
voici les dernières lignes
CODESEXE AGE PROF CODEETUD REGI USAG SEXE ETUDES
M015 1 48 1 2 3 1 Femme Bepc
M016 1 50 1 1 4 2 Femme Primaire
M017 0 49 7 4 4 1 Homme Supérieur
M018 1 44 1 1 2 1 Femme Primaire
M019 1 21 8 4 2 2 Femme Supérieur
M100 1 48 9 4 2 0 Femme Supérieur
voici les colonnes et leur nature
CODESEXE "integer"
AGE "integer"
PROF "integer"
CODEETUD "integer"
REGI "integer"
USAG "integer"
SEXE "character"
ETUDES "character"
On remarquera que du coup les colonnes SEXE et ETUDES n'ont pas été lues et comprise de la même façon :(avant c'était des facteurs, maintenant ce sont des simples chaines de caractères.
Pour lire une feuille particulière, il faut utiliser l'option sheet soit ici, pour la feuille 2 qui est nommée partie 2 :
# chargement du package gdata
library(gdata)
# lecture de feuille 2 du fichier Excel
xmp2 <- read.xls(
xls="exemple2.xls",
encoding="latin1",
row.names=1,
stringsAsFactors=FALSE,
sheet=2
) # fin de read.xls
# taille des données
cat(" dimensions (lignes,colonnes) : ",dims(xmp2),"\n")
# on peut aussi utiliser le nom de la feuille
xmp2autre <- read.xls(
xls="exemple2.xls",
encoding="latin1",
row.names=1,
stringsAsFactors=FALSE,
sheet="partie 2"
) # fin de read.xls
# vérification de l'égalité entre les deux lectures
if (identical(xmp2,xmp2autre)) {
cat("égalité des deux lectures\n")
} else {
cat("les des deux lectures ne sont pas identiques\n")
} # fin de si
dimensions (lignes,colonnes) : 10 x 8
égalité des deux lectures
4.2 Lecture de fichiers XLS avec le package Rcmdr
Pour commencer à utiliser le package Rcmdr il faut le charger avec la commande :
library("Rcmdr")
Il y a de fortes chances que le premier chargement de Rcmdr affiche un message comme
Le package XLConnect n'est pas installé. Voulez-vous l'installer ?
ou un panneau comme
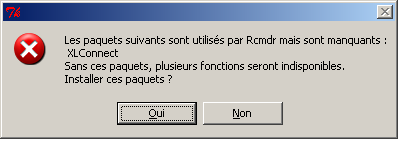
Il faut alors répondre OUI afin de pouvoir lire les fichiers Excel avec Rcmdr qui, vous l'avez compris, utilise le package XLConnect pour ces lectures. XLConnect utilise lui-même Java à travers le package rJava, mais comme pour Perl, vous n'avez bien sûr absolument pas besoin de connaitre Java, il doit simplement être installé.
Si Rcmdr a démarré, son système de menus est visible et il faut utiliser le menu Données/Importer des données pour lire les données Excel comme ci-dessous :
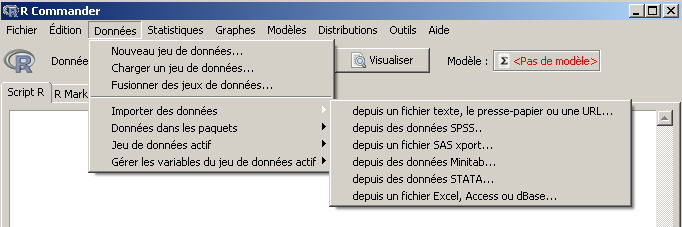
Rcmdr demande alors un nom de dataset c'est-à-dire le nom d'une variable pour les données. On peut par exemple saisir elf et si (cela arrive sur certains ordinateurs) Rcmdr demande de choisir une "table" dans une petite fenêtre qui apparait, il faut choisir la table elf.xls.
Pour connaitre le nom des variables de ce jeu de données, il est ensuite possible d'utiliser le menu Données, sous-menu Jeu de données actif, sous-sous-menu Variables du jeu de données actif mais il faut reconnaitre que taper directement et "Soumettre" names(elf) est beaucoup plus court à écrire ! On peut aussi cliquer sur le bouton Visualiser pour voir les données dans une fenêtre séparée...
4.3 Lecture de fichiers XLSX
Ce qui a été dit au-dessus s'applique tel-quel : les deux packages gdata et XLConnect lisent aussi bien les fichiers XLS que XLSX.
En cas de souci avec la lecture de fichier XLSX, il ne faut pas hésiter à les ouvrir en de hors de R, Rstudio et Rcmdr avec Microsoft Office ou Open Office et les sauvegarder au format XLS ou CSV.
5. Utilisation de l'aide
Où trouver de l'aide et de la documentation sur les fonctions de R ? Et sur les packages ?
Où trouver de l'aide sur Rcmdr ? Et sur Rstudio ? En français ?
Si on veut de l'aide sur une fonction particulière, il suffit de taper help(topic="f") ou, plus simplement, help("f") où f est le nom de la fonction. Par exemple, si on cherche à réaliser un calcul de moyenne tronquée, comme mean est le nom de la fonction qui calcule les moyennes, on tape help("mean") pour voir les paramètres de mean() et leur explication. Si on tape example("mean"), au lieu de l'aide, on a des exemples d'utilisation.
La fonction help() permet aussi d'avoir de l'aide sur les packages. Par exemple help(package="gdata") affiche la page d'aide du package gdata.
Pour des documentations plus fournies, il y a la page des documents du CRAN qui contient de nombreux documents en anglais puis dans de nombreuses autres langues à la rubrique Other Languages (soit en gros une dizaine de livres R en français gratuits et au format PDF).
R a pris tellement d'ampleur depuis le début des années 2010 qu'il suffit de rechercher cran software avec google pour trouver de nombreuses aides. En particulier, nous conseillons le texte de C. Jost dont une copie locale est ici.
On trouve bien sûr des vidéos sur R en France, notamment à Rennes mais aussi chez nos amis suisses : installer-et-lancer-rcmdr.
Le package Rcmdr, en plus de ses fichiers au CRAN dispose de sa propre page : http://socserv.mcmaster.ca/jfox/Misc/Rcmdr/.
Il est par contre un peu plus difficile de trouver de la documentation sur Rstudio en français, mais en cherchant, on trouve par exemple cette présentation de 2012 et cette présentation de 2013 qui devraient suffire pour nos révisions...
On peut aussi utiliser l'aide intégérée à chacun des logiciels avec le menu Aide ou Help (en anglais).
Pour ceux et celles qui préfèrent utiliser un navigateur pour afficher les pages d'aide, on peut aussi taper
help.start()
Ensuite, toute demande d'aide via la fonction help() vient s'afficher dans le navigateur.
6. Gestion de packages
Comment installer un package, comme le package beanplot ?
Et si un package requiert d'autres packages, comment faire pour l'installer ?
Comment charger en mémoire un package ? Et comment le retirer de la mémoire ? Comment lister les objets d'un package dont les données d'exemples du package ? Comment obtenir l'aide sur un package ?
On installe un package avec la fonction install.packages() du package utils. On peut installer plusieurs packages d'un coup avec le paramètre pkgs, spécifier l'endroit où télécharger le package avec le paramètre repos, indiquer s'il faut charger les packages liés avec le paramètre dependencies... Voici quelques exemples d'utilisation que vous pouvez copier/coller :
install.packages(pkgs="beanplot")
install.packages(pkgs=c("beanplot","gdata"),repos="http://cran.univ-lyon1.fr/")
install.packages(
pkgs=c("beanplot","gdata"),
repos="http://cran.univ-lyon1.fr/",
dependencies=TRUE
) # fin de install.packages
Pour désinstaller un package, on utilise la fonction remove.packages() du même package utils :
remove.packages(pkgs="beanplot")
On charge un package en mémoire avec la fonction library() du package base et on le retire avec la fonction detach() de ce même package base.
library(beanplot)
print(head(beanplot)) # OK, le package a été chargé en mémoire
detach(package:beanplot)
print(head(beanplot)) # erreur, le package n'est plus en mémoire
> library(beanplot)
> print(head(beanplot))
1 function (..., bw = "SJ-dpi", kernel = "gaussian", cut = 3, cutmin = -Inf,
2 cutmax = Inf, grownage = 10, what = c(TRUE, TRUE, TRUE, TRUE),
3 add = FALSE, col, axes = TRUE, log = "auto", handlelog = NA,
4 ll = 0.16, wd = NA, maxwidth = 0.8, maxstripline = 0.96,
5 method = "stack", names, overallline = "mean", beanlines = overallline,
6 horizontal = FALSE, side = "no", jitter = NULL, beanlinewd = 2,
> detach(package:beanplot)
> print(head(beanplot))
Erreur dans head(beanplot) :
erreur d'évaluation de l'argument 'x' lors de la sélection
d'une méthode pour la fonction 'head' :
Erreur : objet 'beanplot' introuvable
Avec la fonction data() du package utils on peut lister les données présentes en mémoire ou dans un package. Par exemple :
data(package="gdata")
Pour connaitre tous les objets d'un package, et donc en particulier les fonctions du package, il faut passer par la fonction ls() du package base :
ls("package:gdata")
Attention : avant d'utiliser ls pour connaitre les objets du package, il faut d'abord charger le package car sinon on obtient le message d'erreur suivant :
# essai de ls("package:beanplot")
# alors que le package beanplot n'est pas chargé en mémoire
> ls("package:beanplot")
aucun élément du nom de "package:beanplot" dans la liste de recherche
Comme indiqué à la question précédente, la fonction help() permet d'afficher l'aide sur un package. Par exemple help(package="gdata") affiche la page d'aide du package gdata.
Pour gérer des packages avec Rstudio, il faut utiliser l'onglet packages du panneau inférieur droit (dans la configuration originale) :
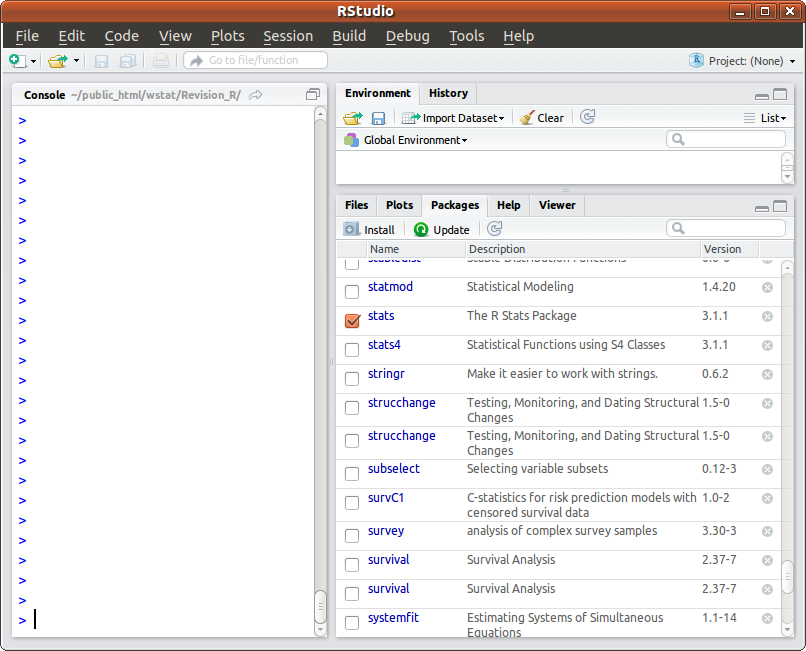
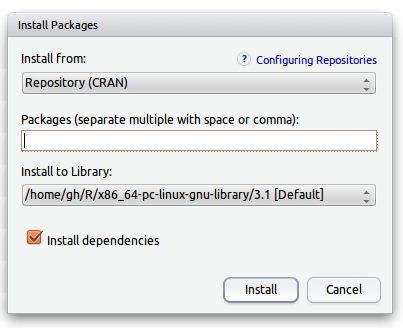
On peut cliquer sur un package installé pour le charger, utiliser le bouton Install pour installer un package :
Pour charger un package avec Rcmdr, il faut aller dans le menu Outils sous-menu Charger des paquets.
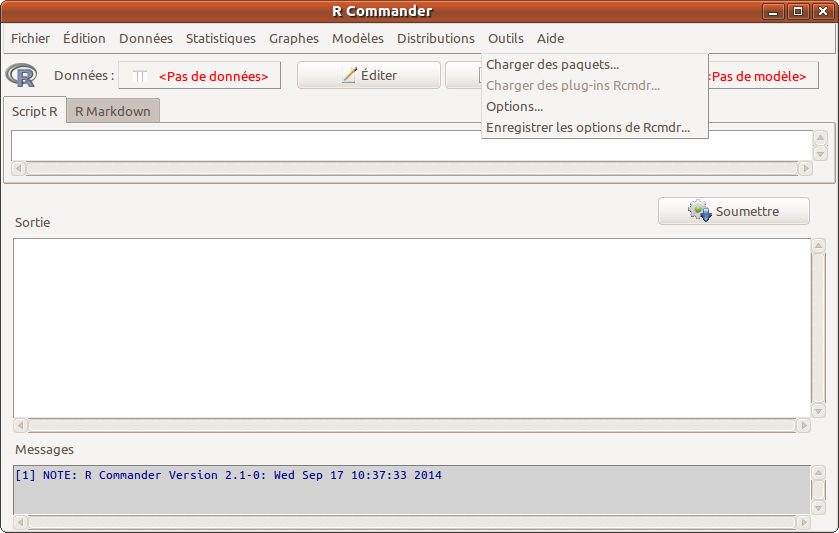
Il est alors possible de choisir un ou plusieurs paquets (packages) dans le panneau qui apparait. Pour une sélection multiple, il faut utiliser la touche CTRL en même temps qu'on effectue un clic droit.
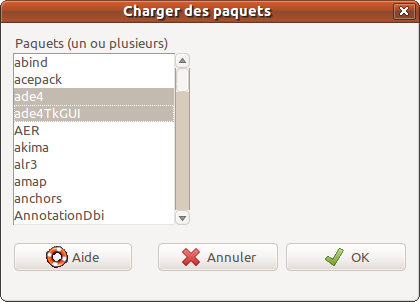
A notre connaissance, il n'est rien prévu pour installer un package avec Rcmdr.
7. Calculs en colonnes
Comment calculer les moyennes pour les trois premières colonnes quantitatives du fichier Excel her.xls puis trier ces moyennes par ordre décroissant ?
Le détail des données Health Exam Results est à l'adresse HER.
On pourra commencer par utiliser Rcmdr avant d'écrire une solution qui ne passe pas par des clic-souris.
7.1 Avec Rcmdr
On commence par lire et charger les données comme à l'exercice 4 via le menu Données, sous-menu Importer des données, sous-sous-menu depuis un fichier Excel, Access ou dBase. On nomme her ces données :
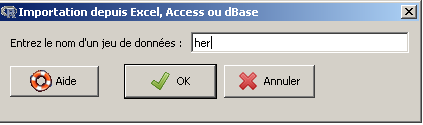
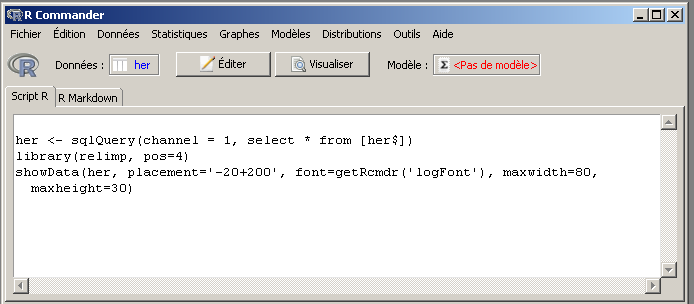
Ensuite, on clique sur le bouton Visualiser pour voir les données.
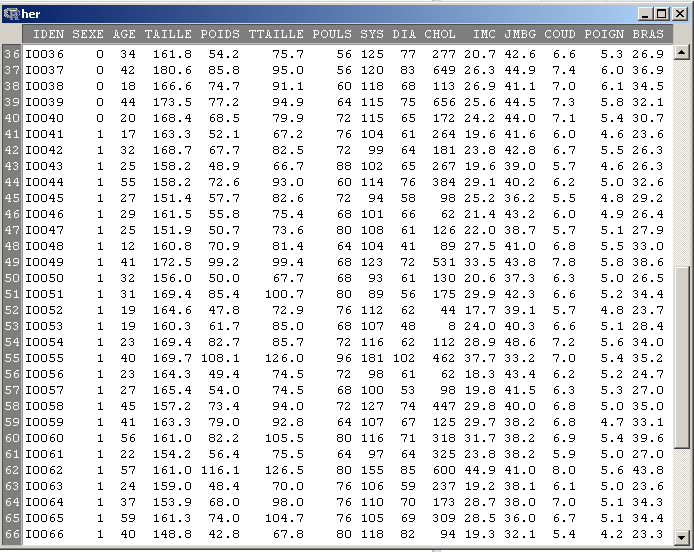
Dans la fenêtre qui apparait, les deux premières colonnes, IDEN et SEXE ne sont visiblement pas quantitatives. Les trois premières colonnes quantitatives sont donc AGE, TAILLE et POIDS (mais on ne connait pas les unités).
Pour calculer les moyennes, on passe par le menu Statistiques, sous-menu Résumés, sous-sous-menu Statistiques descriptives. Là, dans le panneau de Statistiques générales qui apparait, il faut sélectionner nos trois variables AGE, TAILLE et POIDS. C'est un peu compliqué parce qu'il faut faire une sélection multiple et parce que les variables sont rangées par ordre alphabétique. On commence donc par faire un clic gauche sur AGE puis on utilise l'ascenseur jusqu'à voir la variable POIDS et là on appuie sur la touche Ctrl en même temps qu'on fait un clic gauche sur POIDS. On recommence ensuite avec la variable TAILLE. Les trois variables sont alors sélectionnées.
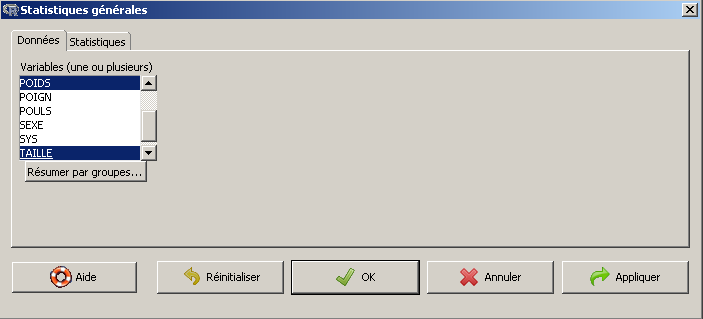
Attention : il ne faut pas tout de suite appuyer sur le bouton OK car comme on ne veut que la moyenne, il faut cliquer sur l'onglet Statistiques et décocher tous les calculs autres que Moyenne.
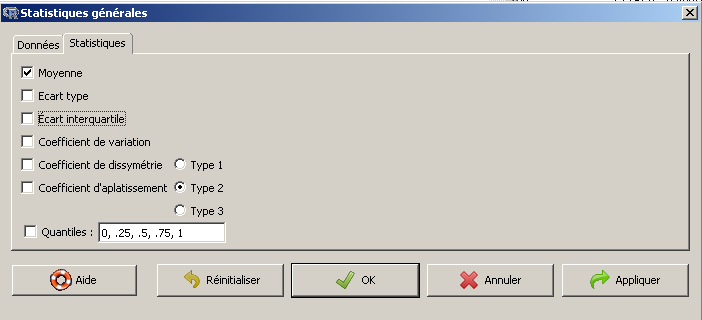
On peut enfin appuyer sur le bouton OK et obtenir les moyennes demandées.
Malheureusement RCmdr ne dispose d'aucun moyen de trier les moyennes.
7.2 Sans passer par Rcmdr, solution naive
Puisqu'on connait le nom des variables, on peut calculer nos trois moyennes avec trois appels successifs à la fonction mean(). Nous montrons au passage différentes façons d'accéder aux variables :
# chargement du package gdata afin d'utiliser la fonction read.xls()
library("gdata")
# lecture du fichier Excel "her.xls" dans la variable her
her <- read.xls("her.xls")
# calcul de la moyenne de l'age via le nom de la variable
moyAge <- mean(her$AGE)
# calcul de la moyenne du poids via le numéro de la variable
moyPoids <- mean(her[,5]) # car poids est la variable numéro 5
# calcul de la moyenne de la taille
# on utilise with() pour accéder aux données
moyTaille <- with(her,mean(TAILLE))
# on forme un vecteur avec ces moyennes avec la fonction c()
moyennes <- c(moyAge, moyPoids, moyTaille)
# on affiche ce vecteur dans cet ordre
cat(" voici les moyennes :")
print( moyennes )
# puis on l'affiche par ordre décroissant
cat(" voici les moyennes décroissantes :")
print( sort(moyennes,decreasing=TRUE) )
voici les moyennes : 34.35 72.2925 167.04
voici les moyennes décroissantes : 167.04 72.2925 34.35
Le tri des moyennes est assuré par la fonction sort() du package base avec le paramètre decreasing pour obtenir l'ordre décroissant. Comme ce n'est pas très lisible, il vaudrait mieux nommer les éléments du vecteur des moyennes comme suit :
# on forme un vecteur avec les moyennes avec la fonction c()
moyennes <- c(moyAge, moyPoids, moyTaille)
# on nomme ce vecteur afin de s'y retrouver
names(moyennes) <- c("AGE","POIDS","TAILLE")
# on affiche ce vecteur dans cet ordre
cat(" voici les moyennes :\n")
print( moyennes )
# puis on l'affiche par ordre décroissant
cat(" voici les moyennes décroissantes :\n")
print( sort(moyennes,decreasing=TRUE) )
voici les moyennes :
AGE POIDS TAILLE
34.3500 72.2925 167.0400
voici les moyennes décroissantes :
TAILLE POIDS AGE
167.0400 72.2925 34.3500
7.3 Sans passer par Rcmdr, solution plus évoluée
Les instructions précédentes sont relativement simples à écrire mais peu pratiques si le nombre de variables est important. Voici donc une solution plus générale. On commence par extraire les données qui nous intéressent avant d'utiliser la fonction apply() du package base sur les colonnes (dimension 2) avec la fonction mean comme paramètre :
# extraction des données
her_selection <- her[ , c("AGE","TAILLE","POIDS") ]
# calcul des moyennes par colonne avec apply
apply(X = her_selection, MARGIN = 2,FUN = mean)
AGE TAILLE POIDS
34.3500 167.0400 72.2925
On remarquera que le vecteur résultat est nommé, ce qui est très lisible.
7.4 Deux remarques pour conclure
Le tri sur les moyennes n'a ici aucun sens. Si on avait utilisé des unités, on aurait eu une moyenne de 34.35 ans pour l'age et 167.04 cm pour la taille. Les comparer est vraiment stupide, mais si vous avez fait ce tri, c'est que vous suivez aveuglément ce cours alors que les statistiques demande à réfléchir...
Même en admettant que le tri sur les moyennes ait un sens, rien ne prouve que les données sont "correctes". Il serait prudent d'afficher au moins le minimum et le maximum des variables pour s'assurer qu'il n'y a pas de donnée aberrante, par exemple comme ceci :
# extraction des données
her_selection <- her[ , c("AGE","TAILLE","POIDS") ]
# calcul par colonne avec apply
res <- apply(X = her_selection, MARGIN = 2,FUN = function(x) return( c(min(x),mean(x),max(x))))
# rappel des calculs
row.names(res) <- c("min","moyenne","max")
# rappel des unités
unites <- c("ans","cm","kg")
# affichage
print( rbind( round(res),unites),quote=FALSE,right=TRUE )
AGE TAILLE POIDS
min 12 145 43
moyenne 34 167 72
max 73 194 116
unites ans cm kg
C'est pourquoi R fournit une fonction summary() dans le package base afin de connaitre un peu les données, fonction que Rcmdr propose dans le menu Statistiques, sous-menu Résumés, sous-sous-menu Jeu de données actif mais attention là encore parce que Rcmdr ne sait pas distinguer les variables qualitatives des variables quantitatives et vient calculer la moyenne du code-sexe !
# extraction des données
her_selection <- her[ , c("AGE","TAILLE","POIDS") ]
# résumé avec summary
summary( her_selection )
AGE TAILLE POIDS
Min. :12.00 Min. :144.8 Min. : 42.80
1st Qu.:23.75 1st Qu.:160.2 1st Qu.: 61.20
Median :32.00 Median :168.0 Median : 73.00
Mean :34.35 Mean :167.0 Mean : 72.29
3rd Qu.:42.50 3rd Qu.:173.5 3rd Qu.: 81.38
Max. :73.00 Max. :193.5 Max. :116.10
8. Réalisation de graphiques
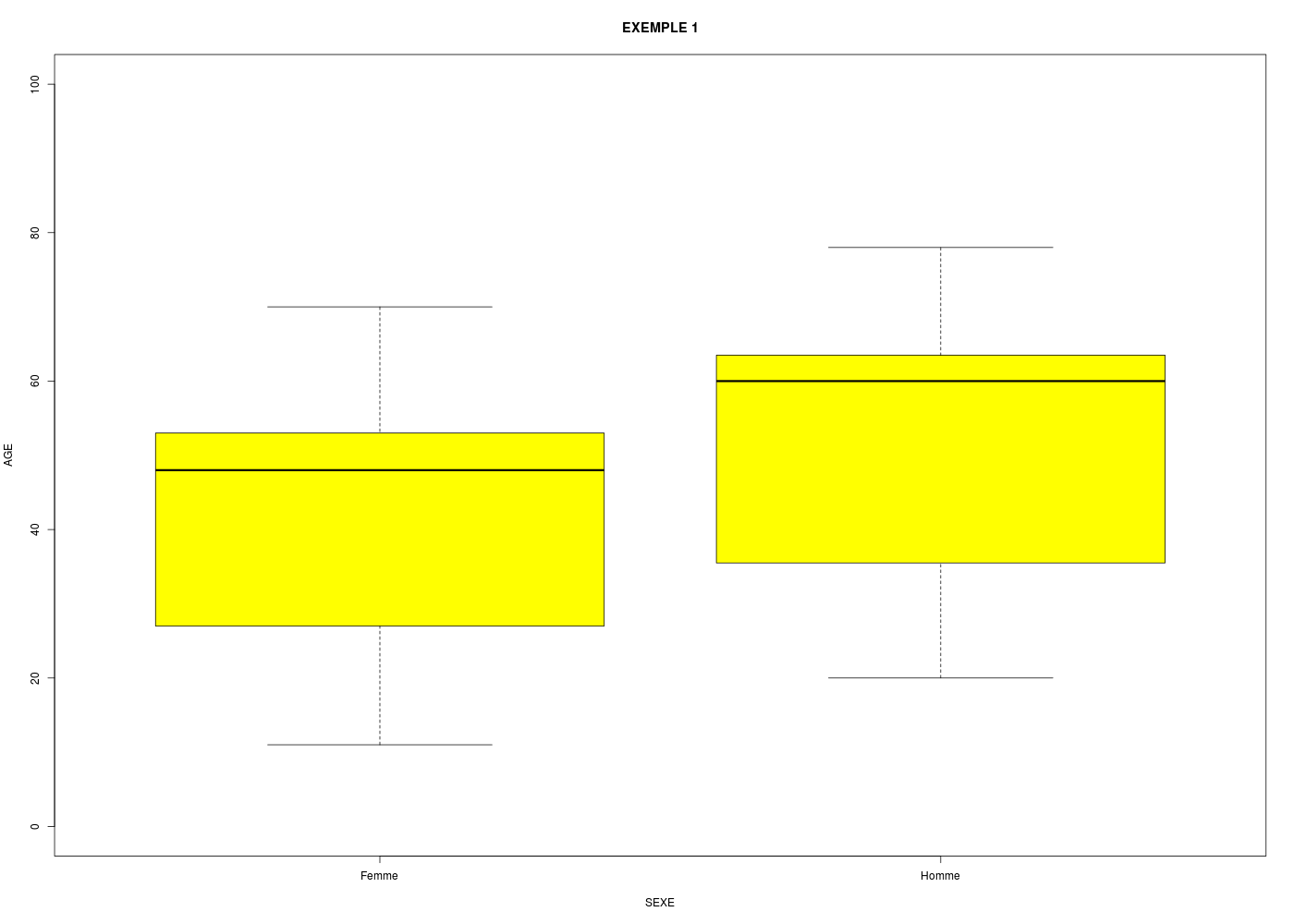
Comment tracer des boxplots pour la colonne AGE en fonction de la colonne SEXE dans le fichier Excel nommé exemple1.xls ?
On essaiera d'utiliser d'abord Rcmdr puis R en direct.
Rcmdr nomme boite de dispersion ce que nous appelons (avec d'autres) boxplot. Pour les tracer avec Rcmdr, après avoir chargé les données, on passe par le menu Graphes, sous-menu Boîte de dispersion. On clique alors sur la variable AGE puis on clique sur le bouton Graphe par groupe et on sélectionne ensuite SEXE dans le panneau qui apparait.
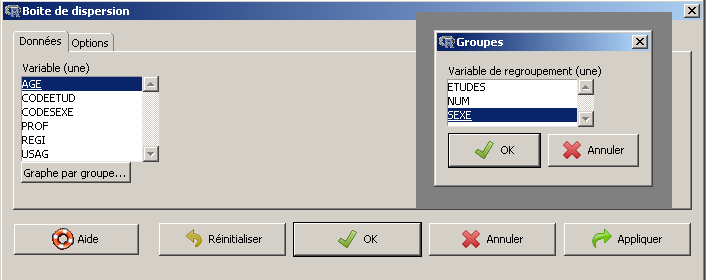
Il ne reste plus qu'à cliquer sur Ok pour voir les boxplots, qu'on peut ensuite exporter au format graphique que l'on veut :
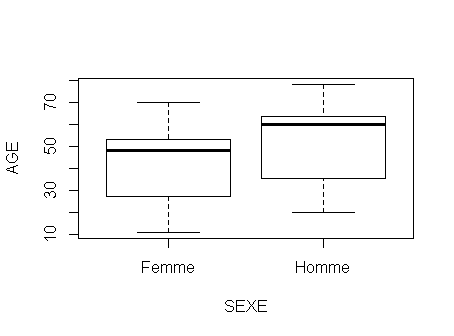
Remarque : Rcmdr ne permet pas d'utiliser de couleur dans les boxplots. Par contre on peut se servir du code fourni par Rcmdr et de l'aide pour faire un graphique plus "clean" :
# réalisation du boxplot de AGE en fonction de SEXE
# pour les données du fichier exemple1.xls
# chargement du package gdata afin d'utiliser la fonction read.xls()
library("gdata")
# lecture du fichier Excel "exemple1.xls" dans la variable xmp1
xmp1 <- read.xls("exemple1.xls")
# réalisation du boxplot
boxplot(
formula=AGE~SEXE,
data=xmp1,
col="yellow",
ylim=c(0,100),
xlab="SEXE",
ylab="AGE",
main="EXEMPLE 1"
) # fin de boxplot
9. Lecture de fichiers-texte
Comment lire les fichiers-texte exemple3.txt et exemple4.txt ?
On essaiera d'utiliser d'abord Rcmdr puis R en direct.
Pour le fichier exemple3.txt, il n'y a aucun problème avec la lecture sous Rcmdr. Il suffit de passer par le menu Données, sous-menu Importer des données, sous-sous-menu depuis un fichier-texte, le presse-papier ou une URL. Cela est du au fait que par défaut R (et donc Rcmdr) ignore les lignes vides et que le symbole dièse est le symbole de commentaire (une ligne de commentaire est ignorée à la lecture).
La lecture en direct avec R est tout aussi immédiate via la fonction read.table() du package utils : il n'y a que les noms de colonne en ligne 1 à préciser comme option de lecture (header=TRUE).
# lecture du fichier exemple3.txt sachant que :
# - on dispose du nom des colonnes comme première ligne de données
# - il y a des lignes vides
# - il y a des commentaires (lignes qui commencent par #)
xmp3 <- read.table(file="exemple3.txt",header=TRUE)
print(xmp3)
Par contre, pour le fichier exemple4.txt, Rcmdr dans ses options de lecture ne permet pas de préciser qu'il faut ignorer les lignes qui commencent par une étoile. On peut préciser que la virgule indique les décimales mais ce n'est pas suffisant : Rcmdr ne peut donc pas lire ce fichier-texte.
Nous allons donc réaliser cette lecture en direct avec R avec les bonnes valeurs de paramètres :
# lecture du fichier exemple4.txt sachant que :
# - on dispose du nom des colonnes comme première ligne de données
# - une étoile indique une ligne de commentaire
# - la virgule indique les décimales
xmp4 <- read.table(
file="exemple4.txt",
header=TRUE,
comment.char="*",
dec=","
) # fin de read.table
print(xmp4)
10. Création de variables
Comment créer une variable à partir d'autres variables, par exemple la surface à partir de la longueur et de la largeur ? Ou le BMI qui est le rapport masse/(taille*taille) ? Comment discrétiser une variable, par exemple pour avoir "jeune" si l'age est inférieur à 30 et "vieux" sinon ? Comment découper en 4 classes si on connait les bornes des classes ?
La fonction "magique" pour les formules se nomme transform(). Pour la discrétisation binaire, il est en général conseillé de passer par ifelse(). Le découpage en plus de deux classes peut se faire via cut(). Voici ces fonctions en action.
La fonction transform() renvoie un data.frame avec la ou les nouvelles variables :
# exemple 1 :
# -----------
# simulation d'un data frame avec des longueurs et des largeurs entières
long <- round(runif(20,min=5,max=20))
larg <- round(runif(20,min=3,max=10))
demo <- data.frame(long,larg)
cat("\ndonnées initiales\n")
print(demo)
# calcul de la surface via transform
demo <- transform(demo,surf=long*larg)
cat("\ndonnées complétées (1)\n")
print(demo)
# peut ajouter plusieurs variables d'un coup
demo <- transform(demo,perim=2*(long+larg),carre=long*long)
cat("\ndonnées complétées (2)\n")
print(demo)
# exemple 2 :
# -----------
# calcul du BMI
#
library(gdata)
her <- read.xls("her.xls")
cat("\ndonnées HER originales\n")
print(head(her))
her2 <- her[,(1:5)]
her2 <- transform(her2,bmi=POIDS*(10**4)/(TAILLE*TAILLE))
cat("\ndonnées HER avec BMI\n")
print(head(her2))
données initiales
long larg
1 12 4
2 8 3
3 12 7
4 14 8
5 17 4
6 7 3
7 18 4
8 10 5
9 6 5
10 5 5
11 18 4
12 20 4
13 8 5
14 15 9
15 16 8
16 11 8
17 8 8
18 5 9
19 9 8
20 17 7
données complétées (1)
long larg surf
1 12 4 48
2 8 3 24
3 12 7 84
4 14 8 112
5 17 4 68
6 7 3 21
7 18 4 72
8 10 5 50
9 6 5 30
10 5 5 25
11 18 4 72
12 20 4 80
13 8 5 40
14 15 9 135
15 16 8 128
16 11 8 88
17 8 8 64
18 5 9 45
19 9 8 72
20 17 7 119
données complétées (2)
long larg surf perim carre
1 12 4 48 32 144
2 8 3 24 22 64
3 12 7 84 38 144
4 14 8 112 44 196
5 17 4 68 42 289
6 7 3 21 20 49
7 18 4 72 44 324
8 10 5 50 30 100
9 6 5 30 22 36
10 5 5 25 20 25
11 18 4 72 44 324
12 20 4 80 48 400
13 8 5 40 26 64
14 15 9 135 48 225
15 16 8 128 48 256
16 11 8 88 38 121
17 8 8 64 32 64
18 5 9 45 28 25
19 9 8 72 34 81
20 17 7 119 48 289
données HER originales
IDEN SEXE AGE TAILLE POIDS TTAILLE POULS SYS DIA CHOL IMC JMBG COUD POIGN BRAS
1 I0001 0 58 179.8 76.7 90.6 68 125 78 522 23.8 42.5 7.7 6.4 31.9
2 I0002 0 22 168.1 65.4 78.1 64 107 54 127 23.2 40.2 7.6 6.2 31.0
3 I0003 0 32 182.1 81.3 96.5 88 126 81 740 24.6 44.4 7.3 5.8 32.7
4 I0004 0 31 174.5 79.7 87.7 72 110 68 49 26.2 42.8 7.5 5.9 33.4
5 I0005 0 28 171.7 69.2 87.1 64 110 66 230 23.5 40.0 7.1 6.0 30.1
6 I0006 0 46 175.8 75.7 92.4 72 107 83 316 24.5 47.3 7.1 5.8 30.5
données HER avec BMI
IDEN SEXE AGE TAILLE POIDS bmi
1 I0001 0 58 179.8 76.7 23.72553
2 I0002 0 22 168.1 65.4 23.14421
3 I0003 0 32 182.1 81.3 24.51719
4 I0004 0 31 174.5 79.7 26.17384
5 I0005 0 28 171.7 69.2 23.47283
6 I0006 0 46 175.8 75.7 24.49391
La fonction ifelse() renvoie un vecteur contenant l'une ou l'autre des valeurs spécifiées en fonction de la condition. Comme c'est une fonction vectorielle, on peut l'utiliser conjointement avec transform(). Si on a plus de deux catégories en sortie, on précise dans cut() les bornes et les labels des classes.
# discrétisation 1 :
# ------------------
# on est jeune quand on est moins de 30 ans...
#
library(gdata)
her <- read.xls("her.xls")
cat("\ndonnées HER originales\n")
print(head(her))
her2 <- her[,(1:5)]
her2 <- transform(her2,adjectif=ifelse(AGE<30,"jeune","vieux"))
cat("\ndonnées HER avec qualificatif (1)\n")
print(head(her2))
# discrétisation 2 :
# ------------------
# on est junior avant 20 ans
# cador entre 20 et 40
# castor entre 40 et 60
# senior après 60
# une solution un peu poussive mais facile à comprendre
her3 <- her[,(1:5)]
her3 <- transform(her3,qualif=NA)
her3[ her$AGE<20, "qualif" ] <- "junior"
her3[ (her$AGE>=20) & (her$AGE<40), "qualif" ] <- "cador"
her3[ (her$AGE>=40) & (her$AGE<60), "qualif" ] <- "castor"
her3[ her$AGE>60, "qualif" ] <- "senior"
cat("\ndonnées HER avec qualificatif (2)\n")
print(head(her3))
# avec la fonction cut
her3 <- transform(her3,qualif2=cut(x=AGE,breaks=c(0,20,40,60,100)))
cat("\ndonnées HER avec qualificatif (3)\n")
print(head(her3))
her3 <- transform(her3,qualif3=cut(x=AGE,breaks=c(0,20,40,60,100),labels=c("Junior","Cador","Castor","Senior")))
cat("\ndonnées HER avec qualificatif (4)\n")
print(head(her3))
données HER originales
IDEN SEXE AGE TAILLE POIDS TTAILLE POULS SYS DIA CHOL IMC JMBG COUD POIGN BRAS
1 I0001 0 58 179.8 76.7 90.6 68 125 78 522 23.8 42.5 7.7 6.4 31.9
2 I0002 0 22 168.1 65.4 78.1 64 107 54 127 23.2 40.2 7.6 6.2 31.0
3 I0003 0 32 182.1 81.3 96.5 88 126 81 740 24.6 44.4 7.3 5.8 32.7
4 I0004 0 31 174.5 79.7 87.7 72 110 68 49 26.2 42.8 7.5 5.9 33.4
5 I0005 0 28 171.7 69.2 87.1 64 110 66 230 23.5 40.0 7.1 6.0 30.1
6 I0006 0 46 175.8 75.7 92.4 72 107 83 316 24.5 47.3 7.1 5.8 30.5
données HER avec qualificatif (1)
IDEN SEXE AGE TAILLE POIDS adjectif
1 I0001 0 58 179.8 76.7 vieux
2 I0002 0 22 168.1 65.4 jeune
3 I0003 0 32 182.1 81.3 vieux
4 I0004 0 31 174.5 79.7 vieux
5 I0005 0 28 171.7 69.2 jeune
6 I0006 0 46 175.8 75.7 vieux
données HER avec qualificatif (2)
IDEN SEXE AGE TAILLE POIDS qualif
1 I0001 0 58 179.8 76.7 castor
2 I0002 0 22 168.1 65.4 cador
3 I0003 0 32 182.1 81.3 cador
4 I0004 0 31 174.5 79.7 cador
5 I0005 0 28 171.7 69.2 cador
6 I0006 0 46 175.8 75.7 castor
données HER avec qualificatif (3)
IDEN SEXE AGE TAILLE POIDS qualif qualif2
1 I0001 0 58 179.8 76.7 castor (40,60]
2 I0002 0 22 168.1 65.4 cador (20,40]
3 I0003 0 32 182.1 81.3 cador (20,40]
4 I0004 0 31 174.5 79.7 cador (20,40]
5 I0005 0 28 171.7 69.2 cador (20,40]
6 I0006 0 46 175.8 75.7 castor (40,60]
données HER avec qualificatif (4)
IDEN SEXE AGE TAILLE POIDS qualif qualif2 qualif3
1 I0001 0 58 179.8 76.7 castor (40,60] Castor
2 I0002 0 22 168.1 65.4 cador (20,40] Cador
3 I0003 0 32 182.1 81.3 cador (20,40] Cador
4 I0004 0 31 174.5 79.7 cador (20,40] Cador
5 I0005 0 28 171.7 69.2 cador (20,40] Cador
6 I0006 0 46 175.8 75.7 castor (40,60] Castor
11. Fusion de données
Je dispose de deux tableaux de données et je voudrais les fusionner. Quelles sont les fonctions R correspondantes ?
Fusionner a plusieurs sens. Pour une simple concaténation de données, il faut utiliser rbind() ou cbind() suivant qu'on aboute dans le sens des lignes ou des colonnes. Pour la "vraie" fusion de données selon des clés d'appariement, il faut passer par merge().
Attention : pour que rbind() et cbind() fonctionnent, il avoir la même taille de données :
# simulation de deux vecteurs entiers de longueur 5
lng <- 5
v1 <- round(runif(lng,min=5,max=20))
v2 <- round(runif(lng,min=5,max=20))
# concaténation en ligne
cl <- rbind(v1,v2)
print(cl)
# concaténation en colonne
cc <- cbind(v1,v2)
print(cc)
# nature des objets
ll <- list(v1,v2,cl,cc)
print(cbind(sapply(X=ll,F=class)))
# comment la concaténation conserve les noms ?
names(v1) <- c("Sophie","Jean","Pierre","Louise","Marie")
names(v2) <- c("Andy","John","Peter","Louisa","Paula")
# concaténation en ligne
cl <- rbind(v1,v2)
print(cl)
# concaténation en colonne
cc <- cbind(v1,v2)
print(cc)
# erreur avec des longueurs différentes :
v1 <- v1[-1] # on supprime le premier élément
# si on essaie d'exécuter cl <- rbind(v1,v2)
# on obtient :
#
# Message d'avis :
# In rbind(v1, v2) :
# number of columns of result is not a multiple of vector length (arg 1)
#
[,1] [,2] [,3] [,4] [,5]
v1 9 7 11 15 12
v2 15 11 14 19 6
v1 v2
[1,] 9 15
[2,] 7 11
[3,] 11 14
[4,] 15 19
[5,] 12 6
[,1]
[1,] "numeric"
[2,] "numeric"
[3,] "matrix"
[4,] "matrix"
Sophie Jean Pierre Louise Marie
v1 9 7 11 15 12
v2 15 11 14 19 6
v1 v2
Sophie 9 15
Jean 7 11
Pierre 11 14
Louise 15 19
Marie 12 6
La fusion générale se fait sur clé d'appariement via merge(). Il y a bien sûr des options pour préciser cette clé, ce qu'il faut faire des données orphelines à droite et à gauche... Les fichiers de données sont partie1.txt et partie2.txt et CLEAP signifie clé d'appariement :
(p1 <- read.table("partie1.txt",head=TRUE) )
(p2 <- read.table("partie2.txt",head=TRUE) )
( communs <- merge(p1,p2,by="CLEAP") )
( toutp1 <- merge(p1,p2,by="CLEAP",all.x=TRUE) )
( toutp2 <- merge(p1,p2,by="CLEAP",all.y=TRUE) )
( toustous <- merge(p1,p2,by="CLEAP",all=TRUE) )
names(p2)[1] <- "ID"
( communs <- merge(p1,p2,by.x="CLEAP",by.y="ID") )
( communs <- merge(p1,p2,by.x=1,by.y=1) )
> (p1 <- read.table("partie1.txt",head=TRUE) )
CLEAP NOM PRENOM AGE
1 801 DUPONT Jean 30
2 202 HUGO Victorine 25
3 403 ZOLA Emile 30
> (p2 <- read.table("partie2.txt",head=TRUE) )
CLEAP POIDS POINTS
1 801 56 300
2 403 50 600
3 705 58 150
> ( communs <- merge(p1,p2,by="CLEAP") )
CLEAP NOM PRENOM AGE POIDS POINTS
1 403 ZOLA Emile 30 50 600
2 801 DUPONT Jean 30 56 300
> ( toutp1 <- merge(p1,p2,by="CLEAP",all.x=TRUE) )
CLEAP NOM PRENOM AGE POIDS POINTS
1 202 HUGO Victorine 25 NA NA
2 403 ZOLA Emile 30 50 600
3 801 DUPONT Jean 30 56 300
> ( toutp2 <- merge(p1,p2,by="CLEAP",all.y=TRUE) )
CLEAP NOM PRENOM AGE POIDS POINTS
1 403 ZOLA Emile 30 50 600
2 705 <NA> <NA> NA 58 150
3 801 DUPONT Jean 30 56 300
> ( toustous <- merge(p1,p2,by="CLEAP",all=TRUE) )
CLEAP NOM PRENOM AGE POIDS POINTS
1 202 HUGO Victorine 25 NA NA
2 403 ZOLA Emile 30 50 600
3 705 <NA> <NA> NA 58 150
4 801 DUPONT Jean 30 56 300
> names(p2)[1] <- "ID"
> ( communs <- merge(p1,p2,by.x="CLEAP",by.y="ID") )
CLEAP NOM PRENOM AGE POIDS POINTS
1 403 ZOLA Emile 30 50 600
2 801 DUPONT Jean 30 56 300
> ( communs <- merge(p1,p2,by.x=1,by.y=1) )
CLEAP NOM PRENOM AGE POIDS POINTS
1 403 ZOLA Emile 30 50 600
2 801 DUPONT Jean 30 56 300
12. Calculs par sous-groupes
Comment effectuer des calculs et des rapports par sous-groupes ? Par exemple, comment effectuer des calculs sur l'age globalement puis pour les hommes seulement et enfin pour les femmes seulement ? Et pour des sous-échantillons plus techniques comme les femmes de plus de 40 ans dont la taille est inférieure à 180 cm ?
La réponse tient en deux fonctions de R : subset() et tapply() mais on peut aussi utiliser split().
urld <- "http://forge.info.univ-angers.fr/~gh/wstat/Introduction_R/personnes.dar"
pers <- read.table(urld,header=TRUE,row.names=1,encoding="latin1")
# on crée une liste avec une composante pour les hommes
# et une composante pour les femmes
decoupe <- split(x=pers$AGE,f=pers$SEXE)
# on peut alors utiliser lapply
print( lapply(X=decoupe,FUN=mean) )
# mais sapply est mieux :
print( sapply(X=decoupe,FUN=mean) )
# en fait, tapply fait tout cela en une ligne :
cats("Résultats avec tapply")
print( moys <- tapply( X=pers$AGE,INDEX=pers$SEXE, FUN=mean ) )
# par croisement
cats("Résultats par croisement")
print( moys <- tapply( X=pers$AGE,INDEX=list(pers$SEXE,pers$ETUD),FUN=mean ) )
$Femme
[1] 32.6
$Homme
[1] 37.05
Femme Homme
32.60 37.05
Résultats avec tapply
=====================
Femme Homme
32.60 37.05
Résultats par croisement
========================
Niveau_bac Secondaire Supérieur
Femme 29.75000 37.60000 19.00000
Homme 34.16667 43.36364 19.66667
Pour subset() il suffit de préciser le filtre des données :
# lecture des données
source("http://www.info.univ-angers.fr/~gh/statgh.r",encoding="latin1")
her <- lit.dar("http://www.info.univ-angers.fr/~gh/Datasets/her.dar")
# sous-ensemble 1
cat("\nHER1\n")
her1 <- subset(her,age>40)
print(her1)
# sous-ensemble 2
cat("\nHER2\n")
her2 <- subset(her,age>40,taille<180)
print(her2)
(gH) version 4.87
fonctions d'aides : lit() fqt() fql() ic() fapprox() chi2() fcomp() datagh()
taper aide() pour revoir cette liste
HER1
sexe age taille poids ttaille pouls sys dia chol imc jmbg coud poign bras
I0001 0 58 179.8 76.7 90.6 68 125 78 522 23.8 42.5 7.7 6.4 31.9
I0006 0 46 175.8 75.7 92.4 72 107 83 316 24.5 47.3 7.1 5.8 30.5
I0007 0 41 168.9 61.2 78.8 60 113 71 590 21.5 43.4 6.5 5.2 27.6
I0008 0 56 170.7 91.4 103.3 88 126 72 466 31.4 40.1 7.5 5.6 38.0
I0010 0 54 166.6 63.0 82.5 60 110 71 578 22.7 36.0 6.9 5.5 29.3
I0012 0 73 173.5 84.6 103.3 72 153 87 265 28.1 36.7 8.1 6.7 30.7
I0013 0 52 185.7 86.7 91.8 56 112 77 250 25.2 48.4 8.0 5.2 34.7
I0017 0 41 155.7 80.1 104.0 84 131 80 972 33.2 40.2 6.7 5.7 33.1
I0018 0 52 193.5 100.1 103.0 76 121 75 75 26.7 46.2 7.9 6.0 32.2
I0026 0 55 176.3 87.9 103.0 68 125 82 31 28.3 41.4 7.2 6.0 33.6
I0027 0 53 175.8 78.4 97.1 60 124 79 189 25.5 42.7 6.6 5.9 31.9
I0034 0 53 174.5 97.3 107.5 56 125 84 288 32.1 42.8 8.2 5.9 37.6
I0037 0 42 180.6 85.8 95.0 56 120 83 649 26.3 44.9 7.4 6.0 36.9
I0039 0 44 173.5 77.2 94.9 64 115 75 656 25.6 44.5 7.3 5.8 32.1
I0044 1 55 158.2 72.6 93.0 60 114 76 384 29.1 40.2 6.2 5.0 32.6
I0049 1 41 172.5 99.2 99.4 68 123 72 531 33.5 43.8 7.8 5.8 38.6
I0058 1 45 157.2 73.4 94.0 72 127 74 447 29.8 40.0 6.8 5.0 35.0
I0059 1 41 163.3 79.0 92.8 64 107 67 125 29.7 38.2 6.8 4.7 33.1
I0060 1 56 161.0 82.2 105.5 80 116 71 318 31.7 38.2 6.9 5.4 39.6
I0062 1 57 161.0 116.1 126.5 80 155 85 600 44.9 41.0 8.0 5.6 43.8
I0065 1 59 161.3 74.0 104.7 76 105 69 309 28.5 36.0 6.7 5.1 34.4
I0067 1 45 152.9 72.4 99.3 104 133 83 280 31.0 31.1 6.4 5.2 35.6
I0068 1 52 171.7 73.8 91.1 88 113 75 254 25.1 39.4 7.1 5.3 31.8
I0073 1 47 147.8 88.7 105.5 88 114 79 293 40.6 27.0 7.5 5.5 41.2
I0079 1 48 170.2 59.1 68.6 124 93 64 207 20.5 41.6 6.0 5.3 23.0
HER2
sexe age taille poids ttaille pouls sys dia chol imc jmbg coud poign bras
I0001 0 58 179.8 76.7 90.6 68 125 78 522 23.8 42.5 7.7 6.4 31.9
I0006 0 46 175.8 75.7 92.4 72 107 83 316 24.5 47.3 7.1 5.8 30.5
I0007 0 41 168.9 61.2 78.8 60 113 71 590 21.5 43.4 6.5 5.2 27.6
I0008 0 56 170.7 91.4 103.3 88 126 72 466 31.4 40.1 7.5 5.6 38.0
I0010 0 54 166.6 63.0 82.5 60 110 71 578 22.7 36.0 6.9 5.5 29.3
I0012 0 73 173.5 84.6 103.3 72 153 87 265 28.1 36.7 8.1 6.7 30.7
I0013 0 52 185.7 86.7 91.8 56 112 77 250 25.2 48.4 8.0 5.2 34.7
I0017 0 41 155.7 80.1 104.0 84 131 80 972 33.2 40.2 6.7 5.7 33.1
I0018 0 52 193.5 100.1 103.0 76 121 75 75 26.7 46.2 7.9 6.0 32.2
I0026 0 55 176.3 87.9 103.0 68 125 82 31 28.3 41.4 7.2 6.0 33.6
I0027 0 53 175.8 78.4 97.1 60 124 79 189 25.5 42.7 6.6 5.9 31.9
I0034 0 53 174.5 97.3 107.5 56 125 84 288 32.1 42.8 8.2 5.9 37.6
I0037 0 42 180.6 85.8 95.0 56 120 83 649 26.3 44.9 7.4 6.0 36.9
I0039 0 44 173.5 77.2 94.9 64 115 75 656 25.6 44.5 7.3 5.8 32.1
I0044 1 55 158.2 72.6 93.0 60 114 76 384 29.1 40.2 6.2 5.0 32.6
I0049 1 41 172.5 99.2 99.4 68 123 72 531 33.5 43.8 7.8 5.8 38.6
I0058 1 45 157.2 73.4 94.0 72 127 74 447 29.8 40.0 6.8 5.0 35.0
I0059 1 41 163.3 79.0 92.8 64 107 67 125 29.7 38.2 6.8 4.7 33.1
I0060 1 56 161.0 82.2 105.5 80 116 71 318 31.7 38.2 6.9 5.4 39.6
I0062 1 57 161.0 116.1 126.5 80 155 85 600 44.9 41.0 8.0 5.6 43.8
I0065 1 59 161.3 74.0 104.7 76 105 69 309 28.5 36.0 6.7 5.1 34.4
I0067 1 45 152.9 72.4 99.3 104 133 83 280 31.0 31.1 6.4 5.2 35.6
I0068 1 52 171.7 73.8 91.1 88 113 75 254 25.1 39.4 7.1 5.3 31.8
I0073 1 47 147.8 88.7 105.5 88 114 79 293 40.6 27.0 7.5 5.5 41.2
I0079 1 48 170.2 59.1 68.6 124 93 64 207 20.5 41.6 6.0 5.3 23.0
13. Automatisation de commandes
Je dispose de plusieurs fichiers Excel comme fic1ser1.xls, fic2ser1.xls et fic3ser1.xls ou de plusieurs feuilles de calcul dans le même fichier Excel comme f07s1, f08s1, f09s1, f10s1, f11s2, f12s2 et f13s2 dans le fichier series.xls. Comment automatiser le traitement de ces fichiers par exemple pour afficher le nombre de lignes de données dans chaque fichier ?
La première chose à faire est d'apprendre à utiliser les fonctions sprintf(), paste() et seq() -- dont le raccourci est le symbole deux-points -- du package base pour générer des listes de fichiers. Par exemple la première série de fichiers se compose des trois lettres "fic" puis d'un nombre qui varie de 1 à 3 puis du texte "ser1.xls" ; on peut donc la générer par :
# on produit la liste de fichier "fic1ser1.xls" "fic2ser1.xls" "fic3ser1.xls"
liste1 <- paste("fic",1:3,"ser1.xls",sep="")
print(liste1)
"fic1ser1.xls" "fic2ser1.xls" "fic3ser1.xls"
Il ne faut pas oublier de mettre sep="" qui indique que le séparateur est une chaine vide parce que sinon R rajoute un espace entre les "fic" et les numéros de fichier. On aurait donc "fic 1 ser1.xls" pour le premier nom de fichier, ce qui n'est pas ce qu'on voulait.
Pour la deuxième série, il y a deux parties. La première n'utilise pas les entiers de 7 à 10 mais des entiers formatés avec un zéro de tête, c'est pourquoi il faut passer par la fonction sprintf() :
# on produit la liste d'onglets
# f07s1, f08s1, f09s1, f10s1, f11s2, f12s2 et f13s2
# en deux temps
partie1 <- paste("f", sprintf("%02d",7:10),"s1",sep="")
partie2 <- paste("f", 11:13 ,"s2",sep="")
liste2 <- c( partie1, partie2)
print(liste2)
"f07s1" "f08s1" "f09s1" "f10s1" "f11s2" "f12s2" "f13s2"
La deuxième chose à faire est d'apprendre à écrire une boucle et à la mettre dans un script.
Commençons par vérifier qu'on sait afficher le nombre de lignes de données du premier fichier, à savoir "fic1ser1.xls" à al'aide la fonction nrow() :
# lecture du fichier "fic1ser1.xls"
# et affichage du nombre de lignes de données
library(gdata)
nomfic <- "fic1ser1.xls"
fdata <- read.xls(xls=nomfic)
cat(" il y a ",nrow(fdata)," lignes de données dans ",nomfic,"\n")
il y a 3 lignes de données dans fic1ser1.xls
Il ne reste plus qu'à utiliser la boucle pour -- for en anglais -- dont l'aide s'obtient par help("for") et qui permet de passer en revue chacun des noms de fichier :
# lecture du fichier "fic1ser1.xls"
# et affichage du nombre de lignes de données
library(gdata)
# on produit la liste des fichiers
liste1 <- paste("fic",1:3,"ser1.xls",sep="")
# puis on utilise une boucle pour afficher
for (nomfic in liste1) {
fdata <- read.xls(xls=nomfic)
cat(" il y a ",nrow(fdata)," lignes de données dans ",nomfic,"\n")
} # fin pour
il y a 3 lignes de données dans fic1ser1.xls
il y a 4 lignes de données dans fic2ser1.xls
il y a 5 lignes de données dans fic3ser1.xls
Mais ce n'est bien sûr pas la seule solution. En voici une autre, plus adaptée à de longues listes de fichiers :
# lecture du fichier "fic1ser1.xls"
# et affichage du nombre de lignes de données
library(gdata)
# on produit la liste des fichiers
liste1 <- paste("fic",1:3,"ser1.xls",sep="")
# puis on utilise une boucle avec indication
# de la progression dans l'affichage
nbf <- length(liste1)
for (numfic in (1:nbf)) {
nomfic <- liste1[numfic]
fdata <- read.xls(xls=nomfic)
cat(" fichier ",numfic," / ",nbf," : ",nrow(fdata)," lignes de données dans ",nomfic,"\n")
} # fin pour
fichier 1 / 3 : 3 lignes de données dans fic1ser1.xls
fichier 2 / 3 : 4 lignes de données dans fic2ser1.xls
fichier 3 / 3 : 5 lignes de données dans fic3ser1.xls
Enfin, si on veut exploiter les résultats, il faut transférer les calculs dans une matrice. Par exemple, voici comment afficher le nombre de lignes et la moyenne de l'age dans chacun des onglets du fichier series.xls :
# calculs pour divers onglet d'un même fichier Excel
# 1. on produit la liste d'onglets
# f07s1, f08s1, f09s1, f10s1, f11s2, f12s2 et f13s2
# (en deux temps)
partie1 <- paste("f", sprintf("%02d",7:10),"s1",sep="")
partie2 <- paste("f", 11:13 ,"s2",sep="")
listeOnglets <- c( partie1, partie2)
nbOnglets <- length(listeOnglets)
# 2. on prépare la matrice de résultats (mdr)
colonnes <- c("Nb_Lignes","Moyenne_Age")
mdr <- matrix(-1,nrow=nbOnglets,ncol=length(colonnes))
row.names(mdr) <- listeOnglets
colnames(mdr) <- colonnes
# 3. on charge la librairie gdata
# puis on parcourt les ongets et on remplit la matrice
# de résultats
library(gdata)
for (idOnglet in (1: nbOnglets)) {
onglet <- read.xls("series.xls",sheet=listeOnglets[idOnglet])
mdr[idOnglet,1] <- nrow(onglet)
mdr[idOnglet,2] <- mean(onglet$AGE)
} # fin pour idongl
# 4. affichage des résultats
print( transform(mdr,Moyenne_Age=round(Moyenne_Age,1)) )
Nb_Lignes Moyenne_Age
f07s1 5 40.4
f08s1 10 46.9
f09s1 20 45.1
f10s1 10 46.9
f11s2 7 41.6
f12s2 20 45.1
f13s2 19 45.0
Modifions maintenant ce script (car ce n'est pas un programme, juste un ensemble d'instructions) pour qu'il affiche aussi le nombre de femmes (code SEXE = 1) par onglet.
# calculs pour divers onglet d'un même fichier Excel
# version 2 avec calcul du nombre de femmes
# 1. on produit la liste d'onglets
# f07s1, f08s1, f09s1, f10s1, f11s2, f12s2 et f13s2
# (en deux temps)
partie1 <- paste("f", sprintf("%02d",7:10),"s1",sep="")
partie2 <- paste("f", 11:13 ,"s2",sep="")
listeOnglets <- c( partie1, partie2)
nbOnglets <- length(listeOnglets)
# 2. on prépare la matrice de résultats (mdr)
colonnes <- c("Nb_Lignes","Moyenne_Age","Nb_Femmes")
mdr <- matrix(-1,nrow=nbOnglets,ncol=length(colonnes))
row.names(mdr) <- listeOnglets
colnames(mdr) <- colonnes
# 3. on charge la librairie gdata
# puis on parcourt les ongets et on remplit la matrice
# de résultats
library(gdata)
for (idOnglet in (1: nbOnglets)) {
onglet <- read.xls("series.xls",sheet=listeOnglets[idOnglet])
mdr[idOnglet,1] <- nrow(onglet)
mdr[idOnglet,2] <- mean(onglet$AGE)
mdr[idOnglet,3] <- sum(onglet$CODESEXE==1)
} # fin pour idongl
# 4. affichage des résultats
print( transform(mdr,Moyenne_Age=round(Moyenne_Age,1)) )
Nb_Lignes Moyenne_Age Nb_Femmes
f07s1 5 40.4 3
f08s1 10 46.9 7
f09s1 20 45.1 13
f10s1 10 46.9 7
f11s2 7 41.6 5
f12s2 20 45.1 13
f13s2 19 45.0 12
Remarque : on aurait pu créer une fonction (par exemple analyseOnglet()) pour remplir chaque ligne de la matrice des résultats (mdr).
# calculs pour divers onglet d'un même fichier Excel
# version 2 avec calcul du nombre de femmes
# 1. on produit la liste d'onglets
# f07s1, f08s1, f09s1, f10s1, f11s2, f12s2 et f13s2
# (en deux temps)
partie1 <- paste("f", sprintf("%02d",7:10),"s1",sep="")
partie2 <- paste("f", 11:13 ,"s2",sep="")
listeOnglets <- c( partie1, partie2)
nbOnglets <- length(listeOnglets)
# 2. on prépare la matrice de résultats (mdr)
colonnes <- c("Nb.Lignes","Moyenne.Age","Nb.Femmes")
mdr <- matrix(-1,nrow=nbOnglets,ncol=length(colonnes))
row.names(mdr) <- listeOnglets
colnames(mdr) <- colonnes
# 3. on charge la librairie gdata
# puis on parcourt les ongets et on remplit la matrice
# de résultats avec une fonction analyseOnglet
analyseOnglet <- function(onglet) {
calcul1 <- nrow(onglet)
calcul2 <- mean(onglet$AGE)
calcul3 <- sum(onglet$CODESEXE==1)
return( c(calcul1,calcul2,calcul3) )
} # fin de fonction analyseOnglet
library(gdata)
for (idOnglet in (1: nbOnglets)) {
ongletc <- read.xls("series.xls",sheet=listeOnglets[idOnglet])
mdr[idOnglet,] <- analyseOnglet(ongletc)
# non conseillé pour des raisons de lisibilité
# mdr[idOnglet,] <- analyseOnglet( read.xls("series.xls",sheet=listeOnglets[idOnglet]) )
} # fin pour idongl
# 4. affichage des résultats
print( transform(mdr,Moyenne_Age=round(Moyenne.Age,2)) )
Nb.Lignes Moyenne.Age Nb.Femmes Moyenne_Age
f07s1 5 40.40000 3 40.40
f08s1 10 46.90000 7 46.90
f09s1 20 45.15000 13 45.15
f10s1 10 46.90000 7 46.90
f11s2 7 41.57143 5 41.57
f12s2 20 45.15000 13 45.15
f13s2 19 45.00000 12 45.00
|
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)




















