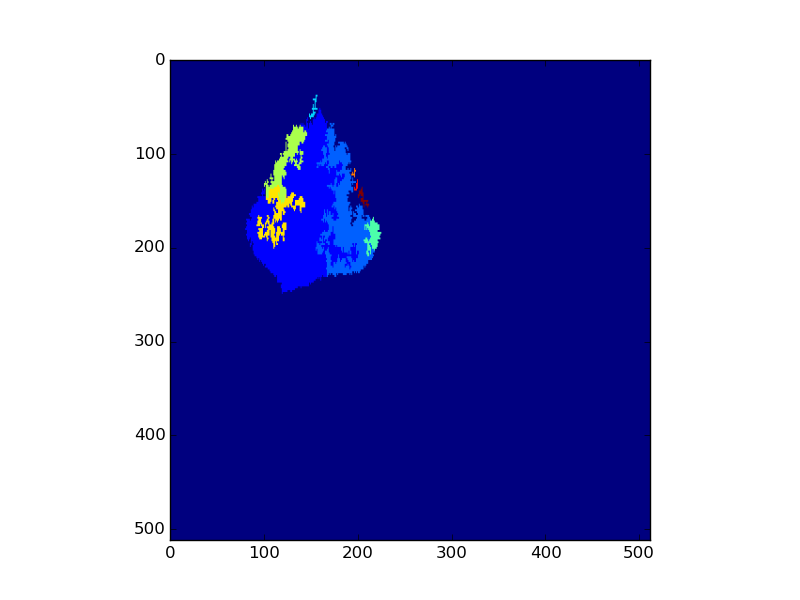
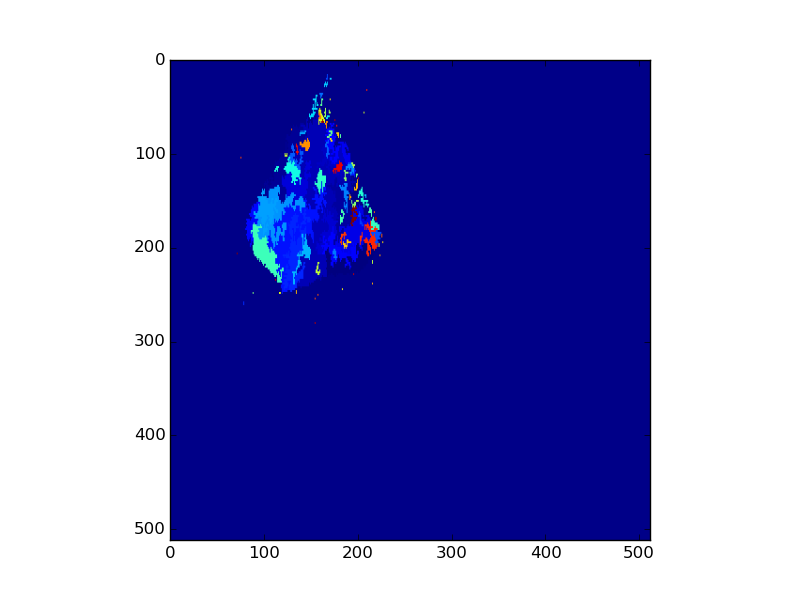
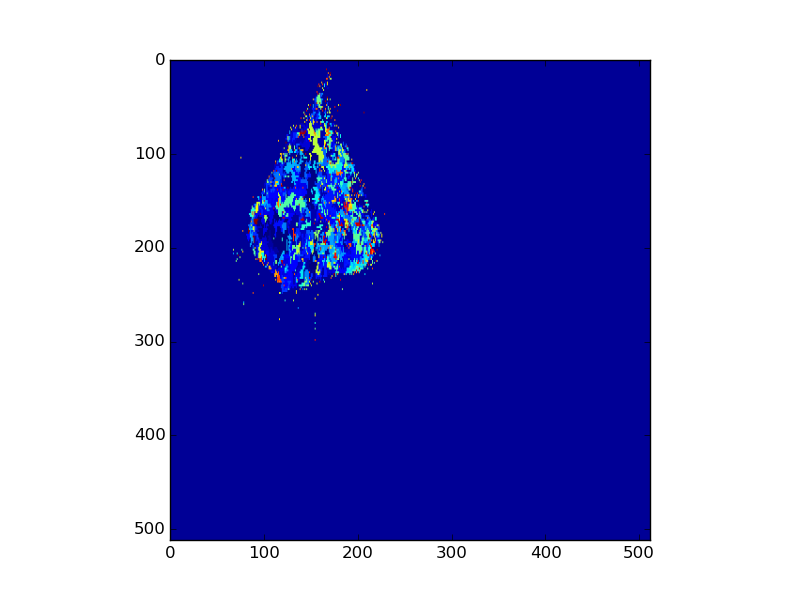
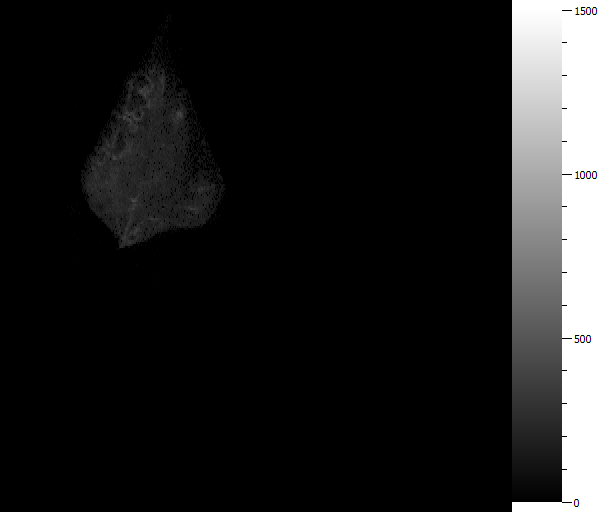
On dispose de différentes images pour une même feuille correspondant à divers paramètres de fluorescence, comme par exemple les images suivantes pour la feuille Feuille003J03-1390.
|
Fm_D1-Feuille003J03-1390.bmp
|

Qp_Lss-Feuille003J03-1390.bmp
|

Qp_D1-Feuille003J03-1390.bmp
|
On voudrait regrouper les pixels semblables en clusters sur l'ensemble des images pour une même feuille, sachant qu'il y a en général quelques dizaines d'images (souvent 60) pour une feuille donnée, que les images ont pour taille 512x612 (dont 512x512 "utiles" puisque la dernière bande 100x512 correspond à l'échelle d'intensité).
Le langage Python dispose de nombreuses bibliothèques scientifiques dont
|
numpy
|
NUMerical computations with PYthon |
|
matplotlib
|
MAThematical PLOTting LIBrary for python |
|
PIL
|
Python Image Library |
|
sklearn
|
Scientific Kit for machine LEARNing in python |
Dans cette dernière bibliothèque, il y a une fonction de classification ascendante nommée AgglomerativeClustering qui pourrait nous être utile. Hélas, avec autant d'informations (n = 60 x 512 x 512 est de l'orde de 10 puissance 7), il est impossible de mettre en mémoire une matrice de distance entres tous les pixels car elle est de taille nxn soit en gros 10 puissance 14.
Donc une démarche directe de calcul via le script python suivant est voué à l'échec, avec un message Memory Error quasi-instantané.
import numpy as np
import matplotlib.pyplot as plt
from PIL import Image
from glob import glob
from sklearn.cluster import AgglomerativeClustering
from sklearn.feature_extraction.image import grid_to_graph
nbClusters = 5000
img_list = glob("./*Feuille003J03-1390*.bmp")
nimg = len(img_list)
print(str(nimg) + " images vues")
X = np.empty((512, 512, nimg))
for i, j in enumerate(img_list):
X[:, :, i] = np.array(Image.open(j))[:, :512, 0]
# fin pour i et j
dshape = (X.shape[0], X.shape[1])
X = np.reshape(X, (-1, nimg))
ward = AgglomerativeClustering(n_clusters=nbClusters, linkage='ward').fit(X)
label = np.reshape(ward.labels_, dshape)
nomFig = "cluster" + str(nbClusters) + ".png"
plt.imshow(label)
plt.savefig(nomFig)
print("image "+ nomFig + " generated")
Heureusement, pyhton sait utiliser la notion de connectivité, c'est-à-dire se servir uniquement de voisins proches pour calculer des distances, ce qui permet en quelques dizaines de secondes de réaliser un "clustering" local un peu grossier mais efficace via le script suivant :
import sys
import numpy as np
import matplotlib.pyplot as plt
from PIL import Image
from glob import glob
from sklearn.cluster import AgglomerativeClustering
from sklearn.feature_extraction.image import grid_to_graph
nbClusters = 5000
img_list = glob("./*Feuille003J03-1390*.bmp")
nimg = len(img_list)
print(str(nimg) + " images vues")
if (nimg==0) :
sys.exit(-1)
# finsi
X = np.empty((512, 512, nimg))
for i, j in enumerate(img_list):
X[:, :, i] = np.array(Image.open(j))[:, :512, 0]
# fin pour i et j
dshape = (X.shape[0], X.shape[1])
connectivity = grid_to_graph(*dshape)
X = np.reshape(X, (-1, nimg))
ward = AgglomerativeClustering(n_clusters=nbClusters,
connectivity=connectivity,linkage='ward').fit(X)
label = np.reshape(ward.labels_, dshape)
plt.imshow(label) # affichage interactif possile avec plt.imshow(label)
nomFig = "cluster" + str(nbClusters) + ".png"
plt.savefig(nomFig)
print("image "+ nomFig + " disponible.\n")
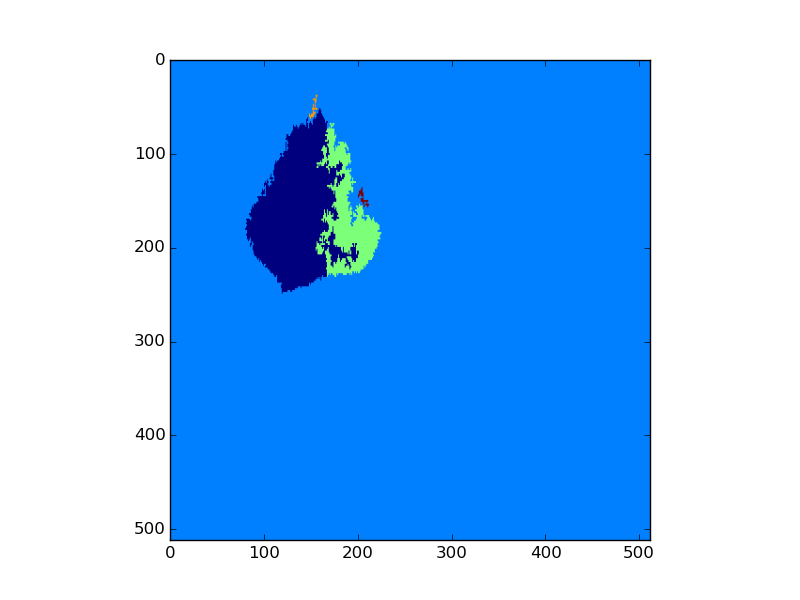
Il faut maintenant regrouper les classes ou "clusters" issues de la première partie de classification après avoir choisi le nombre k de clusters.
Le but est d'arriver à révéler l'architecture de la plante, et en particulier les nervures, ainsi que toute modification de la texture, par exemple sous l'influence d'une bactérie phytopathogène. Voici quelques exemples d'architectures visibles :
Voici le début d'un script (non finalisé) pour effectuer la classification :
import numpy as np
import matplotlib.pyplot as plt
from PIL import Image
from glob import glob
from sklearn.cluster import FeatureAgglomeration
from sklearn.feature_extraction.image import grid_to_graph
img_list = glob("./haricot/"
"*Feuille003J03-1390*.bmp")
nimg = len(img_list)
X = np.empty((nimg, 512, 512))
for i, j in enumerate(img_list):
X[i, :, :] = np.array(Image.open(j))[:, :512, 0]
dshape = (X.shape[1], X.shape[2])
connectivity = grid_to_graph(*dshape)
X = np.reshape(X, (nimg, -1))
n = 300
ward = FeatureAgglomeration(n_clusters=n,
linkage='ward',
pooling_func=np.mean,
connectivity=connectivity).fit(X)
# compute pooling_func (eg. np.mean) on each cluster
coef = ward.transform(X) # shape (nimg, n)
# put coef on each pixel according to clusters
X_ = ward.inverse_transform(coef) # shape (nimg, 512*512)
max_intensity = 60
param = 0
plt.imshow(np.reshape(X_[param], dshape), vmin=0, vmax=max_intensity)
plt.colorbar()
plt.show()
Après examen minutieux, la première image de fluorescence, nommée Fm_D1-Feuille003J03-1390.bmp montre déjà un peu cette architecture.
 Retour à la page principale de
(gH)
Retour à la page principale de
(gH)